Nature | 克隆拷贝数多样性影响肺癌生存
关键词
肺癌、克隆拷贝数、ALPACA方法、染色体不稳定性、生存预后
摘要总结
这篇文章是2025年8月发表在《Nature》杂志上的一篇研究,标题为“Clone copy number diversity is linked to survival in lung cancer”。这篇文章通过开发新型算法ALPACA,探索了肺癌中单核苷酸变异(SNV)和体细胞拷贝数变异(SCNA)的共同演化,这对于揭示肺癌转移机制、提升预后预测具有重要意义。
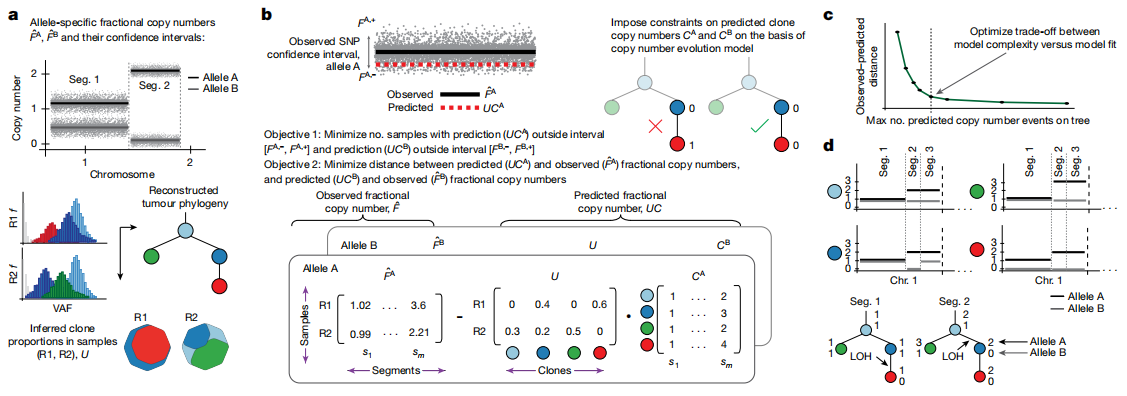
研究背景
癌症发展过程中,SNV和SCNA共同驱动克隆演化,导致肿瘤异质性和染色体不稳定性(CIN)。现有方法难以从bulk DNA测序数据中准确推断克隆特异性拷贝数,导致对肿瘤多样性和转移机制的理解不足。TRACERx421研究提供了多样本肺癌数据,但缺乏整合SNV和SCNA的工具。本研究旨在填补这一空白,探讨克隆拷贝数多样性与肺癌生存的相关性。
TRACERx421,全称是 “Tracking Cancer Evolution through Therapy (Rx)”(通过治疗追踪癌症演化),涉及 421 名未经治疗的非小细胞肺癌(NSCLC)患者。TRACERx421 的目标是揭示癌症克隆演化、转移机制以及与预后相关的基因组特征,为精准医学提供数据支持。
研究方法
研究开发了ALPACA算法,利用SNV频率重建的肿瘤系统树作为框架,结合多样本bulk DNA测序数据推断克隆特异性等位基因拷贝数。输入包括肿瘤克隆树、克隆比例和等位基因分数拷贝数,通过混合整数线性规划和模型选择优化SCNA演化。基准测试使用MASCoTE和Tx模拟肿瘤(n=150),并与HATCHet2、cloneHD等比较。应用到TRACERx421的395个原发肺癌和126个转移肿瘤,分析SCNA模式、事件时序和转移相关性,使用Bradley-Terry模型排序事件,并通过混合效应模型评估拷贝数变化。
研究结果
-
ALPACA在模拟肿瘤中准确率高于现有方法(P<0.001)
ALPACA是一种新算法,用于从多样本bulk DNA测序数据中推断肿瘤克隆特异性拷贝数(包括单核苷酸变异SNV和体细胞拷贝数变异SCNA)。在模拟肿瘤数据中,ALPACA的预测准确率显著优于现有方法(如HATCHet2和cloneHD),统计显著性P值小于0.001,表明结果高度可靠。 -
ALPACA可发现转移播种克隆中的LOH和扩增事件(如BRCA2、RB1损失和CCND1扩增)
ALPACA能够识别转移过程中“播种克隆”(即引发转移的克隆)中的特定基因组改变,包括“失双等位基因”(Loss of Heterozygosity, LOH)和基因扩增。具体例子包括BRCA2、RB1(肿瘤抑制基因)的LOH以及CCND1(细胞周期调控基因)的扩增。 -
肺腺癌(LUAD)中臂级扩增早于焦点扩增,驱动SNV(如EGFR、KRAS)较早发生;肺鳞癌(LUSC)中臂级事件多为树干
研究分析了LUAD和LUSC两种肺癌亚型中基因组事件的演化顺序。LUAD中,染色体臂级扩增(影响较大染色体区域)先于焦点扩增(影响特定基因),而驱动性SNV(如EGFR、KRAS突变)发生在早期。LUSC中,臂级事件(如LOH或扩增)多发生在“树干”(即所有克隆共有的早期祖先克隆)。 -
转移克隆显示更高SCNA速率(P=0.0474)
使用线性混合效应模型分析TRACERx421数据,发现转移克隆在每条系统发育树边上的SCNA数量显著增加。这可能反映转移克隆在适应新微环境中需要更多基因组改变。 -
播种克隆中TSG损失和CCND1扩增富集
引发转移的播种克隆中,肿瘤抑制基因(TSG,如SMARCA4、KEAP1)的LOH以及CCND1扩增事件显著富集,表明TSG损失可能削弱肿瘤抑制,CCND1扩增促进细胞周期加速,为转移提供遗传优势。 -
克隆拷贝数多样性增加与肺癌患者无病生存期缩短相关
肿瘤中克隆拷贝数多样性越高(即不同克隆间拷贝数差异越大),肺癌患者的无病生存期(疾病未复发的时间)越短。
文章的亮点和局限
亮点:ALPACA创新性地整合SNV和SCNA,提供克隆级分辨率,揭示隐藏在minor克隆中的事件,并关联转移和预后,有助于精准医学。
局限:依赖高SNV突变率(如>0.3/细胞分裂),可能不适用于低突变肿瘤;基于bulk数据,可能忽略罕见克隆;模拟和真实数据验证有限,需更多队列确认。
公开数据
The TRACERx primary and matched primary–metastasis processed data have been deposited at Zenodo at https://doi.org/10.5281/zenodo.7822002
(ref. [54](https://www.nature.com/articles/s41586-025-09398-w#ref-CR54 "TRACERx consortium. The evolution of lung cancer and impact of subclonal selection in TRACERx. Zenodo
https://doi.org/10.5281/zenodo.7822002
(2023).")). The single-cell study from the TRACERx PEACE study has been deposited at Zenodo at https://doi.org/10.5281/zenodo.13754279
(ref. [55](https://www.nature.com/articles/s41586-025-09398-w#ref-CR55 "Lucas, O., Zaidi, R., Bunkum, A. & Zaccaria, S. Processed data for “Characterising the evolutionary dynamics of cancer proliferation in single-cell clones with SPRINTER”. Zenodo
https://doi.org/10.5281/zenodo.13754279
(2024).")). Processed data used in this publication have been deposited at Zenodo at https://doi.org/10.5281/zenodo.15519765
(ref. [56](https://www.nature.com/articles/s41586-025-09398-w#ref-CR56 "Pawlik, P. & Grigoriadis, K. Processed data for “Clone copy number diversity is linked to survival in lung cancer”. Zenodo
https://doi.org/10.5281/zenodo.15519765
(2025).")). The WES data (from the TRACERx study) used during this study have been deposited at the European Genome–Phenome Archive (EGA), which is hosted by the European Bioinformatics Institute and the Centre for Genomic Regulation, under the accession code EGAS00001006494; raw single-cell DNA-sequencing data used in this study from the patients enrolled in the TRACERx and PEACE studies have been deposited at the EGA under the accession code EGAD00001015411.
