DockThor: 免费的在线小分子“虚拟筛选”平台
字数 586,阅读大约需 3 分钟
DockThor-VS是巴西国家科学计算实验室开发的 开源蛋白质-配体虚拟筛选平台,面向全球科研人员提供免费服务。其核心引擎采用柔性配体-刚性受体对接算法,结合多解遗传算法与MMFF94S分子力场,可精准预测结合构象,并通过DockTScore评分函数评估亲和力。
平台支持蛋白质质子化状态调整、辅酶建模等预处理功能,内置COVID-19靶点库、FDA批准药物数据集及天然产物库,适配药物设计与老药新用研究。依托SDumont超级计算机,可高效处理大规模筛选任务,单次提交支持最多2000个化合物。其自动化流程与高性能计算优势,显著提升虚拟筛选效率,已应用于抗新冠药物研发等场景。

网站地址:https://dockthor.lncc.br/v2/
使用教程
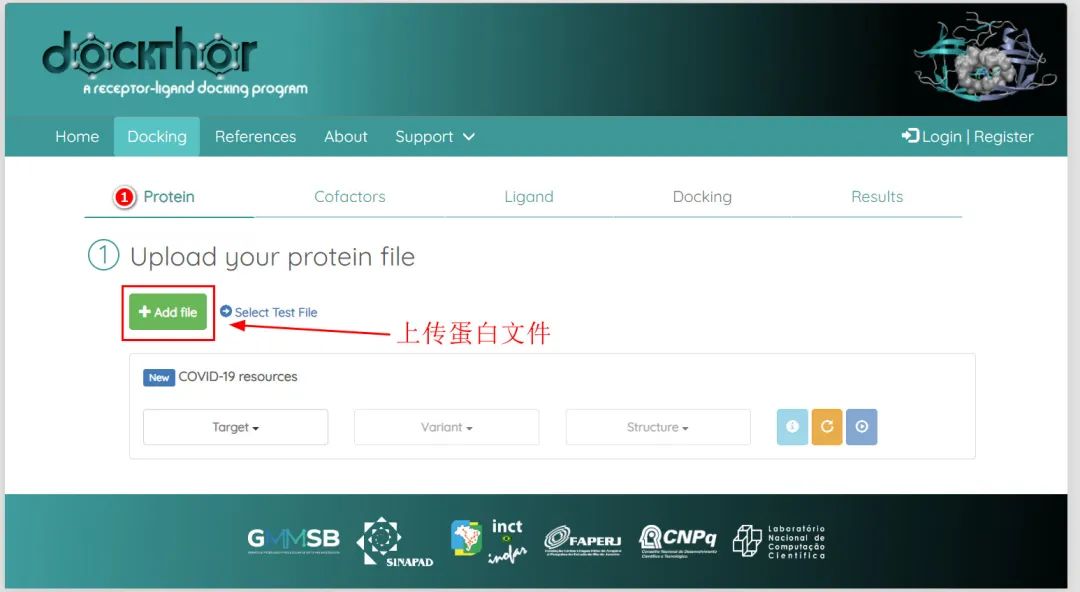
1.上传蛋白文件
将蛋白中的非保守水分子,辅助结晶试剂分子等删除干净后再上传。
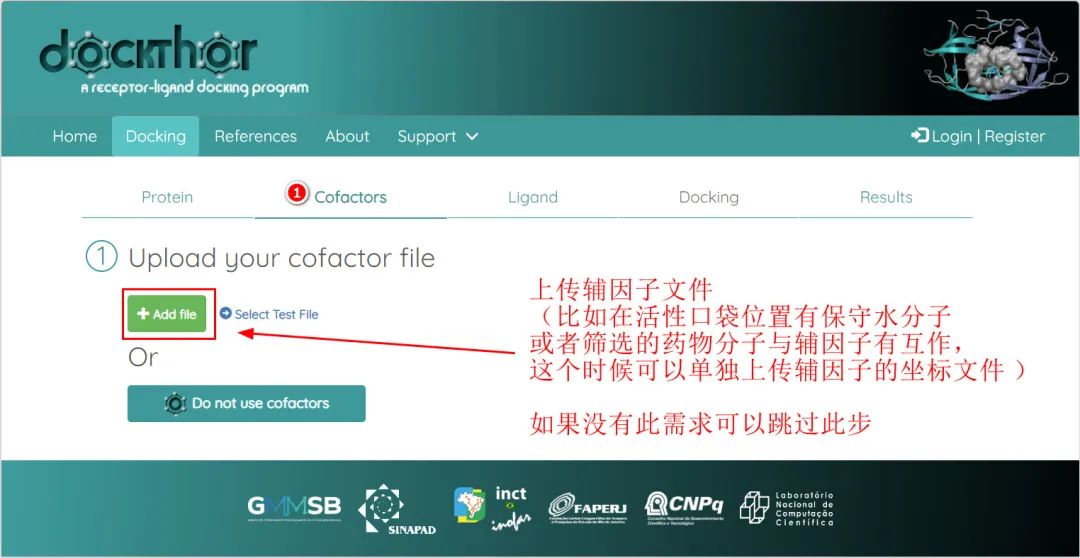
2. 上传辅因子文件(非必须)
如果你的蛋白活性位点中有必须保留的辅酶分子或者保守水分子,亦或是功能性金属离子。可以在上一步中单独存储为pdb文件,然后在这一步上传。如果没有上述情况可以跳过此步。
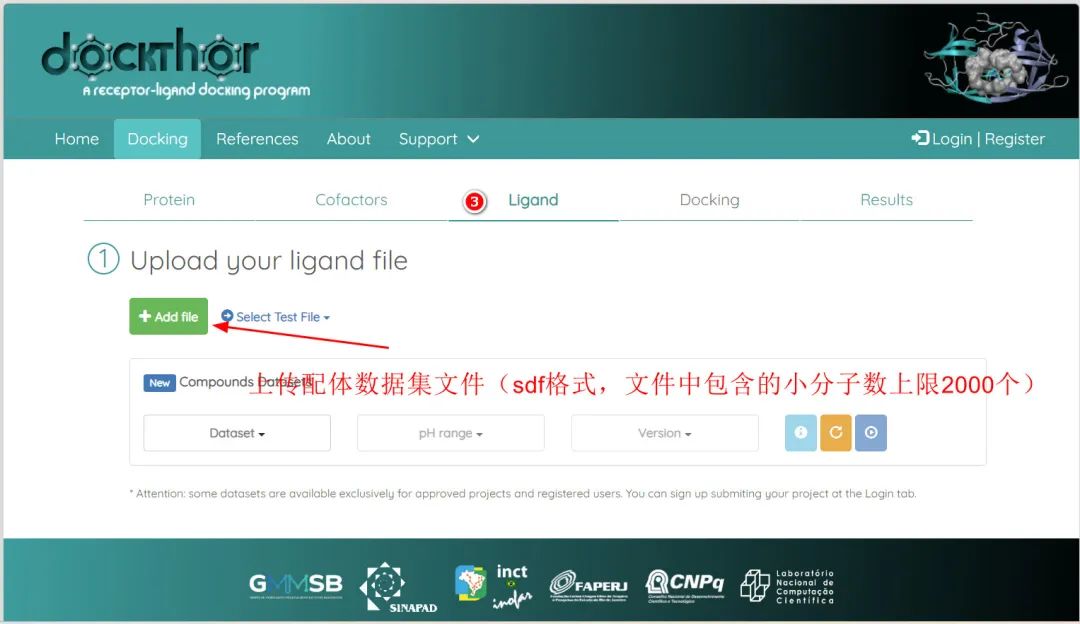
3. 上传配体文件
推荐上传sdf文件类型。该文件类型支持存储多个配体分子坐标信息。如果你不了解该文件类型,应该稍微花5-10分钟学习一下。磨刀不误砍柴工!!!
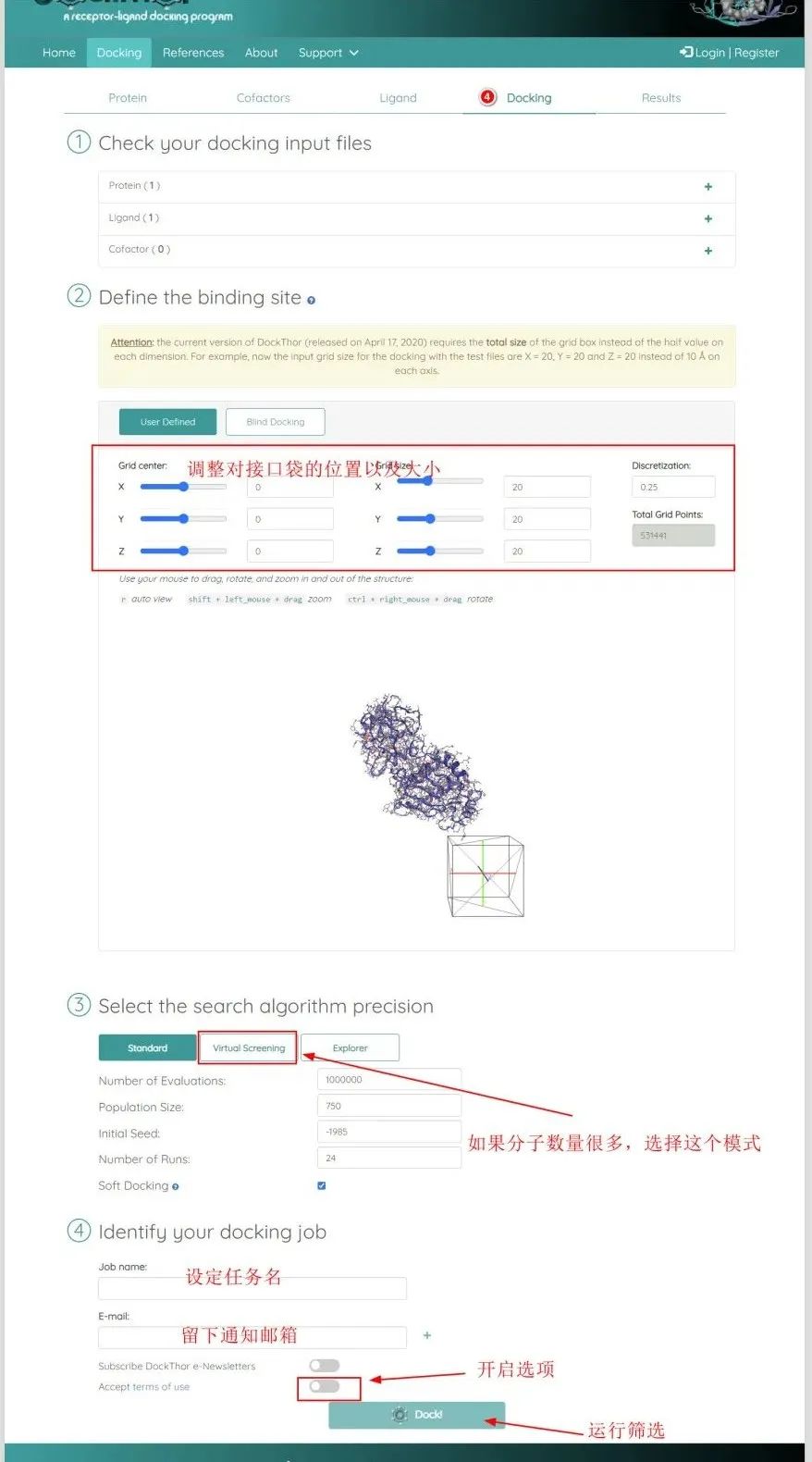
4. 虚拟筛选参数设置
-
如果你清楚地知道蛋白活性位点信息,可以选择
User Defined模式,在此模式下输入配体结合位点的信息。蛋白活性位点信息获取方式可以参考往期推送:

-
如果你不清楚小分子该结合在那里,可以选择
Blind Docking盲对接模式。
5. 结果页面
如果你所提交的小分子数量很多,则不可能很快得到结果!!!在提交任务前,强烈推荐在上一步中留下邮箱,待任务结束后你会收到邮件通知。
友情提醒:得到结果的速度不仅取决于你的任务量,还受制于同时段其他用户的提交运算量,使用前要做好心理准备!!!

