论文学习:《RNADiffFold:使用离散扩散模型的生成RNA二级结构预测》
原文标题:RNADiffFold: generative RNA secondary structure prediction using discrete diffusion models
原文链接:https://academic.oup.com/bib/article/26/1/bbae618/7907889
RNA二级结构预测方法
RNA的二级结构是通过氢键配对形成的二维拓扑结构。
RNA二级结构预测方法分为三个主要类别:基于能量的,基于协方差和基于深度学习的。
基于能量的方法主要利用实验确定的参数来计算RNA结构的自由能,并通过动态编程来识别最稳定的二级结构。该类方法被广泛采用并能够提供相对准确的预测,但这些方法受到限制,因为它们仅考虑嵌套的基础配对并受限于更复杂的结构(例如假结结构),而且随着RNA的长度的增加,这些方法的计算复杂性显著增加。
基于协方差的方法通过考虑RNA序列与结构之间的共进化关系来推断二级结构。这些方法可以在某些条件下提供高度准确的预测,但是在处理来自同源序列数据的有限信息时可能会面临挑战。
基于深度学习的方法:其中一些方法使用神经网络,如Bi-LSTM,Transformer和UNet来计算碱基配对概率,以捕获长程相互作用。另一些方法融合热力学知识,采用迁移学习策略,或融合进化和突变耦合信息,优化预测结果,缓解预测偏差。但是现有的基于深度学习的方法在模型泛化性能方面仍然存在不足,尤其是在对未知RNA家族进行建模时。这种局限性是因为它们的模型参数往往来自于有限的已知结构库,从而限制了它们对新数据的适应性。
扩散模型
扩散模型是一类概率生成模型,在扩散过程中具有两条马尔科夫链:一条将数据解构为噪声的前向链,一条从噪声中重构数据的反向链。具体来说,在扩散概率模型( DDPMs )中,给定数据分布x0~q(x0),前向扩散过程q在时间步长t处通过添加噪声产生从x1到xT的潜态序列,方差调度βt∈( 0,1 )。过渡核的定义如下:
反向扩散过程p由先验分布xT~N ( 0,I )和可学习的转移核pθ ( xt-1 | xt )参数化,定义如下:
其中,θ表示模型参数,μθ和θ分别表示t时刻分布的均值和方差。 训练目标是学习参数θ,使得储备轨迹pθ逼近前向轨迹q。它是通过优化一个关于负对数似然的变分上界来实现的:
由于引入了高斯噪声作为先验,大多数扩散模型在连续状态空间中有效运行;然而,它们可能无法有效地处理离散数据。为了解决这个问题,人们提出了一些方法来生成高维离散数据。例如,D3PM 考虑了具有吸收状态或采用离散化、截断高斯分布的转移矩阵。VQ-扩散采用惰性随机游走或随机掩蔽操作来容纳离散数据。
RNA基础模型
RNA-FM 是一种基于BERT语言模型架构的RNA基础模型,建立在基于Transformers的12个双向编码器块之上。该模型由预训练和微调两个阶段组成。在预训练阶段,RNA-FM以自监督的方式在大量未标记的RNA序列数据上进行训练,使其能够捕获潜在的结构和功能信息,并提取有意义的RNA表示。它的预训练策略与BERT类似,将代表核苷酸的15 %的碱基令牌随机掩码,并训练模型从剩余序列中重建掩码令牌。训练完成后,RNA-FM可以为每个长度为L的RNA序列生成一个640 × L的嵌入矩阵,这些嵌入矩阵为下游任务提供了丰富的特征表示。在任务特异性微调阶段,预训练的RNA-FM模型可以生成针对下游模块需求定制的序列嵌入,可直接用于各种RNA相关的机器学习任务。
值得注意的是,最近的一项研究进一步验证了RNA-FM中多头注意力机制输出对于捕获结构信息的重要性。这些注意力图不仅揭示了RNA序列中不同位置之间的关联强度,而且为理解RNA二级结构和功能提供了新的视角。
本文提出了一种新的基于离散扩散模型的RNA二级结构预测框架RNADiffFold。该框架旨在以生成式的方式预测确定性的RNA二级结构。RNADiffFold首先将RNA二级结构表示为二元接触图(碱基直接是否配对)。接触图的大小为L × L (其中L是RNA序列的长度),图中的每个点可以分为两类:'1'表示配对,'0'表示非配对。该方法将复杂的RNA二级结构预测任务简化为像素级别的图像分割任务。
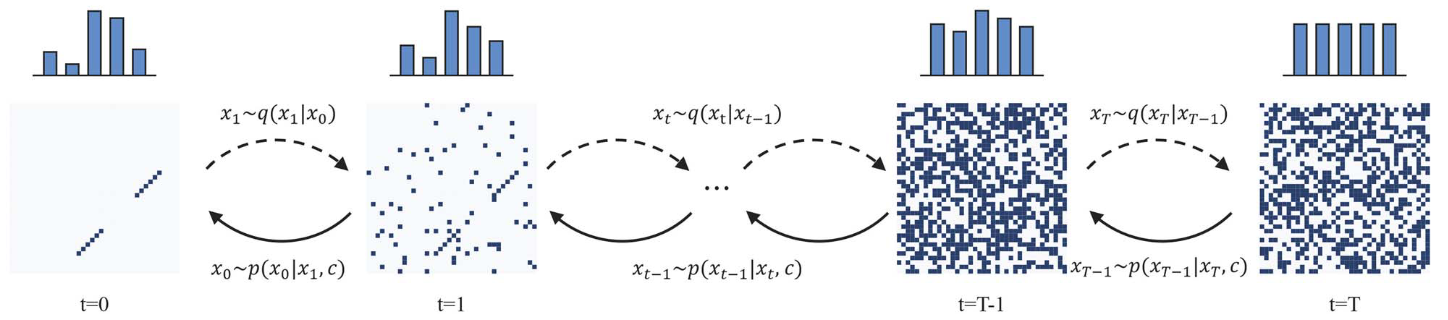
在扩散过程q ( xt | xt-1 )中,逐渐引入离散噪声,从左到右破坏接触映射。
在去噪过程pθ ( xt-1 | xt , c)中,一个生成模型从右到左学习去噪。
RNADiffFold包括两个主要部分:扩散模型和条件控制。扩散模型组件是基于离散数据空间的多项式扩散。正如图1所示,在前向扩散过程中,真实的接触映射x0通过加入服从均匀分布的噪声而逐渐退化。当到达时间步长T时,xT转变为完全随机噪声状态。在反向扩散过程中,我们采用U-Net 作为学习网络,并加入条件控制,逐步去噪,恢复原始接触图。条件控制部分包含了RNA的序列信息,包括序列的one-hot编码,来自Ufold打分网络的概率图,以及来自RNA基础模型( RNA-FM )的高维嵌入和注意力图等特征,并通过不同的MLPs进行降维。在逆扩散过程的每个时间步,将所有这些序列特征与中间状态xt进行融合。这种设计使得RNADiffFold能够利用扩散模型强大的预测RNA二级结构的能力,同时整合各种序列特征来提高预测的准确性和稳定性。
RNADiffFold的架构
如图2A所示,RNADiffFold由扩散模型组件和条件构造单元组成。在左分支中,输入的RNA序列经过条件构造单元得到四种类型的特征表示:独热编码、概率图、RNA-FM的嵌入和RNA-FM的注意力图。在右分支中,RNA二级结构表示为一个L × L的二元接触图。在扩散过程中,逐渐注入离散噪声以打乱原始接触图,经过T个时间步后,接触图转变为完全随机噪声。在反向扩散过程中,利用U-Net去噪网络进行去噪,结合条件控制单元输出的序列特征,逐步恢复原始接触图。一旦模型训练完成,给定一个随机采样的噪声xT和一个RNA序列,渐进去噪过程可以预测二级结构接触图。
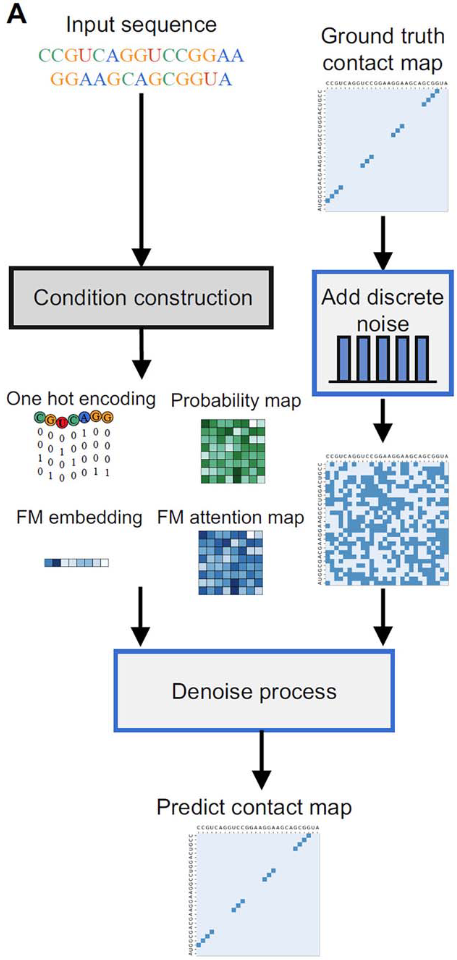
给定输入序列,我们通过条件构造阶段来构造表示。
在训练阶段,将离散噪声增量地添加到真实接触地图中。
然后,我们逐步对序列表示条件下的地图进行去噪。
在预测阶段,给定从类别分布中随机采样的序列和地图,
用不同的种子生成候选地图,并投票选出最合理的地图。
扩散过程
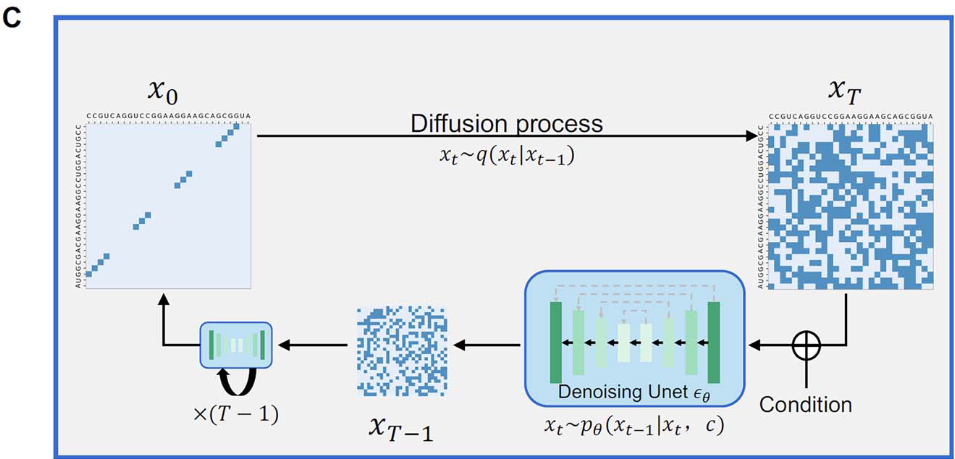
如图2C所示,RNADiffFold使用一个共享权重的神经网络来学习数据在T步上的渐进重建。RNADiffFold的扩散过程是基于多项式扩散实现的,但存在一定的差异。具体来说,该模型处理的是二值接触图,其中像素值被限制为两种表示:0和1。初始x0是一个具有确定0-1关系的L × L张量,其中L表示序列长度.为了通过U-Net进行去噪学习,将每个像素嵌入到一个8维向量中,得到一个L × L × 8的张量表示。随后,利用Gumbel - Softmax方法,通过如下定义的向前过程,在每个时间步t将离散噪声逐渐添加到样本中:
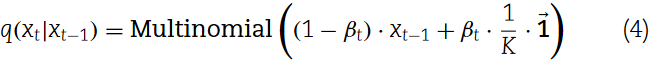
→1为全一矩阵,βt为均匀重采样另一种配对可能性的机会。
本文应用余弦时间表来避免在高噪声问题上花费许多步骤。当t趋近于T时,βt调整为近似1,使得分布更接近于均匀分布。
当一个随机过程在给定现在状态及所有过去状态情况下,其未来状态的条件概率分布仅依赖于当前状态;换句话说,在给定现在状态时,它与过去状态(即该过程的历史路径)是条件独立的,那么此随机过程即具有马尔可夫性质。
由于是马尔科夫的,因此可以直接基于x0对任意xt进行采样为:
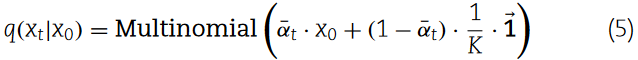
利用式(4)和式(5)可以得到如下形式的分类后验q( xt-1|xt , x0):
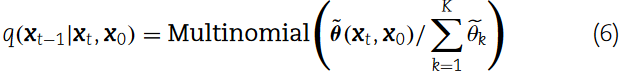
Multinomial:多项
生成扩散过程定义为:
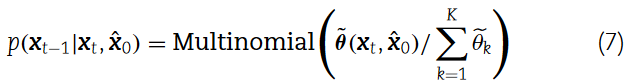
用于预测初始接触矩阵^x0给定前一步状态xt和条件c∈{ conehot,cu,cemb,cattn }。
本文利用q( xt -1 | xt , ^x0)的概率向量对p ( xt-1 | xt )进行参数化。这两个过程的主要区别在于前向扩散过程引入了数据,使其与数据或条件无关。相反,生成式扩散过程依赖于所提供的条件和对前一步的全面观察。
该扩散过程的训练目标是最小化方程7和方程6之间的期望库尔贝克-莱布勒( KL )散度,其形式与方程3类似:
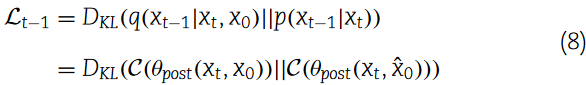
考虑到x0是one-hot编码,L0的计算可以表示为:
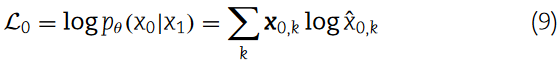
U-Net网络的去噪细节如附图S1所示。得益于U-Net处理不同大小输入的灵活性,我们还可以处理变长序列数据。
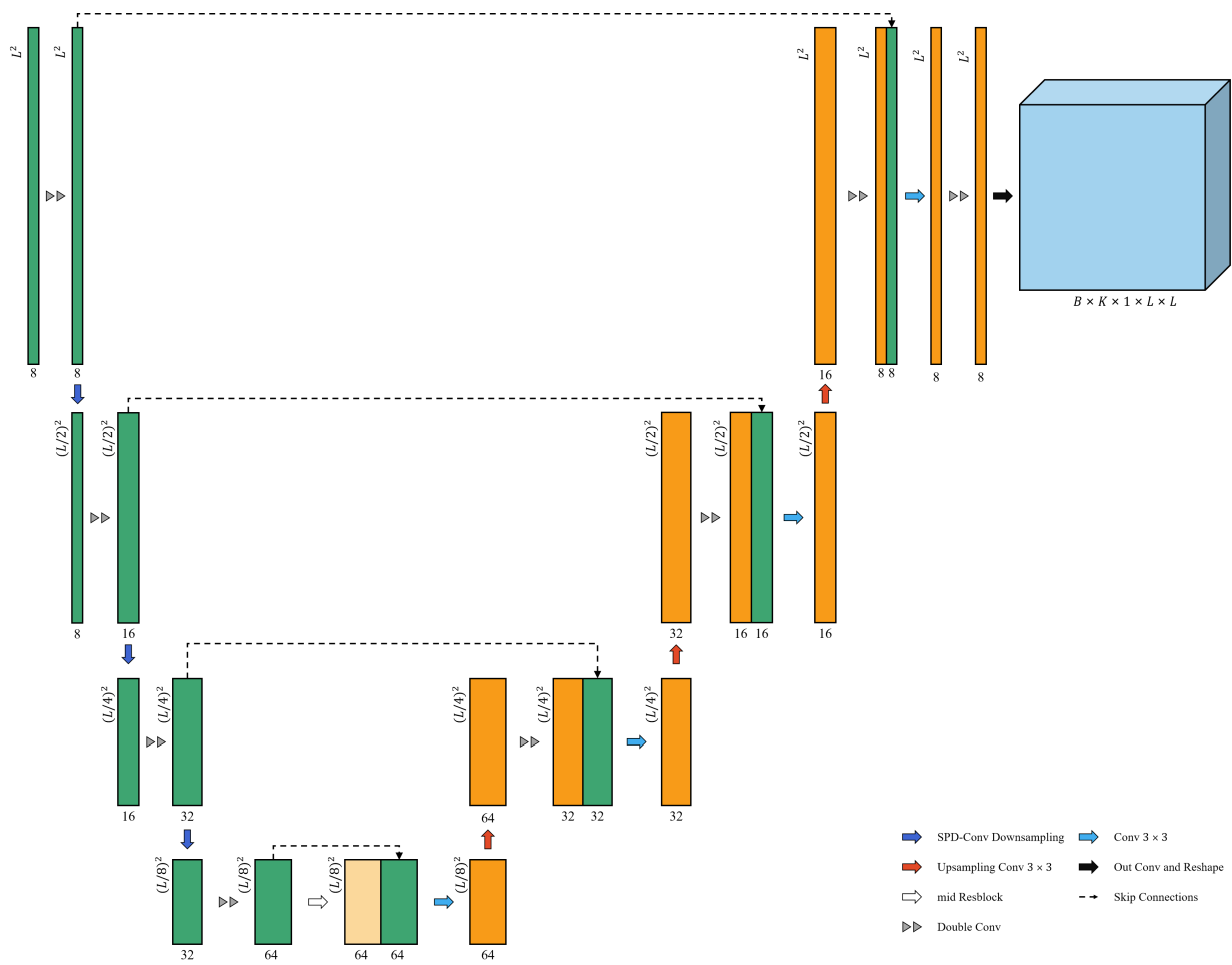
条件建设单位
经过相应的操作,得到了四种条件表示:一次热编码、概率图、FM嵌入和FM注意力图。
克罗内克积(Kronecker product)是两个任意大小的矩阵间的运算,表示为⊗。
简单地说,就是将前一个矩阵的每个元素乘上后一个完整的矩阵。
条件构建如图2B所示。为了将输入序列的特征融入到反向扩散过程中,最直观的策略是对序列进行独热编码。实验结果表明,当使用one-hot编码的特征Conehot作为条件时,RNADiffFold表现出一定的预测能力,在一些数据集上可以与当前最先进的方法相媲美。然而,当面对更为复杂的场景时,其预测性能有所下降。因此,需要构建额外的神经网络来处理输入序列,从而提取更有意义的特征,生成包含丰富额外信息的序列表示。
如图2B所示,本文采用两个神经网络作为特征提取器,即Ufold打分网络和预训练的RNA-FM模型。这里,来自Ufold的条件表示由一个概率矩阵Cu组成,而来自RNA-FM的条件表示由一个序列嵌入Cemb和一个注意力映射Cattn组成。给定一个长度为L的RNA序列,记为s = ( s1 , s2 , ... , sL),其中每个si∈{ A,U,C,G,N },N表示一个未知的状态:
Conehot是one-hot编码的序列特征,Conehot∈{ 0,1 } 4 × L。编码规则如下:A:( 1、0、0、0),U:( 0、1、0、0),C:( 0、0、1、0),G:( 0、0、0、1),N:( 0、0、0、0)。
Cu是评分网络从Ufold输出的概率矩阵。具体来说,首先,在Conehot和自身之间计算克罗内克尔乘积,然后调整维度,将Conehot转换为张量Ckronecker∈{ 0,1 } 16 × L × L。随后,为了解决类表示中的稀疏性问题,将Ckronecker与CDPFold中使用的额外配对概率矩阵进行串联,得到大小为17 × L × L的张量Cinput。因此,得到的特征表示在不施加显式约束的情况下考虑了所有潜在的配对可能性,从而能够预测更复杂的结构。最后,将Cinput输入到U-Net网络中产生概率张量Cu,其维度为8 × L × L。在此过程中,为满足扩散过程的维度要求,对U-Net网络的最后一层进行调整,使其输出维度恰好为8。值得注意的是,除了最后一层,U-Net的初始权重来自预训练的Ufold模型。随后,进行微调操作,以确保整个网络对于当前任务的最佳性能。
Cemb和Cattn分别是RNA-FM的一维序列嵌入和二维注意力图。具体来说,将RNA序列输入到预训练的RNA-FM模型中。从每个编码块的多头注意力( MHA )层得到维度为20 × L × L的注意力图,从输出层得到维度为640 × L的序列嵌入图,其中640表示嵌入维度,20表示注意力头数。随后,将来自12个编码器块的注意力图进行整合,形成最终的维度为240 × L × L的注意力图集。Cemb包含有意义的生物信息。正如文献所述,Transformer编码器模块中不同层的多个注意力头在理论上应该通过关注输入序列中不同位置之间的关联强度来捕获结构信息。因此,Cattn包含了与结构相关的信息。
与基线模型的比较
表1:RNADiffFold在长程碱基配对的TS0数据集上与其他基于学习的方法进行了F1分数的比较
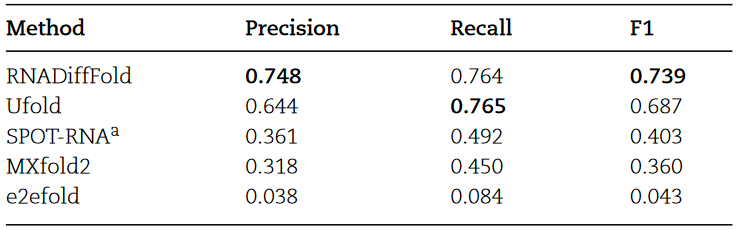
将RNADiffFold与其他主流方法进行比较,结果如表1所示,证明了RNADiffFold在长距离碱基对RNA数据上的出色表现。与UFold相比,RNADiffFold的预测准确率和召回率更接近,预测性能更稳定。这种稳定性可能源于预训练的UFold打分网络与RNA-FM输出特征的有效整合,为RNADiffFold提供了丰富的上下文和结构信息,从而提高了其预测长程碱基对的准确性。
