AM J BOT | 黄芪稳健骨架树构建
Astragalus(黄芪属,豆科,含约 3,400 种)是最大的被子植物属之一,其多样化在北半球多个地区的植被形成与生物多样性格局中扮演了重要角色。然而,由于属内物种数量庞大、形态复杂且演化历史可能受到网状进化的影响,该类群的系统发育关系长期以来一直未能得到可靠解析。研究开发并应用了一个进化枝特异性的靶向富集诱饵组(clade-specific target enrichment bait set),覆盖 819 个核基因,以便在属级尺度上获得稳健的系统发育框架。我们从 107 个样本中成功回收序列,其中包含 80 个来源于标本馆的样本(采集年代最早可追溯至 1899 年)。核基因的物种树分析为 Astragalus 提供了一个支持度极高的骨干系统树,但在某些关键节点处,基因树之间存在显著的不一致性。同时,我们利用非靶向回收的叶绿体基因组序列重建的质体树,与核树在若干位置也表现出明显冲突。进一步的网络分析表明,这些冲突可能源于不完全谱系整理(ILS)与历史上的杂交/基因流事件的叠加。
本研究证明,进化枝特异性靶向富集方法不仅能够高效回收老旧标本的核与质体数据,而且能够在“超级大属”的系统学研究中构建稳健的骨干框架。同时,我们的结果也强调:在多样化速率高、演化复杂的大属中,系统发育信号往往受到 ILS 与古代杂交的显著影响,因此必须结合共祖模型与网络方法加以解析。
文章来源:https://doi.org/10.1002/ajb2.70084
一 方法
总体设计与目的
使用为 Astragalean 设计的 819-locus clade-specific bait set,以标本馆样本为主(有的标本采集于 1899 年),结合公开转录组数据,重建 Astragalus 的核骨干树并比较核/质体信号以检测 cytonuclear 冲突与网状(reticulate)演化证据。
1. 取样(Taxon sampling)
总共选取 ~107 个样本(作者表述略有处),其中 80 个为本研究新测(77 属于 Astragalus,3 属于其它 Astragalean 属用于对照),并整合公开转录组/基因组数据以补充序列。
2. DNA 提取与文库构建(Herbarium-friendly wet lab)
从 ~20 mg 干标本组织提取(NucleoSpin Plant II),为老标本对步骤作了具体修改(如:600 μL PL1、无 RNase、1.5 h 裂解、额外清洗、50 μL 洗脱),用 Qubit 和 TapeStation 质控。
声波断裂至 ~350 bp(Covaris M220),NEBNext Ultra II 文库构建,接头后按样本片段大小微调筛选,PCR 扩增 8 周期。16 联样 pooled,myBaits 富集(hybridization temp = 60°C,wash temp = 60°C),富集后再扩增 10 周期;测序使用 Illumina NextSeq 1000。
3. 读段质控与组装(Read QC & locus assembly)
FastQC / MultiQC 检查;去 PCR 重复(ParDRe);Trimmomatic 去接头并丢弃 Q < 20 或 <25 bp 的读段。
用 HybPiper v2.1.5 进行靶位组装:
SPAdes 的 coverage cutoff 为每样本单独决定(先跑初始组装再计算平均覆盖);
Diamond mapping(
--diamond),Exonerate 和 sliding-window 阈值设为 85(--thresh和--exonerate_hit_sliding_window_thresh= 85);处理 chimeric 相关参数(
--chimeric_stitched_contig_edit_distance = 0等);并用 HybPiper的paralog_retriever回收长 paralog。
4. 同源/正交映射与基因筛选(Orthology inference)
序列用 MACSE 对齐;用 Pxclsq 清理碱基列(核数据 min occupancy 10%);用 TreeShrink 去除异常长分支,mask 单系/并系 tip(保留信息量最多的 tip)。
正交推断采用 monophyletic outgroup (MO) 方法(Yang & Smith 2014)与 Morales-Briones 等(2022)流程生成同源/正交 fasta
5. 基因树与物种树构建(Phylogenetic inference)
基因树:IQ-TREE v2(ModelFinder
-m TEST,1000 ultrafast bootstrap)。物种树(nuclear):并行使用(a)ASTRAL(quartet-based coalescent 方法)、(b)ASTRAL-Pro(可处理多拷贝/并行基因)和(c)concatenated ML(把所有 ortholog 拼接成 supermatrix 用 IQ-TREE)。这样一套并行策略可对比 ILS / paralogy 的影响。
6. 基因不一致性与网状演化检测(Discordance & networks)
PhyParts(统计基因与物种树的一致/不一致)与 QuartetSampling (QS)(评估四分体支持和替代拓扑频率)用于量化基因树不一致。
PhyloNet(maximum pseudo-likelihood)在代表性 22 个样本(覆盖 11 大类群、576 个基因)上推断带 reticulations 的物种网络,检验 0–6 个杂交事件(reticulations)情形并以 log-likelihood 比较模型优劣。
7. 叶绿体(plastome)数据的回收与分析
利用 off-target reads 用 FastPlast / SPAdes 组装 plastome,导入 Geneious 以 A. pattersonii(NC_063490)为参考做高灵敏 mapping,手工去除 discordant contigs,合并并对 CDS/分区建树(IQ-TREE),并用 Pxclsq 清除列缺失 >40% 的位点。这样得到独立的 plastome 树,用以对比核树。
二 结果
1) 测序与靶位回收(数据量与覆盖)
每样本原始 paired-end reads 在 1.2M 到 12M 之间。
平均每样本 ≥75% 长度回收的靶位为 701.1 个(范围 484–767),表明 bait set 对许多老标本亦有效。且作者未检测到标本年代(1899–2014)与回收位点数的明显相关性(Supporting Fig. S1)。
2) 并行/副本与正交序列数
HybPiper 报告的 paralog warnings 平均 33.4 个/样本(范围 3–252),显示在某些样本/基因上并行拷贝问题明显。平均每个样本检索到 ~599.8 个 ortholog 序列(范围 230–718)。最终筛得 781 个 MO orthologs(每个基因至少含 20 个样本)。
3) 矩阵规模(拼接矩阵)
拼接超级矩阵长度 778,623 个对齐碱基,总体矩阵占有率约 73%(约 27% 缺失)。叶绿体矩阵为 ~114,580 列,对齐占有率约 74.6%。
4) 核树(ASTRAL / ASTRAL-Pro / concatenated)– 总体拓扑与支持
三种方法(coalescent、ASTRAL-Pro、concatenated ML)产生的主干拓扑非常相似,背骨(backbone)节点普遍具有很高支持(LPP=1 / BS=100 在许多关键节点)。Astragalean 整体与 Eu-Astragalus 被强烈支持为单系群。
部分细节差异:ASTRAL-Pro 在某些深节点(例如 Hypoglottis)与 concatenated 方法给出略异结论(ASTRAL-Pro 使 Hypoglottis 单系),另一些类群(Astracantha、Hamosa)的支持值在方法间也存在差别(见文中数值)。
5) 核—质体(cytonuclear)不一致
虽然核树给出高支持的 backbone,但 plasmome(叶绿体)树在少数关键处与核树冲突:例如 Ophiocarpus 在核树中呈散布/多系,而叶绿体树支持 Ophiocarpus 单系且与 Glottis 为姐妹(BS=100)。这提示曾发生器官体交换或古代基因流/捕获事件。
6) 基因不一致量化(PhyParts & QuartetSampling)
某些大节点表现出高度一致性(例如 Astragalean:628/628 informative genes concordant,QS score = 1/–/0.96;Eu-Astragalus + Oxytropis:728/741,QS = 1/–/0.96),显示这些节点几乎所有基因都支持相同拓扑。
然而,沿 Astragalus 背骨(尤其是 Meso-Astragalus 区域)存在高水平基因树不一致与 QS 对替代拓扑的强烈偏斜。举例:Hypoglottis 与 Diholcos 这一节点,只有 23 个 informative genes(out of 668) 给出该 QS 指标 0.27/1/0.48,虽然物种树的支持值仍显示 LPP = 1 / BS = 100——说明高支持度的节点可能由少数强信号或合并效应驱动,但基因层面替代拓扑频率不低。
7) 网状演化证据(PhyloNet)
在用 22 个代表性类群(576 loci) 进行 PhyloNet 分析时,作者检验了 0–6 个 reticulation 的模型,发现最优模型为 6 个 reticulations(log-likelihood ≈ –392,572),并在多个主类群间重建出复杂的 reticulation 事件。
许多推断出的杂交事件涉及 “ghost lineage”(未采样或已灭绝谱系) 或祖先节点(例如 Trimeniaeus 与 Neo-Astragalus 祖先的相互作用),且某些事件在不同 reticulation 假设下重复出现(例如 Trimeniaeus ↔ Diholcos),增强了“真实存在历史混合”的可信度;作者仍建议更密集取样以稳健确认细节。
8) 质控与其他观察
标本年代不显著影响基因回收量(支持用标本馆样本做大规模靶位富集的可行性)。
并行/副本(paralogy)在 Astragalus 中较为常见(个别样本 paralog warnings 高达数百),因此作者采用 HybPiper 的 paralog_retriever、ASTRAL-Pro 等工具来降低 paralogy 对物种树推断的干扰。
三 关键发现
1.系统发育重建:核基因数据(781个直系同源位点)支持黄芪属分为11个主要分支,但发现Ophiocarpus分支在核基因组中多系分布,而在叶绿体基因组中单系。
2.核质冲突:叶绿体系统发育与核基因组存在显著不一致,特别是在Ophiocarpus、Hypoglottis和Diholcos等分支的拓扑结构上。
3.网状进化证据:系统发育网络分析(PhyloNet)检测到至少6次杂交事件,主要发生在Meso-Astragalus类群间,遗传贡献概率在0.135-0.4之间。
四 关键图解析
图1:基于ASTRAL的黄芪属物种树
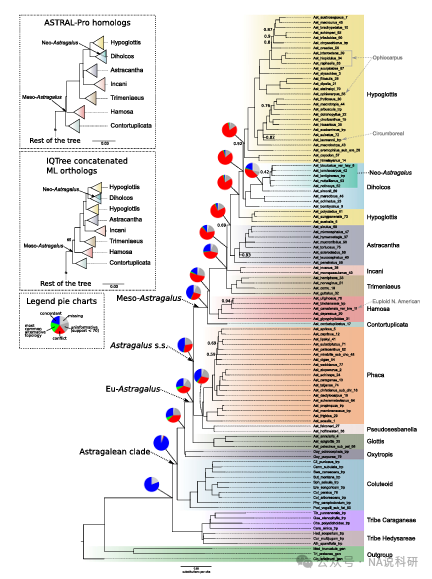
基于ASTRAL软件构建的黄芪属种系树,采用781个正交基因座数据。节点支持度(局部后验概率,LPP)≥0.95(未特别标注时)。饼图显示主干节点处基因不一致性(由PhyParts计算)。欧黄芪属内各分支的彩色标注名称遵循Azani等(2017)及Su等(2021)的命名体系。插图展示主干沿线主要差异,采用标注方法进行比较,省略拓扑结构相同的树形部分。
图2:核质基因组冲突与网状进化证据
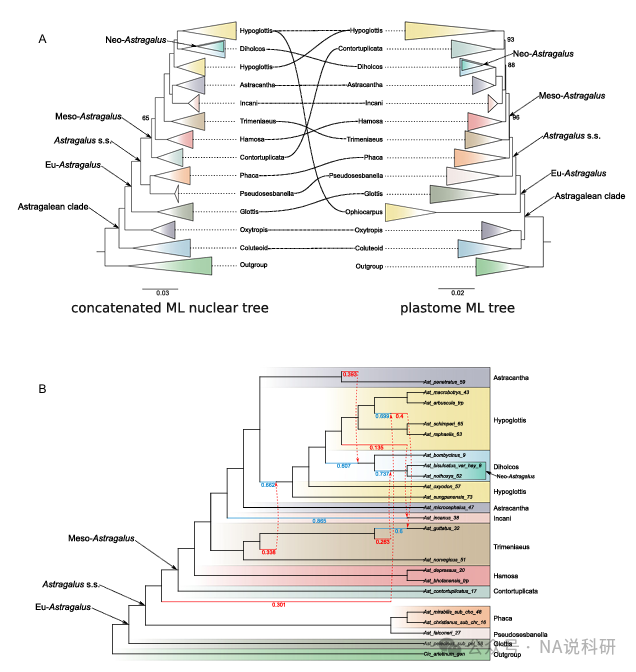
图2A:核质基因组拓扑结构比较
核基因组树(左图)特征:
Ophiocarpus分支:多系分布于Hypoglottis类群中(红色标记),与形态学分类冲突
Diholcos分支:嵌套于Hypoglottis内部,形成两个独立的高支持度亚支(BS=100)
Contortuplicata分支:与Hamosa分支形成姐妹群关系
叶绿体树(右图)特征:
Ophiocarpus分支:单系且与Glottis分支形成姐妹群(BS=100)
Hypoglottis分支:单系且与Contortuplicata分支姐妹关系
Diholcos分支:与Astracantha分支姐妹关系
关键冲突点:
Ophiocarpus的单系性(叶绿体)vs多系性(核基因)
Diholcos在核基因组中嵌套于Hypoglottis,而在叶绿体中与Astracantha关联
Contortuplicata与不同分支的关联关系
图2B:系统发育网络分析
网络结构特征:
主要杂交事件:6个明确信号(红色箭头),涉及Trimeniaeus、Hypoglottis和Diholcos等分支
遗传贡献概率:
Trimeniaeus→Hypoglottis+Diholcos共同祖先(0.3)
灭绝谱系→Contortuplicata(0.3)
Trimeniaeus→Neo-Astragalus祖先(0.4)
图3:与他人研究的比较
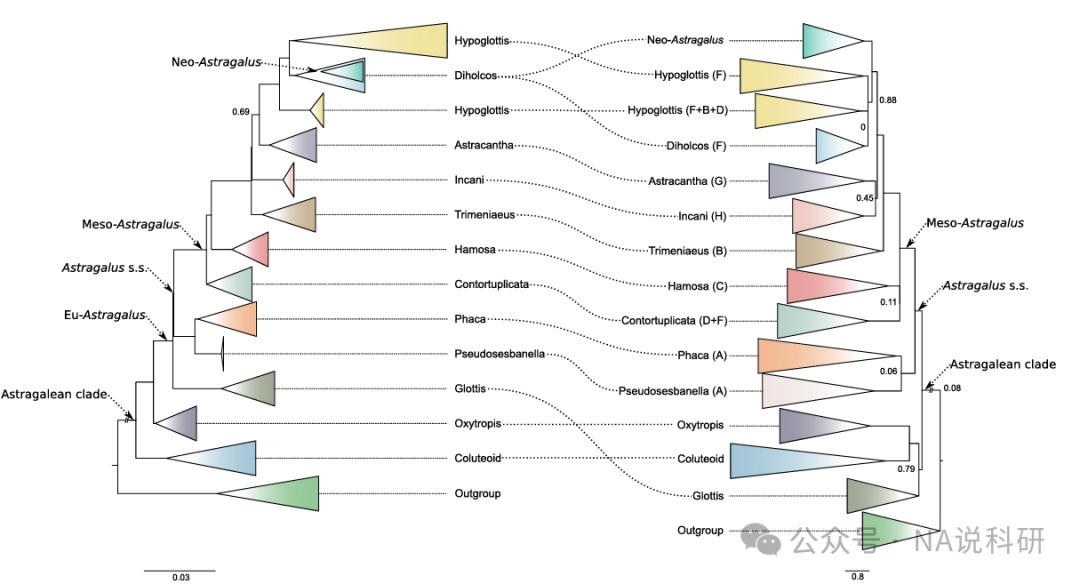
左图:本研究结果,Glottis分支(蓝色)位于Eu-Astragalus基部
右图:Folk等人的结果,Glottis代表种A. epiglottis被置于Astragalean clade之外
差异:节点支持度(LPP)和Neo-Astragalus的位置存在显著不同
