【数据分析】一种用于校正微生物组数据中批次效应的多变量框架
禁止商业或二改转载,仅供自学使用,侵权必究,如需截取部分内容请后台联系作者!
文章目录
-
- 介绍
- 加载R包
- 数据描述
- 导入数据
- 数据预处理
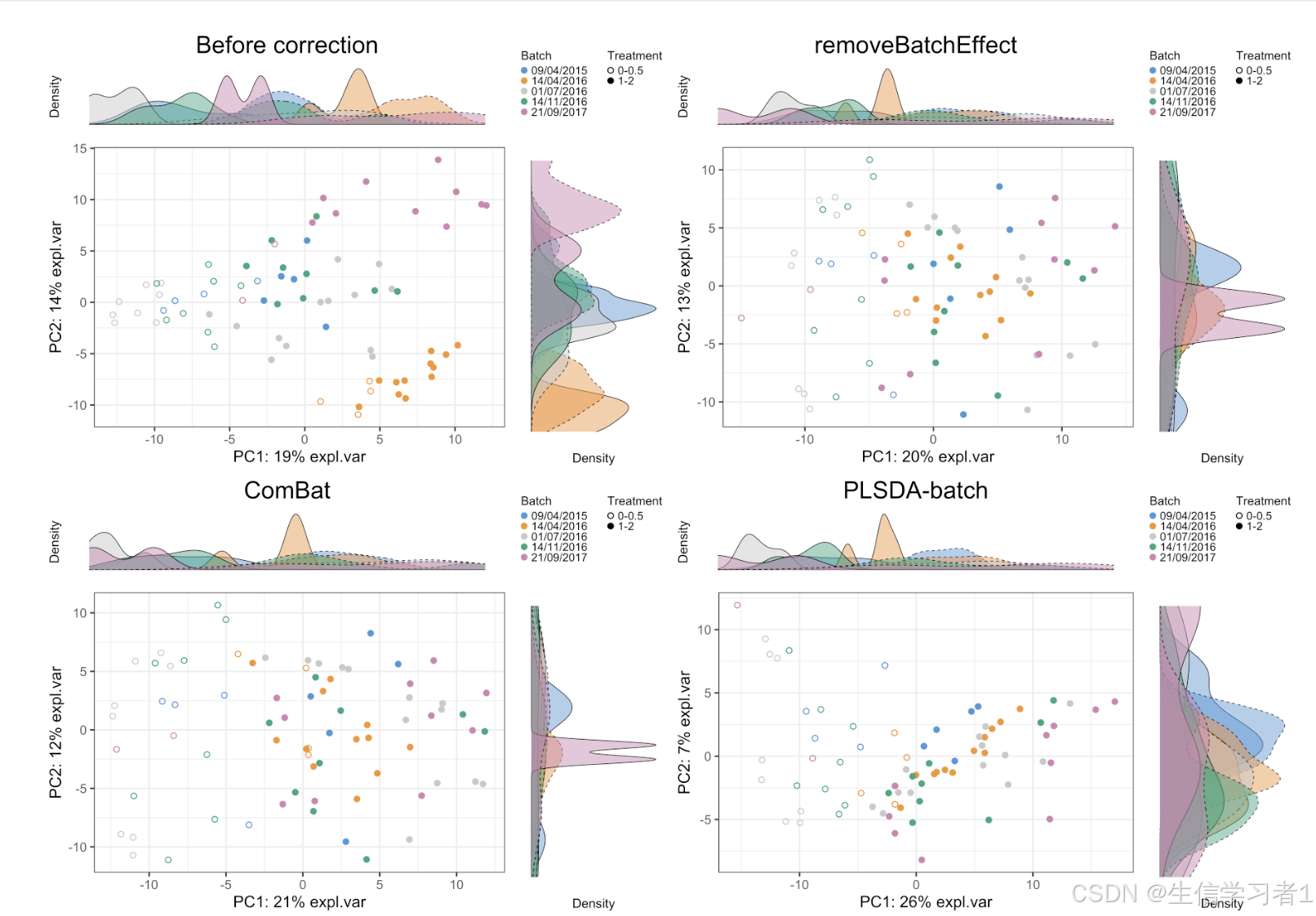
- 识别批次效应
-
- PCA
- 密度图
- 线性回归
- 热图
- pRDA
- 处理批次效应
-
- 零膨胀高斯混合模型
- 其他降低批次效应方法
- 矫正批次效应
-
- removeBatchEffect
- ComBat
- PLSDA-batch
- sPLSDA-batch
- Percentile Normalisation
- RUVIII
- 评估批次效应
-
- PCA
- pRDA
- 选择特征
- 总结
- 系统信息
- 参考
介绍
近年来,研究微生物组成与表型之间的关系已成为研究的热点,因为微生物在农业、医疗保健、食品生产、工业和气候变化等众多领域中发挥着关键作用(C. H. Wang 等人 2020 年;Ray 等人 2020 年;Fan 和 Pedersen 2020 年;Poirier 等人 2020 年)。特定环境中的微生物及其基因组的集合被称为微生物组(Marchesi 和 Ravel 2015 年)。可以通过 16S rRNA 基因测序或全基因组测序来分析微生物组。微生物组数据以每个分类群的每个样本的计数丰度表的形式呈现。这类数据具有其固有的特征,包括零膨胀、不均匀的文库大小、组成结构和多变量性质,这些限制了统计分析。
微生物组研究面临着不同研究之间结果可重复性的挑战。其潜在原因在于实验设计欠佳以及缺乏严格的操作流程,这会导致数据出现偏差(即批次效应),从而掩盖了我们所关注的效果。由于微生物群落具有动态特性,微生物组数据极易受到批次效应的影响(Y. 王和勒考 2020 年)。众多研究都报告了由测序批次引入的批次效应(希肯等人 2016 年)、独立研究的纳入(杜瓦莱特等人 2017 年)、地理因素、年龄、性别、健康状况、压力和饮食等因素(吉布森等人 2004 年;洛祖普诺等人 2013 年;哈罗等人 2016 年;金等人 2017 年)。人们曾试图通过标准化设计来减
