比较转录组-油料作物-文献精读133
Comparative transcriptomic analysis provides insights into the genetic networks regulating oil differential production in oil crops
比较转录组分析揭示了油料作物中调控油脂差异性合成的基因网络机制
摘要
背景 植物种子油含量(SOCs)存在三倍以上的差异。大豆(Glycine max)、棉花(Gossypium hirsutum)、油菜(Brassica napus)和芝麻(Sesamum indicum)是四种重要的油料作物,它们在种子油含量和脂肪酸组成上有显著差异。
结果 与玉米和水稻等谷物作物相比,油料作物中扩展的酰基脂质代谢基因和相对较高的种子油合成(SOS)相关基因表达水平促进了种子油的积累。在本研究中,我们对具有两种不同种子油含量材料的油料作物进行了比较转录组分析。共同发现,二氢脂肪酰胺脱氢酶、硬脂酰酰基载体蛋白脱饱和酶、磷脂:二酰甘油酰基转移酶以及油体蛋白基因在每种作物的高油和低油材料之间都有差异表达。通过比较SOS网络的功能组成,我们发现“糖酵解/葡萄糖异生”和“脂肪酸合成”中的基因之间的强相关性在谷物和油料作物中都得到保留,其中丙酮酸激酶是影响淀粉和脂质积累的共同因素。网络比对还发现油料作物中影响种子油积累的保守聚类,这一发现已在拟南芥中得到了验证。不同之处在于,次级代谢和蛋白质代谢在不同作物中对油脂合成的影响程度不同,高油含量是由于相同前体物质的竞争较少。通过比较拟南芥突变体与野生型,发现我们识别出的保守调节因子——肉桂醇脱氢酶9是导致种子中木质素与油脂相对含量不同的因素。脂质和蛋白质的相互联系在作物之间是共同的,但表现出不同的方式,这在一定程度上导致了油脂生产的差异。
结论 本研究超越了对单一物种研究的观察,从多物种的角度提供了关于哪些基因和网络可能对种子油积累至关重要的新见解。
背景
油料作物为人类饮食、动物饲料和工业生产提供了丰富的可再生植物油和蛋白质来源。根据美国农业部发布的油料种子生产、供应和分布报告(数据截至2022年4月),在过去5年中,全球植物油消费量已达到2.09亿公吨,增长了9.1%,其中棕榈油(35.3%)、大豆油(28.5%)、油菜油(13.9%)和葵花籽油(9.5%)占据了较大的市场份额。因此,为了满足快速增长的食品需求和非食品利用,提高油脂生产力和改善种子油质量是油料作物育种的主要目标。自然选择已产生了种子油含量(SOCs)差异显著的作物,提供了通过比较生物学研究油脂积累大幅变化潜在机制的机会。大豆(Glycine max)、棉花(Gossypium hirsutum)、油菜(Brassica napus)和芝麻(Sesamum indicum)是四种传统的油料作物,作为食用油来源,它们的种子油含量依次为20%、30%、40%和60%[1, 2]。大豆油是最广泛消费的食用油,富含多不饱和脂肪[3]。棉籽油含有大量饱和脂肪酸(FAs),使其比其他食用油更稳定[4]。另一个重要的油料作物是油菜,其富含不饱和脂肪[5],但也含有较高水平的芥酸和芥菜甙[6],这些物质有害并严重限制了油菜作为食用油和油饼的应用。芝麻则是油脂含量最高的作物,被认为是富含油酸(18:1)和亚油酸(18:2)的丰富来源[7]。这四种油料作物的种子油含量和脂肪酸组成差异显著,为研究种子油积累提供了吸引人的模型。
植物油以三酰甘油(TAGs)的形式储存在高等植物的种子中。许多涉及酰基脂质代谢(ALM)的基因在拟南芥中已有详细描述[8]。种子油合成(SOS)中酶的基因工程成功地调整了脂肪酸的水平和组成,如二酰甘油酰基转移酶(DGAT)[9, 10]、磷脂:二酰甘油酰基转移酶(PDAT)[10]和乙酰辅酶A羧化酶[11, 12]。然而,植物脂肪酸和脂质的合成、去饱和、储存和降解途径涉及一系列复杂且相互连接的多酶系统网络,基因家族成员之间的补偿机制也使得某些基因突变体没有表现出种子油表型[13, 14]。更重要的是,使用基因操作在模式植物中实验性实现的油脂积累增加,与物种间SOCs的差异相比,仍显得微不足道。此外,种子储存的主要成分是淀粉、脂质和储存蛋白,种子中主要储存化合物的分解代谢和合成代谢过程是耦合的,并处于动态平衡状态,这在一定程度上通过代谢水平的碳分配进行严格调控[15, 16]。因此,深入研究种子中物质的分配和积累是提高油料作物的重要方向。对油料作物进行大规模的比较转录组分析,研究它们在种子油含量上的差异,将有助于揭示油脂积累的标志性机制。
随着下一代高通量测序的常规应用,物种间的比较研究为发现调控相同性状的共同进化基因提供了新的视角。例如,玉米KRN2和水稻OsKRN2受到趋同选择,并通过相似的途径增强谷物产量[17]。G基因在大豆、水稻和番茄中具有保守功能,调控种子休眠[18]。与此同时,网络方法目前被用于通过探索观察到的基因产物之间的关系来研究各种生物系统,弥合从单个基因到系统生物学的差距。共表达网络的系统分析表明,基因模块在遥远物种之间可以高度保守[19]。
在本研究中,我们收集了四种种子油含量显著不同的油料作物的全面RNA-seq资源,包括大豆、棉花、油菜和芝麻(图1a),并进行了比较转录组分析,研究影响种子油合成和积累的保守机制的差异。此外,还将玉米(Zea mays)和水稻(Oryza sativa)纳入分析,以提供关于种子储存之间相互转化的见解。研究结果旨在丰富和扩展脂质生物合成代谢机制,帮助育种家通过基因和代谢工程改良油脂含量,并设计具有理想脂肪酸组成的油料作物。
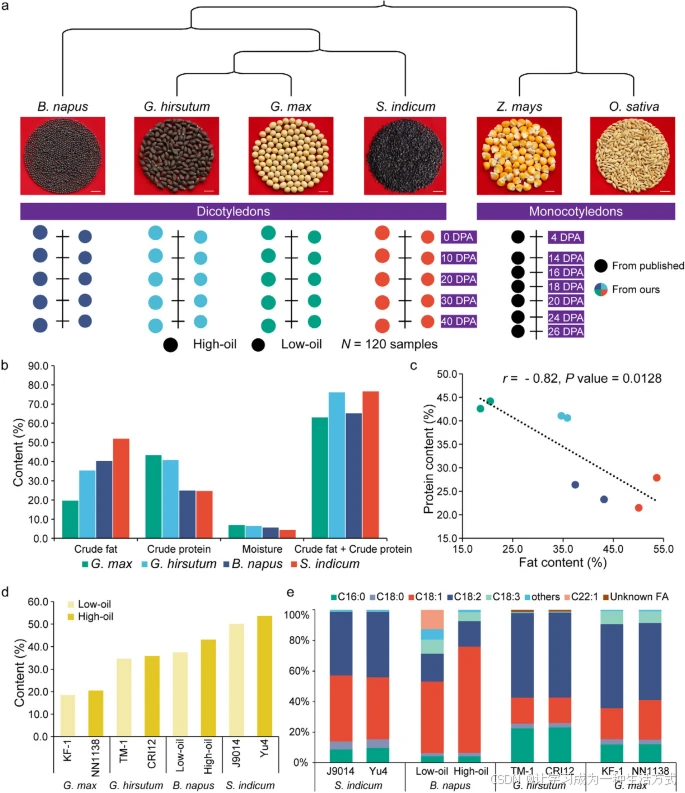
分析物种及种子成分含量概述
a 比较转录组分析所采样的不同物种种子。样本采自四种油料作物高油与低油材料的五个不同发育阶段。玉米的发育转录组数据源自已发表的研究。 b 成熟种子的粗脂肪、蛋白质和水分含量(占种子干重的百分比)。 c 油料作物种子中脂肪与蛋白质含量的相关性。Pearson 相关系数 r = −0.82,P 值 = 0.0128。 d 四种油料作物高油与低油材料的种子油含量(占种子干重的百分比)。 e 四种油料作物成熟种子的脂肪酸组成含量。
结果
四种油料作物的种子油含量(SOCs)与脂肪酸(FAs)组成存在差异
本研究纳入了每种油料作物的高油与低油材料,以进行物种间与物种内的交叉比较,并评估了其成熟种子的粗蛋白、脂肪、水分含量和脂肪酸组成。与谷物作物不同,这些油料作物种子中沉积的碳主要以脂肪和蛋白形式存在,平均占各物种种子干重的 63%–77%(图1b与附加文件1:表S1)。大豆、棉花、油菜和芝麻种子的粗脂肪含量在 20%–52% 之间变化,而其粗蛋白含量则依次从 43% 降至 25%(图1b与附加文件1:表S1),显示脂肪与蛋白质含量之间存在显著负相关关系(Pearson r = −0.82,P = 0.0128,图1c)。在每个物种中,高油与低油材料的 SOC 也存在差异(图1d)。
脂肪酸组成分析显示,棕榈酸(C16:0)、油酸(C18:1)和亚油酸(C18:2)是这些作物种子中三种主要脂肪酸,总占比达 69%–94%,但各物种之间的比例有所不同(图1e与附加文件1:表S2)。在大豆和棉籽油中,亚油酸(C18:2)含量分别为 52.5% 和 54.7%,是油酸(C18:1)含量(分别为 23.1% 和 16.7%)的两到三倍。油菜中油酸含量为 58.0%,约为亚油酸(17.4%)的三倍。芝麻中油酸(42.0%)与亚油酸(42.1%)几乎相等(图1e与附加文件1:表S2)。除了亚油酸与油酸的比例外,不同作物还表现出各自的脂肪酸特征。例如,棉籽油中饱和脂肪酸含量为 26.2%,显著高于其他作物(芝麻为14.6%,油菜为6.6%,大豆为15.3%)(附加文件1:表S2)。大豆与油菜中α-亚麻酸(C18:3)含量(8.1%、7.5%)也远高于棉籽(0.4%)与芝麻(0.3%)(图1e与附加文件1:表S2)。我们还发现,各作物高油与低油材料的脂肪酸谱相似,唯一的例外是高油油菜中未检测到芥酸(C22:1),而低油油菜中含量为12.6%(图1e与附加文件1:表S2)。这些结果表明,由于遗传和生理背景的相似性,各物种脂肪酸组成较为稳定。总体而言,不同的种子油含量和脂肪酸组成差异为深入研究油脂合成和积累的遗传基础提供了良好模型。
更高的基因数量与表达水平塑造种子油表型
基因家族的数量变化及其差异表达是驱动植物形态和代谢多样性的主要因素,并在进化与发育过程中发挥重要作用。在拟南芥中,已有773个参与TAG、叶绿体脂肪酸、内膜脂质、生物储存等16条途径的酰基脂代谢(ALM)基因被鉴定(含5个伪基因)[8](数据截止2020年7月)。通过比对这些ALM相关基因,分别在大豆、棉花、油菜、芝麻、玉米和水稻中鉴定到1081、1450、1883、612、619和548个与拟南芥同源的候选基因[20–25],占各物种蛋白编码基因总数的1.40%–2.25%(图2a,附加文件1:表S3,附加文件2:数据集S1)。其中芝麻在油料作物中ALM基因比例最高(2.25%)(附加文件1:表S3)。拟南芥用于验证分析流程的有效性与准确性(详见“方法”)。与以淀粉为主(约占80%)的水稻相比,油料作物中ALM相关基因数量显著更多(P < 0.01,双尾Fisher检验,图2a与附加文件1:表S3),而玉米与水稻之间无显著差异(P = 0.05910,图2a与附加文件1:表S3)。不过,7个物种中ALM基因按16条代谢路径分类的比例分布相似(图2b)。
以拟南芥为双子叶植物对照,油料作物之间ALM相关基因家族的比较显示,油菜在基因家族成员数量上变异最大(附加文件3:图S1)。这些变异主要集中于TAG合成相关基因,例如油体蛋白(OBO)家族基因(油菜25个 vs 拟南芥8个)、磷脂酰胆碱:二酰甘油胆碱磷酸转移酶家族基因(油菜8个 vs 拟南芥2个)(附加文件3:图S1)。基因家族扩张与收缩分析也表明,油菜中多个脂质代谢相关基因家族快速扩张(分支特异性P < 0.01,家族整体P < 0.05),包括蛋白酶抑制剂/种子储存蛋白/LTP家族(PF00234)、烯酰基-酰基载体蛋白还原酶(PF13561)、膜结合O-酰基转移酶家族(PF13813)、GDSL样脂肪酶/酰基水解酶(PF00657)、丙酮酸激酶(PF00224)及2型磷脂酸磷酸酶超家族(PF01569)(附加文件3:图S1与附加文件4:数据集S2)。此外,大豆中GDSL编码基因也显著扩张,并表现出较大的变异(分支特异性P = 0.000150,附加文件3:图S1与附加文件4:数据集S2)。
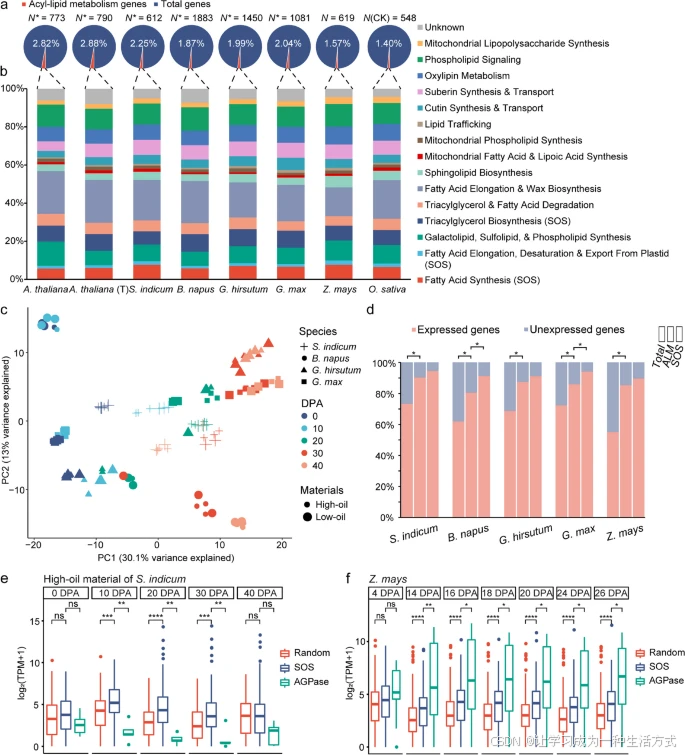
油料作物与玉米的基因数量与转录组模式比较
a 各物种中已鉴定的酰基脂质代谢(ALM)基因在全部基因中的占比。图中 N 表示各物种中 ALM 基因的总数,饼图上的数值表示 ALM 基因占该物种所有基因的比例。不同物种与水稻之间的比较通过双尾 Fisher 精确检验进行,* 表示 P ≤ 0.05。 b 各物种中参与 ALM 16条代谢通路的基因百分比。 c 基于每个物种表达变异最大的 500 个基因进行的主成分分析(PCA)。每个点代表每个物种中不同发育阶段材料的生物学重复。 d 不同作物中总基因、ALM 基因、SOS(种子油合成)基因的表达比例。* 表示 P ≤ 0.05,双尾 Fisher 精确检验。 e, f 芝麻和玉米高油材料中在不同发育阶段随机基因、SOS 基因与 AGPase 基因的表达水平比较。ns 表示 P > 0.05,* 表示 P ≤ 0.05,* 表示 P ≤ 0.01,** 表示 P ≤ 0.001, 表示 P ≤ 0.0001,Wilcoxon 检验。
系统分析种子油积累的转录组特征
我们从四种油料作物中采集了覆盖高低油材料的 0、10、20、30 和 40 天开花后(DPA)胚珠样本。过滤掉低质量和过短的 reads 后,从 51.9 亿条原始 reads 中获得了总计 46.1 亿条干净 reads,覆盖 120 个发育中种子样本(附加文件5:数据集S3)。作为对照,玉米在 4、14、16、18、20、24 和 26 DPA 的种子转录组数据来自公开文献 [26]。油料作物样本的平均比对率为 85.40%(附加文件5:数据集S3),并采用相同流程分析玉米数据(详见“方法”)。各物种样本的 Spearman 等级相关系数均超过 85%(附加文件3:图S2–S6)。主成分分析中,第一主成分(PC1,解释 30.1% 表达变异)根据种子发育阶段区分样本,第二主成分(PC2)按物种区分样本(图2c与附加文件3:图S7)。在各物种的 PCA 中,大多数变异(84.23 ~ 91.80%)都与发育阶段相关(附加文件3:图S7)。多物种共表达网络的转录组分析流程如附加文件3:图S8所示。
基于各物种中鉴定的 ALM 基因,我们发现这些 ALM 基因(在至少三个文库中有十个以上 reads)表达比例均较高,芝麻最高(90.36%,图2d)。其中,“脂肪酸合成”、“脂肪酸延伸、去饱和和从叶绿体输出”以及“三酰甘油生物合成”中的基因直接参与种子油合成,归为 SOS 基因。这些基因在油菜和大豆中的表达比例尤高(图2d)。与油料作物和玉米中的随机基因相比,SOS 基因的表达水平明显更高,且在不同油料作物中持续时间不同,反映出油脂积累关键阶段的差异(图2e与附加文件3:图S9)。相反,玉米中编码淀粉生物合成关键酶 ADP-葡萄糖焦磷酸化酶(AGPase)的基因表达量显著高于 SOS 基因,而在油料作物中无此差异(图2e, f 与附加文件3:图S9),显示谷物作物与油料作物在物种间存在显著差异。
种间油脂积累调控因素的差异与共性
为揭示物种内油脂积累的变异来源,我们对各油料作物的高油与低油材料进行了差异表达分析(DEA)。基于差异表达基因(DEGs)的功能富集,我们提取出与脂质积累密切相关的关键通路,包括光合作用、碳水化合物代谢、脂质代谢、氨基酸代谢、次级代谢和蛋白质代谢。结果显示,光合作用、次级代谢与蛋白质代谢在高油与低油材料间存在显著差异(附加文件3:图S10)。
我们还在相邻发育阶段之间对油料作物样本和 PCA 中差异明显的玉米样本进行了差异表达分析(附加文件3:图S7)。在多个油料作物中,“10 DPA 对比 0 DPA”或“20 DPA 对比 10 DPA”阶段的差异表达基因数量最多,说明胚珠早期发育阶段存在显著转录变化(图3a)。有趣的是,在芝麻中,与脂质代谢相关的基因表达下调,而蛋白代谢相关基因上调;而在大豆中,脂质和蛋白代谢基因则同步上调(图3b),GO 富集分析结果一致(附加文件3:图S11)。在每种油料作物中,活跃的脂质代谢常常伴随着次级代谢基因的协同上调或下调(图3b)。这表明,尽管四种油料作物中次级代谢和蛋白代谢通路均对油脂积累产生重要影响,但脂质与次级代谢或蛋白代谢之间的竞争与协调机制因作物而异。
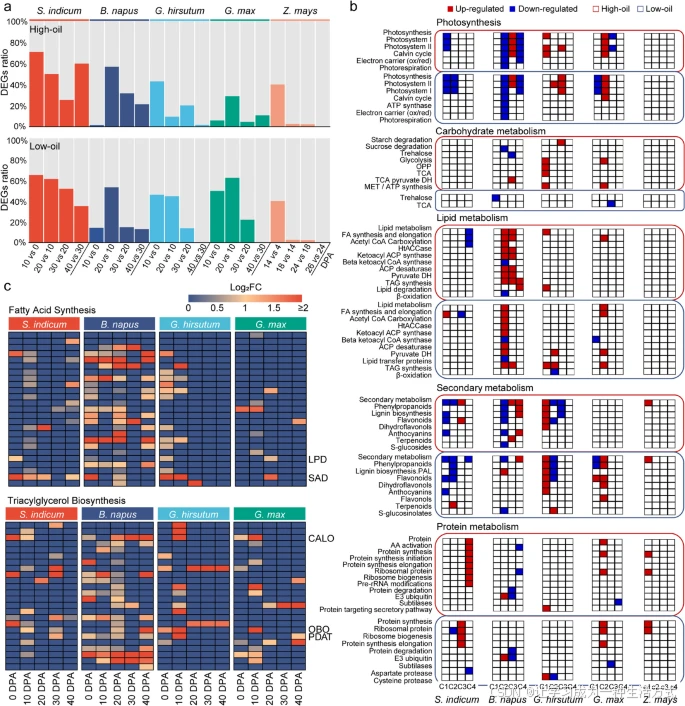
不同物种中基因的差异表达分析
a 展示了在各油料作物的高油与低油材料中,相邻发育阶段间差异表达基因(DEGs)占总表达基因的比例。 b 展示了高油与低油材料在相邻发育阶段间的差异表达基因功能富集结果。图中红框代表相关通路中基因上调,蓝框代表下调;红色边框代表高油材料,蓝色边框代表低油材料。C1、C2、C3、C4 分别表示油料作物中“10 vs 0 DPA”、“20 vs 10 DPA”、“30 vs 20 DPA”和“40 vs 30 DPA”;c1、c2、c3、c4 分别表示玉米中的“14 vs 4 DPA”、“18 vs 14 DPA”、“24 vs 18 DPA”和“26 vs 24 DPA”。 c 展示在每种油料作物中,“脂肪酸合成”和“三酰甘油生物合成”通路中高油材料相较于低油材料上调的基因的热图。图中 log₂FC 中位数表示该基因家族的表达变化倍数(对数值)。 缩略词:LPD,二氢脂酰胺脱氢酶;SAD,硬脂酰-ACP 脱饱和酶;CALO,caleosin;OBO,油体蛋白;PDAT,磷脂:二酰甘油酰基转移酶。
进一步地,我们总结了在“脂肪酸合成”和“三酰甘油生物合成”通路中,高油与低油材料之间及不同相邻发育阶段之间差异表达的基因,仅考虑上调的基因。在四种油料作物中,“脂肪酸合成”通路中编码二氢脂酰胺脱氢酶(LPD,丙酮酸脱氢酶复合物的 E3 成分)与硬脂酰-ACP 脱饱和酶的基因在高油材料中均表现出上调表达(图3c)。在“三酰甘油合成”通路中,PDAT、OBO 和 caleosin 基因在每种油料作物的高油材料中均显著上调(图3c)。
在相邻发育阶段的比较中,“脂肪酸合成”通路中的多个基因,如 KAS I、KAS II、KAS III、酮酰基-ACP 还原酶、β-丙酮酸脱氢酶、生物素羧基载体蛋白、生物素羧化酶和 α-丙酮酸脱氢酶,在高油与低油材料中均表现出差异表达(附加文件3:图S12)。这些基因主要参与从丙酮酸向丙二酰辅酶A的转化及脂肪酸合成循环。作为脂肪酸合成反应的辅因子及从叶绿体输出脂肪酸的激活因子,ACP 和长链酰基-CoA 合成酶编码基因在各物种的种子发育过程中均呈现保守的动态表达变化(附加文件3:图S12)。
此外,在“三酰甘油合成”通路中,PDAT、OBO、脂肪酸脱饱和酶2、FUSCA3、脱落酸不敏感蛋白 ABI3 和 ABI4、Caleosin 以及 HSI2/VAL1(糖诱导基因2/活胎素1/ABI3-Like1)等基因在各物种的高油与低油材料中均表现出活跃表达,主要作为转录因子或脂滴蛋白(附加文件3:图S13)。构建的种子油脂合成通路模型及基因表达谱显示,脂肪酸合成(FAS)基因在高油作物中表达较高,且常与糖酵解相关基因聚集表达;ABI3 与 ABI4 基因的表达在芝麻、棉花和大豆中与油体蛋白基因一致,但在油菜中不一致(附加文件3:图S14)。
糖酵解在种子油脂积累中的保守作用
为探索与 SOS(种子油合成)基因紧密关联的基因,我们对各物种进行了加权基因共表达网络分析(WGCNA)。芝麻、油菜、棉花、大豆和玉米的表达基因分别被划分为 11、10、9、7 和 7 个模块(附加文件1:表S4与附加文件3:图S15)。每种作物中我们鉴定出 2–3 个与种子油积累密切相关的模块(附加文件3:图S16与附加文件6:数据集S4,详见“方法”部分)。
为寻找跨物种与 SOS 相关的转录组特征,我们按基因与模块特征基因(eigengene)的 Pearson 相关系数对 SOS 模块中的所有基因进行排序,并开展基因集富集分析(GSEA)。结果显示,不同物种的 SOS 模块显著富集于碳水化合物代谢和氨基酸代谢通路(附加文件3:图S17)。有趣的是,“糖酵解/糖异生”在碳代谢中是所有物种中共同富集的通路(GSEA P < 0.05,FDR Q < 0.1)(图4a与附加文件7:数据集S5)。
在 SOS 模块中,“脂肪酸合成”通路的基因表达水平显著高于模块外该通路基因,表明其网络功能的关键性(图4b与附加文件3:图S18)。此外,在 SOS 模块中,油菜与棉花中“脂肪酸合成”通路基因的表达水平显著高于“糖酵解/糖异生”通路,而在芝麻和大豆中两者无显著差异;而在玉米中,恰好相反,“糖酵解/糖异生”基因表达显著高于“脂肪酸合成”(图4c与附加文件3:图S18)。
我们进一步计算了 SOS 模块中“糖酵解/糖异生”与“脂肪酸合成”基因之间的 Spearman 相关系数,结果发现:在芝麻和油菜中,这两类基因间几乎全部呈正相关,而在棉花、大豆和玉米中则多为负相关(图4d)。模块特征连接度(kME)作为模块中基因的中心性指标,其在芝麻、油菜和大豆中显示脂肪酸合成基因具有较高的中心性(图4e),提示其在网络中可能起着核心调控作用。
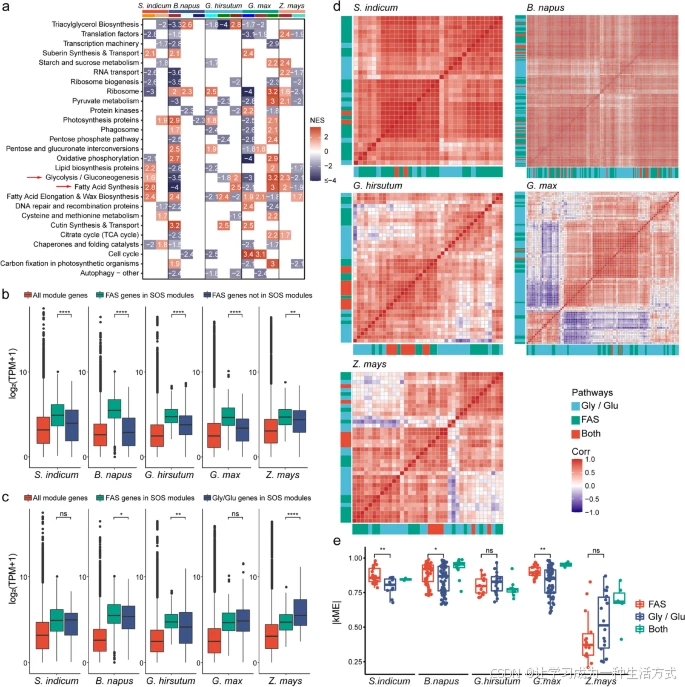
“脂肪酸合成”与“糖酵解/糖异生”在不同物种 SOS 模块中的基因关系
a 对各油料作物与玉米的 SOS 模块进行 GSEA 的 KEGG 富集分析(展示至少在三个物种中显著富集的通路)。NES 表示标准化富集得分,反映 KEGG 通路在按照模块成员 kME 排序的基因列表中的分布情况。
b 比较各油料作物高油材料中,处于 SOS 模块内与不在模块内的脂肪酸合成(FAS)基因的表达水平。ns 表示 P > 0.05,* 表示 P ≤ 0.05,* 表示 P ≤ 0.01,** 表示 P ≤ 0.001, 表示 P ≤ 0.0001,Wilcoxon 检验。
c 比较 SOS 模块中“脂肪酸合成(FAS)”基因与“糖酵解/糖异生(Gly/Glu)”基因在高油材料中的表达水平。显著性同上。
d 展示 FAS 基因、Gly/Glu 基因以及两者间交叉基因的 Spearman 相关系数热图。
e 比较不同作物中 FAS 基因、Gly/Glu 基因及交叉通路基因的模块成员归属度(|kME|)绝对值。
网络比对识别油脂积累保守调控机制
网络比对是一种整合多种复杂网络数据以识别跨物种功能保守网络的有效方法。为揭示进化上保守的调控油脂积累的网络,本研究整合了各油料作物的 SOS 模块作为输入,最终在 4 种作物中发现 692 个具有相似网络结构和序列同源性的对齐基因(附加文件8:数据集S6)。
这些对齐基因的蛋白结构域包括蛋白激酶、受体酪氨酸激酶、细胞色素 P450、MYB 等,四种作物中共有 465 个 Pfam 结构域(附加文件3:图S19)。图 5a 显示了包含 SOS 基因的一个保守模块(clique)。
为了验证保守基因对种子油积累的作用,我们测定了 5 个保守模块基因在拟南芥突变体中的种子油含量(SOC)。结果显示,pdat1 突变体的 SOC 比野生型增加了 8%,而其他突变体的 SOC 均下降了 15%–56%(图5b,附加文件9:数据集S7)。
进一步分析发现,各油料作物 SOS 网络中均包含的“糖酵解/糖异生”通路基因,如 SIN_1016466、BnaC01G0366100ZS、GH_D12G2756 和 Glyma.10G201100,均编码丙酮酸激酶(PK),该酶催化磷酸烯醇式丙酮酸向丙酮酸的转化。上述 4 个基因均具有较高的模块成员归属度(|kME| ≥ 0.75,附加文件10:数据集S8),显示其在模块中连接性强。
我们收集了与 PK 基因连接权重最高的前 30 个基因,发现大多数与 PK 的表达呈显著正相关(图5c)。这些基因在各油料作物的高油与低油材料间表达水平也存在差异(图5d)。
在玉米中,PK 也表现出较高的模块连接度(|kME| = 0.76,附加文件10:数据集S8),其权重前 30 的连接基因中包含多个与淀粉代谢相关的基因,如:STARCH SYNTHASE 2(淀粉合酶2)、STARCH BRANCHING ENZYME 2.2(淀粉支链酶2.2) 和 AGPase(ADP-葡萄糖焦磷酸化酶)(附加文件3:图S20)。这些基因在发育各阶段中表达普遍较高。
综上所述,我们通过跨物种的筛选,识别出调控碳源分配与油脂积累的保守路径调控因子,提供了关于种子油合成调控机制的关键遗传信息。
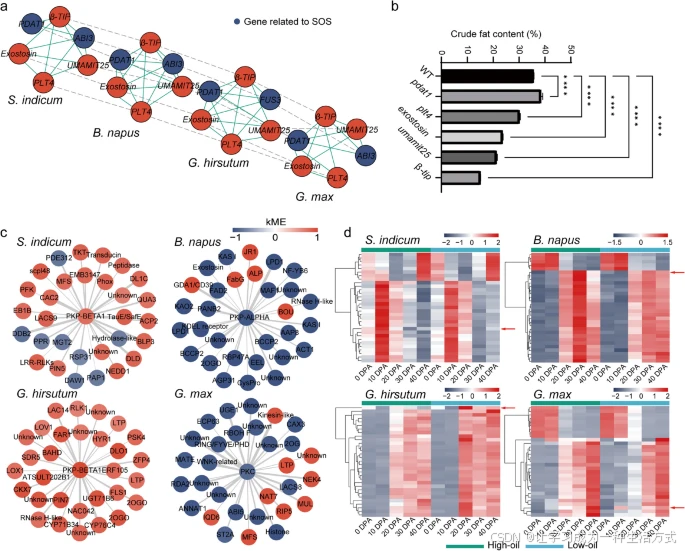
油料作物中 SOS 模块的网络比对
a 不同物种间 SOS 共表达网络的一个保守模块(clique)图。 b 拟南芥中保守模块中各单基因突变体的种子油含量(SOC)。P 值 ≤ 0.0001,单因素方差分析(ANOVA)检验。 c 与丙酮酸激酶(PK)连接权重最高的 30 个基因,节点颜色表示其在网络中的模块归属度(kME)值。 d 图 c 中各油料作物 PK 网络基因的表达热图,红色箭头指向 PK,表达值按行居中并标准化处理。
脂质代谢的竞争通路在不同物种中具有独立的调控机制
基于差异表达分析(DEA),我们发现次级代谢和蛋白质代谢在不同油料作物中对脂质代谢的影响程度不同。因此,我们进一步探讨并比较了四种油料作物中参与这两类代谢的基因与 SOS 基因之间的关系。
通过对 SOS 模块进行 GSEA 分析发现,“氨基酸代谢”在所有油料作物和玉米中均与网络中心基因(hub genes)显著相关,而“其他氨基酸代谢”则仅在大豆中显著相关(GSEA P < 0.05,FDR Q < 0.1,附加文件3:图 S17 与附加文件7:数据集 S5)。此外,不同类型的氨基酸代谢在各物种中也与 SOS 网络中心密切相关,例如:
-
芝麻中富集“半胱氨酸与蛋氨酸代谢”;
-
油菜中富集“苯丙氨酸、酪氨酸与色氨酸生物合成”以及“甘氨酸、丝氨酸与苏氨酸代谢”;
-
棉花中富集“缬氨酸、亮氨酸和异亮氨酸生物合成”;
-
大豆中富集“丙氨酸、天冬氨酸和谷氨酸代谢”(附加文件7:数据集 S5)。
其中,只有“半胱氨酸与蛋氨酸代谢”在四种油料作物中相对较为常见,但该通路在芝麻与棉花中与“三酰甘油生物合成”呈负相关,在棉花中还与“脂肪酸合成”负相关,而在大豆中却与“脂肪酸合成”呈正相关(图4a),表明其在不同作物中的调控方向不同。
次级代谢路径与油脂合成网络的互作机制
此外,在油菜和棉花中,“其他次级代谢产物的生物合成”通路显著富集于 SOS 网络中心(GSEA P < 0.05,FDR Q < 0.1,附加文件3:图 S17 与附加文件7:数据集 S5)。网络比对同时识别出在该通路中保守存在于四种油料作物 SOS 网络中的基因(SIN_1005794、BnaC03G0683600ZS、GH_D03G0499、Glyma.14G221200),它们编码肉桂醇脱氢酶(CAD),该酶催化苯丙烷合成途径中特异合成木质素单体的最后一步反应 [27]。
进一步分析发现,油菜与棉花中的 CAD 基因在网络中的连接度最高(|kME| ≥ 0.90,附加文件10:数据集 S8),而芝麻与大豆中则相对较低(图6a),说明在油菜和棉花中,CAD 可能通过调控脂质代谢与木质素生物合成之间的协调关系发挥作用。
我们还收集了与 CAD 连接权重最高的 30 个基因(图6a),发现 CAD 的表达水平在棉花和油菜中均显著高于其连接的其他基因(图6b)。尤其是在棉花中,GhCAD9 在油脂快速积累的关键期(20–30 DPA)持续高表达(图6b)。
进一步,我们获得了拟南芥的 CAD9 突变体(图6c),测定其种子的粗脂肪与木质素含量,结果表明:cad9 突变体中粗脂肪含量下降 52%,而木质素含量上升 36%(图6d 与附加文件9:数据集 S7),表明 CAD9 可调节种子中油脂与木质素的相对积累。
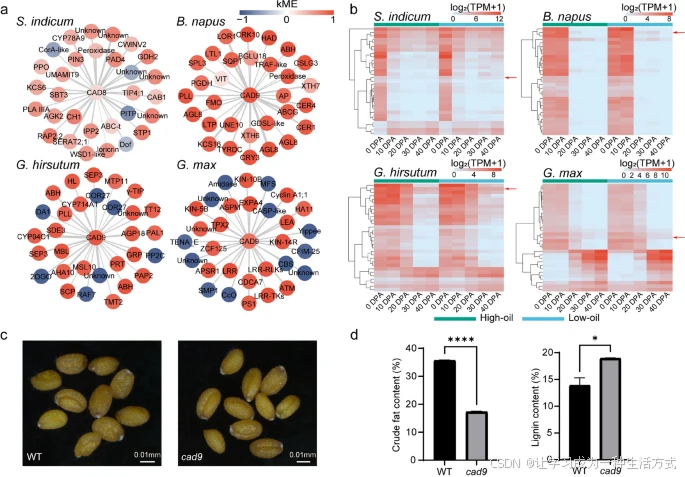
不同物种 CAD 网络的比较及拟南芥 cad9 突变体的种子油含量与木质素含量
a 与 CAD 连接权重最高的 30 个基因,节点颜色表示其在网络中的模块归属度(kME)值。 b 图 a 中各油料作物 CAD 网络基因的表达热图,红色箭头指向 CAD。表达值按行居中并标准化处理。 c 拟南芥野生型与 cad9 突变体的成熟干种子。 d 不同基因型种子的平均油含量与木质素含量。误差棒表示三组生物学重复计算的标准误差。*P ≤ 0.05,P ≤ 0.0001,单因素方差分析(ANOVA)检验。
讨论
粮食作物与油料作物种子储藏积累差异的决定因素
禾本科粮食作物和双子叶油料作物在种子储藏物质积累和结构上存在显著差异。玉米籽粒中约 70% 的重量是淀粉,主要集中在胚乳中,而其油脂主要存在于胚中 [28]。相比之下,油料作物的胚是主要结构,也是脂质合成和储存的关键部位。
本研究在比较不同作物种子储藏物质积累的基因组基础时发现,油料作物中酰基脂质代谢(ALM)相关基因占总基因的 2%,显著高于玉米和水稻等单子叶粮食作物(P < 0.01,双尾 Fisher 精确检验),提示基因组变异的选择在种子储藏积累差异中发挥了重要作用。
我们进一步在统一框架下,从基因共表达关系出发分析了油料与粮食作物储藏积累的进化过程。结果表明,“糖酵解/糖异生”通路中的基因在粮食与油料作物中均显著富集于 SOS 网络的中心,且与脂肪酸合成(FAS)基因紧密连接。这印证了糖酵解是油脂合成前体物质的主要来源 [29],也表明该机制在积累不同储藏物质的物种间具有保守性。
更具体地说,这两条通路中基因的正相关或负相关关系也会影响油脂积累。基因间的协同表达对于网络中层级调控至关重要,正向相关的基因若在功能上具有相同目标,其合作将通过中心节点放大,对表型造成显著影响。因此,通过敲除或沉默与目标路径呈负相关或属于竞争路径的中心基因,有望高效改良作物性状。
值得注意的是,只有在玉米中,“糖酵解/糖异生”基因的表达水平显著高于“脂肪酸合成”基因,这提示除了脂肪酸合成外,还有其他“糖酵解产物的获取者”参与了碳源的利用。但总体而言,植物种子储藏物质积累的调控网络仍不清晰。
已有研究表明,拟南芥的 TRANSPARENT TESTA GLABRA 1 在调控种子储藏积累中发挥重要作用 [30]。本研究通过网络比对发现,丙酮酸激酶(PK)在油料作物的 SOS 网络中表现出高度保守性和中心性。在玉米中,PK 也处于网络枢纽位置,且与其高权重连接的基因多数涉及淀粉合成 [31–33]。已有研究表明:拟南芥中降低质体 PK 活性可导致种子油含量下降 60% [34];水稻 OsPKpα1 突变体中质体 PK 活性下降,导致淀粉含量显著减少 [35];而 OsPK3 的功能缺失则影响蔗糖由源端向汇端转运,致使籽粒灌浆受阻 [36]。然而,PK 在不同油料作物中调控脂质积累的保守作用仍少有报道,其在碳源在淀粉与脂质间分配中的功能亟待深入研究。
控制竞争通路是提高种子油含量的潜在途径
非光合质体是淀粉、脂肪酸及氮同化合成氨基酸的重要合成场所 [37]。种子中蛋白与油脂含量呈负相关的现象已在大豆 [38–40]、芥菜 [41]、棉籽 [42]、油菜 [43]、芝麻 [44] 和藜麦 [45] 中观察到,而在玉米 [46] 和燕麦 [47] 中则为正相关。
长期以来,关于种子中油脂与蛋白含量之间的关键调控因子的研究相对有限。通过对各油料作物高油与低油材料在各发育阶段的差异表达基因功能富集分析,我们发现,在芝麻和油菜的高油材料中,与蛋白代谢相关的基因显著下调,可能有助于提高油含量。
此外,虽然“氨基酸代谢”通路与油料作物和玉米的 SOS 基因显著相关,说明脂质与蛋白质代谢间在多物种中广泛互作,但这种互作关系存在显著差异。例如,不同作物中关联最紧密的氨基酸代谢类型为:
-
芝麻:“半胱氨酸与蛋氨酸代谢”;
-
油菜:“苯丙氨酸、酪氨酸与色氨酸生物合成”及“甘氨酸、丝氨酸与苏氨酸代谢”;
-
棉花:“缬氨酸、亮氨酸和异亮氨酸合成”;
-
大豆:“丙氨酸、天冬氨酸和谷氨酸代谢”。
其中,仅“半胱氨酸与蛋氨酸代谢”在四种油料作物中相对共通,但其与“三酰甘油生物合成”或“脂肪酸合成”的相关性在作物间存在差异。这揭示了不同物种在种子蛋白与油脂的转化与积累方面的差异性,提示需要结合物种特性探索关键调控节点。
次级代谢生物合成对油脂积累的影响
研究还发现,油菜与棉花的 SOS 网络中,次级代谢物生物合成通路显著关联网络枢纽,说明次级代谢的竞争可能显著影响这两种作物的种子油积累。这两种作物中,与 SOS 网络枢纽高度相关的次级代谢通路包括苯丙烷生物合成与黄酮类生物合成。
其中,肉桂醇脱氢酶(CAD)被鉴定为调控油脂与木质素的潜在关键因子,其催化苯丙烷通路中合成木质素单体的最终步骤 [27]。已有研究也表明:油菜种皮中木质素含量与种子油含量显著负相关 [48, 49]。CAD 在各油料作物的 SOS 网络中具有功能保守性,而其调控机制在不同作物中的差异对油脂积累具有关键影响。
本研究通过对拟南芥 cad9 突变体与野生型的比较,验证了 CAD9 对种子油脂与木质素含量的调控作用。已有研究报道 cad4 与 cad5 对成株茎部木质素含量有影响,但 cad9 的诱导并未补偿 cad4 和 cad5 的缺失 [50],提示 CAD9 的功能分化及其调控作用仍需进一步深入研究。
调控保守的SOS基因是提高种子油含量的一个重要途径
迄今为止,通过阐明和基因改造油脂生物合成途径,成功地提高了种子油含量。然而,在模型生物中通过基因操作实验实现的油脂积累增量,与物种间在种子油含量(SOC)上的差异相比,显得微不足道。显著提高不同油料作物的SOC已成为亟待解决的关键问题。
不同油料作物高油和低油材料之间一致的差异表达基因(DEGs)具有作为关键调节因子来提高种子油含量的潜力。在每种油料作物中,与低油材料相比,编码LPD的基因在高油材料中均呈上调状态。已经证实,质体内的PDHC在种子脂质合成过程中形成乙酰辅酶A的主导作用 [51]。LPD是一个大的黄素蛋白氧化还原酶家族的成员,完成催化周期是通过重新氧化脂酰胺辅因子 [52]。然而,关于LPD如何调节植物种子中的脂质积累仍知之甚少。此外,我们发现,编码硬脂酰-ACP脱饱和酶的基因在不同油料作物的高油和低油材料中表现出差异表达,这些作物的脂肪酸组成存在差异,表明它在物种间具有功能保守性,并且具有工程化特定种子油组成的潜力。本研究还发现,高油和低油材料之间PDAT的差异表达在物种间的一致性,提示其在脂质积累中的关键作用,这在个别物种中已有证明 [10, 53]。我们还识别了一个保守的基因模块,包括PDAT、ABI3及其他基因的共表达关系。ABI3也发现与编码油体蛋白的基因表达一致,这些蛋白围绕存储油脂的离散小器官。因此,尽管PDAT的研究从未停止,但关于PDAT与ABI3在SOS网络中的关键交互作用仍需进一步了解。对拟南芥pdat1突变体与野生型之间种子油含量的比较显示,PDAT显著增加了油脂积累。先前的研究表明,PDAT1和DGAT1具有重叠功能:dgat1-1 pdat1-2双突变导致无性花粉,且缺乏可见的油体,在dgat1-1背景下RNAi沉默PDAT1或在pdat1-1背景下沉默DGAT1会导致油含量下降70%至80% [10]。这解释了为什么pdat1突变体中SOC的增加可能是由于PDAT和DGAT之间的相互补偿机制,且这一机制可能由DGAT主导。
总结了在发育阶段中表现出一致差异表达变化的基因,大多数基因在脂质积累中的重要性已在一些物种的先前研究中得到验证。ACETYL-CoA CARBOXYLASE和PDHC的调控模型已经得到了很好的建立 [54,55,56,57]。在本研究中,编码它们亚基的基因在种子发育过程中表现出积极的表达。参与脂肪酸合成循环的基因在不同作物的脂肪酸合成关键时期被上调,表明脂肪酸碳链延长在物种间高度保守,凸显了它们的重要性。FUSCA3、ABI3和ABI4是三种在所有物种中在种子发育过程中积极表达的转录因子,它们在脂质积累中的积极作用已在多个物种中得到证明 [58,59,60,61,62,63,64]。此外,HSI2/VAL1对种子成熟的负调节作用已被提出 [65,66],但其在种子油积累中的作用尚未得到强有力的确认,本研究推测HSI2/VAL1在不同油料作物的三酰甘油合成中的作用是保守的。
更重要的是,基因共表达网络作为单个基因的功能放大器和传递器,不能脱离群体独立工作。正如本研究所发现的,所有物种中,网络模块中FAS基因的表达高于非网络基因的表达。在这里,我们识别了在物种进化历史中维持共表达关系的基因模块。因此,研究结果超越了个别物种的观察,为揭示哪些基因和机制可能是种子油积累的基础提供了新的见解。例如,基因协作在“脂肪酸合成”和“糖酵解/糖异生”通路中的积极作用是油脂积累的关键,这不仅体现在这两条通路中基因表达的相关性,还体现在功能富集模块的数量上。
结论
总体而言,我们生成了四种具有显著不同SOC的物种的种子发育RNA-seq数据集,提供了对基因表达和协作的种子油积累特征的全面比较分析。我们探讨了种子中主要储存化合物积累过程的关联性,发现了谷物和油料作物之间的保守性和差异性。共表达网络对齐提供了一种发现不同物种间关键保守基因和模块的方法。我们的研究超越了个别物种研究的观察,为从多物种的角度提供哪些基因和机制可能是种子油积累的基础提供了新的见解。我们希望我们的研究能够为油料作物的高效改良提供有价值的参考。
方法
植物材料和生长条件
我们实验室的棉花材料是G. hirsutum acc. TM-1(低SOC)和G. hirsutum cv. CRI12(高SOC)。芝麻材料J9014(低SOC)和Yu4(高SOC)来自中国农业科学院油料作物研究所。大豆材料KF-1(低SOC)和NN1138(高SOC)来自南京农业大学国家大豆改良中心。不同SOC的油菜材料来自南京农业大学作物遗传与种质创新国家重点实验室。所有材料均在田间种植。
对照野生型为拟南芥(A. thaliana)Columbia-0种源。T-DNA插入株系pdat1(SALK_032261)、plt4(SALK_097021)、exostosin(SALK_018694)、umamit25(SALK_140423)、β-tip(SALK_125353)和cad9(SALK_081375)来自AraShare(AraShare科学)。通过三引物(LBb1.3 + LP + RP)法(T-DNA Primer Design)鉴定纯合突变体。我们在播种前用75%酒精对所有种子进行表面消毒,然后将其播种在0.8%(w/v)琼脂固体培养基上(半强度Murashige和Skoog培养基,1%蔗糖,pH 5.8–6)。种子在4°C黑暗条件下打破休眠2天后,允许其发芽,并在22°C的培养室中生长7天,然后将幼苗移植到土壤中进一步生长。
脂质和蛋白质分析
从冷冻的成熟种子中提取总脂肪,并按Kang和Rawsthorne的描述进行定量 [67]。各脂肪酸(FA)的含量通过GC/MS测定 [68]。总脂肪酸的量通过将所有主要成分的量相加来计算。为测定蛋白质含量,将10毫克种子与1毫升50 mM 4-(2-羟乙基)-1-哌嗪乙烷磺酸钠/NaOH缓冲液(pH 7.4)混合,在全玻璃均质器中均质。
RNA测序数据分析
当这些材料开始开花时,在0至40天后(DPA)期间,每10天随机标记和采样一次。每个时间点取三个生物学重复样本。来自不同材料的五个阶段(0、10、20、30和40 DPA)的总RNA使用Illumina HiSeq 2500系统,采用双端100-bp模式进行测序。大豆、棉花、油菜、芝麻和玉米的参考基因组已发布并更新(附录1:表S5)[21,22,23,24,25]。清洁的RNA-seq读取数据分别使用HISAT 2.0 [69]与参考基因组比对。TPM(每百万映射读取的外显子模型转录本数)和映射的读取计数分别通过StringTie(版本1.3.5)[70]和featureCounts(版本1.6.4)[71]计算。使用Fisher精确检验 [72] 比较二元变量(所有基因和参与ALM和SOS的基因是否表达)。
通过主成分分析(PCA)(FactoMineR版本2.4)[73]评估样本重复之间的测序库质量,并通过R(版本3.6.1)中Spearman方法进行相关性检验。全球和物种特异性的PCA分析使用prcomp R包(R: The R Project for Statistical Computing)对来自读取计数的前500个变异基因表达矩阵进行分析,应用DESeq2(版本1.12.4)[74]中实施的方差稳定转化。相关性低于0.85的重复样本所在的数据集被移除。
ALM相关基因的鉴定与比较
从Acyl Lipids: pathways获取了总共768个与拟南芥ALM相关的基因(排除了5个伪基因)。拟南芥的蛋白质序列用于鉴定过程的测试。所鉴定的基因必须满足以下三个条件:(i)目标序列与查询序列的相似度超过33.3%,通过Diamond(v0.9.29.130)[75] BLASTP通过;(ii)使用HMMER3.1 [76]预测6种作物的蛋白质序列的蛋白质结构域,显著性阈值设置为“-E 1e-5”和“–domE 2e-20”。选择具有相同结构域的基因对;(iii)使用OrthoFinder(版本2.4.0)[77]的默认参数(−I = 1.5)为每个作物和拟南芥分配唯一的同源组。使用“双侧”Fisher精确检验 [72] 比较二元变量(是否与某种物种和水稻相关的ALM基因)。
基因家族扩展和收缩分析
为了了解这四种油料作物中脂质代谢相关基因家族的进化变化,针对四种作物、玉米、水稻和拟南芥进行了基因家族扩展和收缩分析。首先,从OrthoFinder(版本2.4.0)[77]的先前鉴定结果中收集所有七个物种的同源基因和旁系基因,并用于构建STAG物种进化树。收集水稻和拟南芥的预测分歧年份作为标准,从Timetree(TimeTree :: The Timescale of Life)获取数据,用r8s(v. 1.81)构建过度度量树,调整系统发育树的时间尺度。然后,使用CAFÉ(版本4.2.1)[78]确定显著进化的同源组,排除具有显著拷贝数变异的基因家族(仅存在于一个物种且N > 100)。为了确定同源组扩展和收缩的显著性,基于蒙特卡罗重采样程序计算每个同源组的P值,并将显著扩展和收缩的阈值设置为P值 < 0.05(全家族P值)。通过Viterbi方法获得分支特异性P值,随机生成似然分布。此方法计算系统发育树所有分支之间父母和子代家族大小变化的确切P值。较低的分支特异性P值(< 0.01)表示该分支迅速进化。最后,根据Pfam数据库(Pfam is now hosted by InterPro)使用HMMER [76]对同源组进行注释。
差异基因表达分析和功能富集分析
差异基因表达分析在R(版本3.6.1)中使用DESeq2(版本1.12.4)[74]包进行。以featureCounts(版本1.6.4)[71]提供的计数表作为输入,只保留在至少三个文库中达到最低10次读取的基因。P值的选择通过BH方法 [79]在α = 0.05下控制假发现率(Q值)。通过Mercator(Mercator4 - plant protein functional annotation)构建五个物种的统一标准全基因组功能注释后,使用Mapman [80]对每个物种的DEG进行功能富集分析。模型使用ORA-FISHER,Benjamini Yekutilie用于多重测试校正。DEG的GO富集分析通过R(版本3.6.1)中的ClusterProfiler(v3.14.0)[81]包进行。
加权基因共表达网络分析
加权基因共表达网络分析(WGCNA)包 [82] 在R(版本3.6.1)中用于构建单物种的加权基因共表达网络。使用来自DESeq2(版本1.12.4)[74]的高油和低油材料合并基因表达矩阵的rlog标准化输出作为WGCNA的输入。通过使用每个作物的适当软阈值构建所有基因对在样本间Pearson相关矩阵的邻接矩阵。使用动态树切割算法 [83] 定义网络模块,模块的最小大小为100。属于不同模块的基因被分配不同的颜色,未分配颜色的基因为灰色。模块特征基因表达量作为标准化模块表达谱的第一个主成分进行计算。通过Pearson相关计算基因的模块成员关系kME,表示模块内的连接性 [84]。
种子油合成模块的鉴定与基因集富集分析
SOS模块的鉴定通过两种方法进行 [85]。涉及脂肪酸合成、脂肪酸延伸、去饱和和从质体出口以及TAG生物合成的基因被选为SOS基因。首先,使用“greater” Fisher精确检验 [72] 将每种油料作物和玉米中的SOS基因富集到网络模块中。因此,选择包含更多SOS基因的模块(P值 < 0.05)。第二种方法使用基因集富集分析(GSEA)[86] 选择SOS基因在模块中发挥重要枢纽作用的模块(GSEA P值 < 0.05,FDR Q值 < 0.1)。与SOS相关的基因集和按kME排名的基因被输入到R包ClusterProfiler(v3.14.0)[81]中的GSEA功能,以比较不同物种中SOS共表达网络的组织情况。通过使用来自京都基因与基因组百科全书(KEGG)数据库的基因集,对每个物种中的SOS网络枢纽进行功能富集分析,显著性阈值为P < 0.05,FDR Q < 0.1。
网络对齐分析
通过NetCoffee(版本1.0)[87] 对物种间的多个基因共表达网络进行全局对齐分析。NetCoffee通过模拟退火最大化目标函数来搜索全局对齐。NetCoffee所需的网络文件来自SOS模块的合并共表达网络,并通过权重 > 0.1的阈值进行过滤。每对物种的序列相似性文件通过Diamond(版本0.9.29)[75] BLASTP序列比对结果获得。表示拓扑结构对齐分数贡献度的参数设置为0.5(−alpha = 0.5)。使用Cytoscape v3.9.0 [88] 进行网络可视化。Cytoscape v3.9.0的Gasoline [89] 插件用于对NetCoffee中的保守网络进行局部对齐。还需要BLAST E值作为相似性信息。所有参数设置为默认值(密度阈值为0.8;sigma代表初始对齐蛋白质的最小节点度,设为3)。
数据与材料的可获取性
所有棉花材料可从通讯作者处获得。本文生成的四种油料作物的原始RNA测序数据可以通过国家生物技术信息中心(NCBI)SRA下载,项目代码为PRJNA862748(https://www.ncbi.nlm.nih.gov/bioproject/PRJNA862748)[90]。本文使用的玉米原始RNA测序数据可在NCBI SRA中通过访问号SRP037559(https://www.ncbi.nlm.nih.gov/sra/?term=SRP037559)[91] 获得。
