CellCharter | 入门了解
Background — cellcharter
内容来源于以上链接,下面大多是原教程的翻译,稍加自己的思考。
这是第一期,后面会有第二期:详细教程
一、背景
空间聚类(Spatial Clustering)(或空间域识别,spatial domain identification)能确定以这些群体的特定混合为特征的细胞生态位(cellular niches)。它根据细胞的内在特征(也可以叫做分子特征,例如protein或 mRNA expression)以及组织中邻近细胞(neighboring cells)的特征,将细胞分配到不同的cluster中。
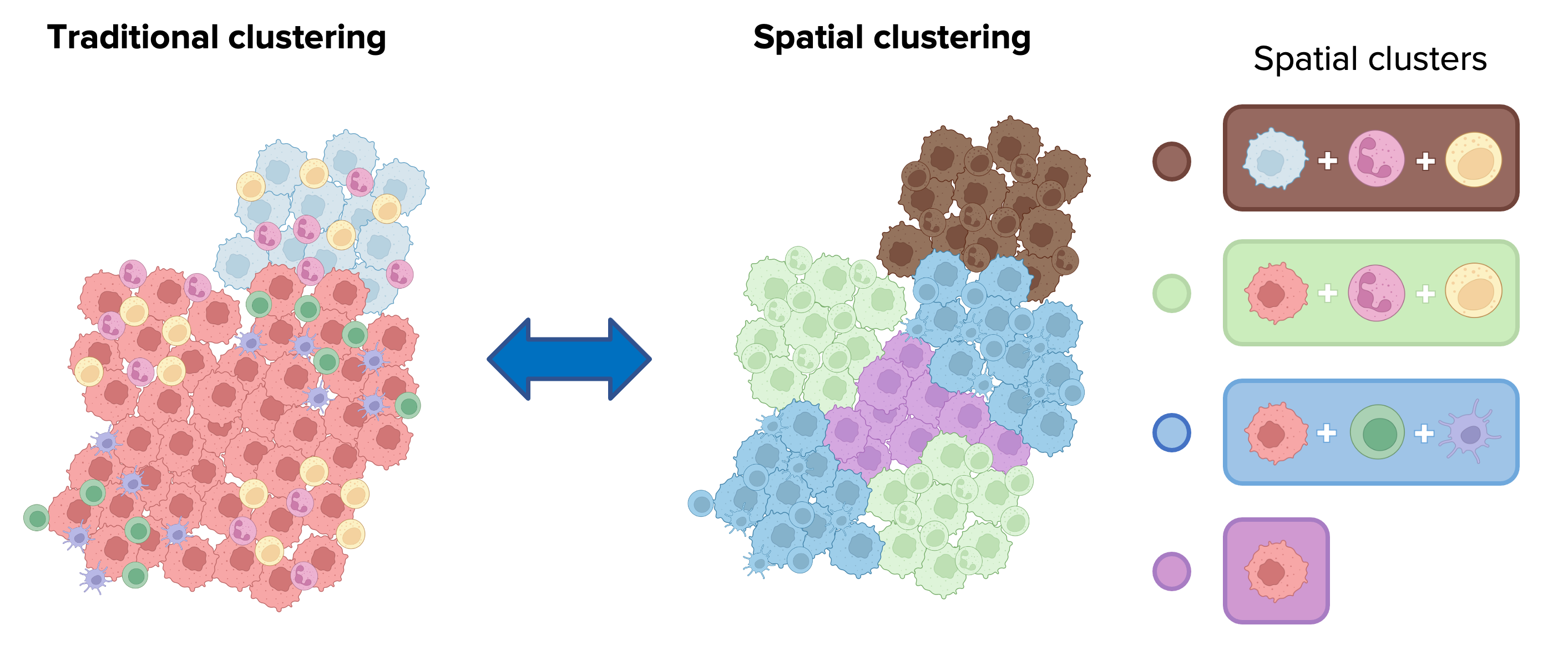 CellCharter 能够自动identify spatial domains,并提供一套用于cluster characterization和comparision的方法。
CellCharter 能够自动identify spatial domains,并提供一套用于cluster characterization和comparision的方法。
二、功能
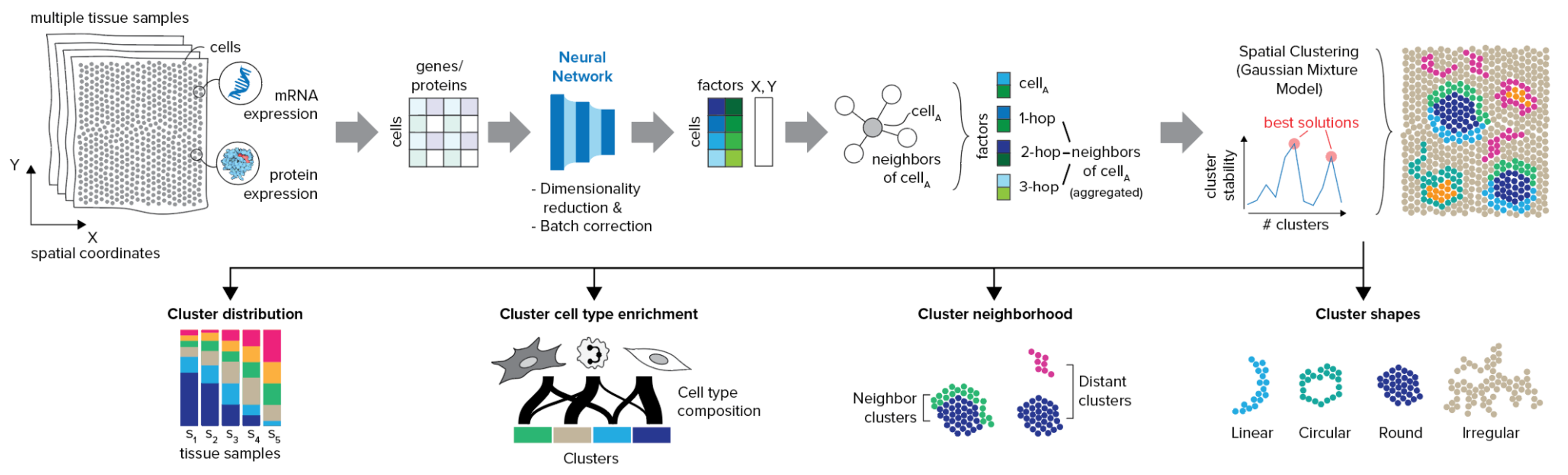
1、identify多个样本的niches:通过结合 scVI 和 scArches 的优势,CellCharter 能够同时为多个样本识别niche,即便存在batch effects也不例外。
2、Scalability:CellCharter 能够处理包含millions of cells和thousands of features的大型数据集。在 GPU 上运行它的可能性使其速度更快。
3、Flexibility:CellCharter 可用于不同类型的空间组学数据,如空间转录组学、蛋白质组学、表观基因组学和多组学数据。唯一的区别在于用于dimensionality reduction 和 batch effect removal的方法。
1)Spatial transcriptomics:CellCharter 已在采用零膨胀负二项分布的 scVI 上进行了测试。
2)Spatial proteomics:CellCharter 已在 scArches 的一个版本上进行了测试,该版本经过修改,使用均方误差损失代替默认的负二项损失。
3)Spatial epigenomics:CellCharter 已在采用泊松分布的 scVI 上进行了测试。
4)Spatial multiomics:可以使用诸如 MultiVI,或者使用来自不同模型结果的拼接。
4、最佳域数量候选:CellCharter 提供了一种寻找多个最佳域数量候选的方法(best candidates),该方法基于特定数量的domain在多次运行中的稳定性。
5、域特征分析:CellCharter 提供了一套用于 characterize 和 compare spatial domains 的工具,例如domain proportion、cell type enrichment、(differential)neighborhood enrichment、domain shape characterization等。
从 CellCharter 0.3.0 版本开始,我们将高斯混合模型(GMM)的实现从PyCave(已不再维护)迁移到了TorchGMM,这是 CSO 团队维护的 PyCave 的一个分支。这一变化使我们能够拥有一个更稳定且持续维护的 GMM 实现,该实现与最新版本的 PyTorch 兼容。
三、Getting Started
三个链接:
项目总位置:https://cellcharter.readthedocs.io/
-
API documentation.
-
Tutorials
四、Installation
1、创建 conda 或 pyenv 环境
2、安装 Python >= 3.8 和 PyTorch >= 1.12.0。如果计划使用 GPU,请确保先点这个链接Get Started并安装正确版本的 PyTorch。
3、根据你计划分析的数据类型,安装用于dimensionality reduction和batch effect removal的库:
1)scVI 适用于空间转录组学和 / 或表观基因组学数据,例如 10x Visium 和 Xenium、Nanostring CosMx、Vizgen MERSCOPE、Stereo-seq、DBiT-seq、MERFISH 和 seqFISH 数据。
2)scArches 的 TRVAE 模型的修改版本适用于空间蛋白质组学数据,例如 Akoya CODEX、Lunaphore COMET、CyCIF、IMC 和 MIBI-TOF 数据。
4、使用 pip 安装 CellCharter:
pip install cellcharter我们建议使用mamba来安装依赖项。安装依赖项的最新版本(特别是scvi-tools和spatialdata)可能会导致依赖冲突。不过,这应该不是问题,因为 CellCharter 不会使用任何不匹配的功能。
在此我们提供一个旨在分析空间转录组学数据(因此需要安装scvi-tools)的安装示例。该示例基于带有 NVIDIA A100 GPU 的 Linux CentOS 7 系统。
conda create -n cellcharter-env -c conda-forge python mamba
conda activate cellcharter-env
mamba install pytorch torchvision torchaudio pytorch-cuda=12.4 -c pytorch -c nvidia
pip install scvi-tools
pip install cellcharter注意:不同的系统可能需要不同的命令来安装 PyTorch 和 JAX。有关更多详细信息,请参考它们各自的文档。
五、Graph/Tools/Plotting
这是一个生物信息学Python包 cellcharter 的API文档页面,专门展示图形分析(Graph)相关的功能模块。
当前页面功能
显示的是Graph(图形分析)模块,包含以下主要函数:
空间邻域分析:
gr.aggregate_neighbors- 聚合每个neighborhood layers的features并将其连接gr.connected_components- 计算spatial graph的connected components
差异分析:
gr.diff_nhood_enrichment- 不同条件间的差异邻域富集gr.enrichment- 计算标签在组中的富集程度gr.nhood_enrichment- 邻域富集分析
图形处理:
gr.remove_long_links- 移除距离过大的细胞间连接gr.remove_intra_cluster_links- 移除同一cluster内的细胞连接
Tools模块 (工具模块)
1. 聚类分析工具
tl.cluster- 基于CellCharter邻域聚合特征进行cell或spot聚类tl.clusterAutoK- 自动识别最佳聚类数量的候选值tl.TRVAE- 适用于图像蛋白质组学数据的scArches trVAE模型
2. 拓扑形态分析
这是cellcharter的核心创新功能,用于量化细胞群体的空间形态特征:
tl.boundaries- 计算细胞集合的拓扑边界tl.curl- 计算边界的卷曲度(curl score)tl.elongation- 计算边界的伸长度tl.linearity- 计算边界的线性度tl.purity- 计算边界的纯度
Plotting模块 (可视化模块)
1. 聚类质量评估
pl.autok_stability- 绘制聚类稳定性图表pl.boundaries- 可视化cluster边界
2. 富集分析可视化
pl.enrichment- 绘制标签在组中富集程度的点图pl.nhood_enrichment- 邻域富集分析的可视化(改进版squidpy功能)pl.diff_nhood_enrichment- 不同条件间邻域富集差异的可视化
3. 比例和形态分析
pl.proportion- 绘制不同键值的比例图pl.shape_metrics- 两种条件间形状指标的箱线图对比