【医学影像 AI】YoloCurvSeg:仅需标注一个带噪骨架即可实现血管状曲线结构分割
【医学影像 AI】YoloCurvSeg:仅需标注一个带噪骨架即可实现血管状曲线结构分割
- 0. 论文简介
- 0.1 基本信息
- 0.2 摘要
- 1. 引言
- 2. 相关工作
- 2.1 曲线结构分割
- 2.2 弱监督分割
- 2.3 医学图像合成
- 3. 方法
- 3.1 曲线结构生成
- 3.2 用于背景提取的修复技术
- 3.2.1 网络架构
- 3.2.2 目标函数
- 3.2.3 训练
- 3.3 基于块状对比学习的合成
- 4. 实验
- 4.1 数据集与预处理
- 4.2. 实现细节
- 4.3 合成性能
- 4.4 与最先进方法的比较
- 4.4.1 与WSL方法的比较
- 4.4.2 与NLL方法的比较
- 4.5 鲁棒性分析与消融研究
- 4.6 与最先进全监督方法的比较及讨论
- 5. 结论
- 参考文献
0. 论文简介
0.1 基本信息
2023 年 南方科技大学 Li Lin,Xiaoying Tang(唐晓颖) 等在 Medical Image Analysis 发表论文 “YoloCurvSeg:仅需标注一个带噪骨架即可实现血管状曲线结构分割(YoloCurvSeg: You Only Label One Noisy Skeleton for Vessel-style Curvilinear Structure Segmentation)”。
本文亮点:
- 提出了一种用于单次涂鸦标注监督的曲线结构分割新颖框架。
- 开创性利用噪声稀疏标注数据的弱监督方法。
- 通过四个步骤创新地将弱监督学习问题转化为全监督问题。
- 具备噪声鲁棒性、样本不敏感性,并可轻松扩展至各类曲线结构。
- 在OCTA500、CORN、DRIVE、CHASEDB1和DCA1数据集上进行评估。
- YoloCurvSeg以显著优势超越最先进弱监督及噪声标签学习方法。
- 仅需一个带噪声骨架标注即可达到全监督性能97%的效果。
- 本研究将为弱监督学习与曲线数据集构建的后续工作提供重要启发。
论文下载: arxiv.org, sciencedirect
项目下载: GitHub
引用格式:
Li Lin, Linkai Peng, Huaqing He, et al., YoloCurvSeg: You only label one noisy skeleton for vessel-style curvilinear structure segmentation, Medical Image Analysis, 2023, Vol 90, 102937,
https://doi.org/10.1016/j.media.2023.102937.
(https://www.sciencedirect.com/science/article/pii/S1361841523001974)
接受日期:2023年1月

0.2 摘要
弱监督学习(WSL)通过采用稀疏粒度(即点级、框级、涂鸦级)标注来缓解数据标注成本与模型性能之间的冲突,并显示出良好的性能,尤其在图像分割领域。然而,由于监督信息有限,特别是在仅有少量标注样本可用时,这仍然是一个非常具有挑战性的任务。此外,现有弱监督分割方法几乎都是针对星凸结构设计的,与血管、神经等曲线结构存在显著差异。
本文提出了一种名为YoloCurvSeg的新型稀疏标注曲线结构分割框架。该框架的核心组成部分是图像合成技术:具体而言,背景生成器通过修复扩张骨架来生成与真实分布高度吻合的图像背景;随后通过基于空间殖民算法前景生成器生成的随机模拟曲线,并经过多层块状对比学习合成器进行融合。这种方法仅需消耗一个或少量带噪声的骨架标注,即可获得同时包含图像和曲线分割标签的合成数据集。最后,使用生成的数据集和可能的未标注数据集训练分割器。
所提出的YoloCurvSeg在四个公开数据集(OCTA500、CORN、DRIVE和CHASEDB1)上进行评估,结果表明其性能大幅优于最先进的弱监督分割方法。在仅使用一个带噪声骨架标注(分别相当于全标注量的0.14%、0.03%、1.40%和0.65%)的情况下,YoloCurvSeg在各数据集上均达到全监督性能97%以上的效果。代码与数据集将在https://github.com/llmir/YoloCurvSeg发布。
关键词:稀疏标注;单样本学习;曲线结构分割;弱监督学习;医学图像合成
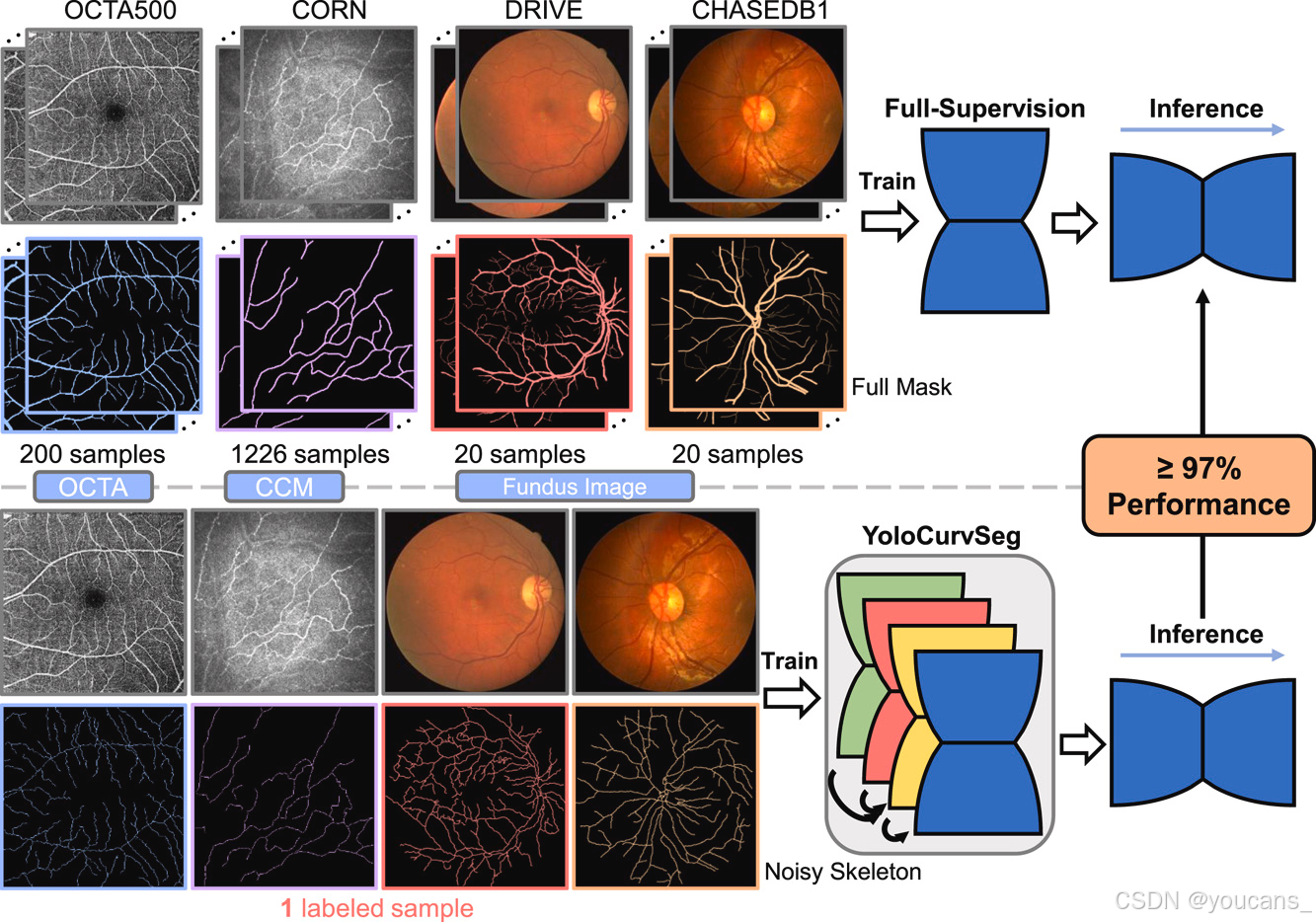
图1. YoloCurvSeg仅需利用一条带有噪声的骨架标注,在四个代表性数据集上均达到了超过97%的全监督性能,这意味着医生可以大幅节省标注时间,同时仍能获得令人满意的分割结果。
1. 引言
曲线结构是细长的、弯曲的、多尺度的结构,通常呈现树状形态,常见于自然图像(如裂缝和航空道路图)和生物医学图像(如血管、神经和细胞膜)。对这些曲线结构进行自动精确分割在计算机视觉和生物医学图像分析中都具有重要意义。例如,道路测绘是自动驾驶和城市规划的先决条件。在生物医学领域,研究(Pritchard等,2014;Lin等,2021c;Kawasaki等,2009;Lin等,2020)表明特定曲线解剖结构(如视网膜血管和角膜神经纤维)的形态和拓扑结构与多种疾病的存在或严重程度高度相关,例如高血压、小动脉硬化、角膜炎、年龄相关性黄斑变性、糖尿病视网膜病变等。视网膜血管可在视网膜眼底图像和光学相干断层扫描血管成像(OCTA)图像中观察到,而角膜神经纤维可在共聚焦角膜显微镜(CCM)图像中识别。许多眼科疾病的早期征兆通过微血管和毛细血管异常反映(Allon等,2021;Lin等,2021b)。总体而言,对各种曲线结构进行准确分割对于计算机辅助诊断、定量分析和多种疾病的早期筛查至关重要,特别是在眼科领域。
近年来,受益于深度学习(DL)的发展,许多基于DL的曲线结构分割算法被提出,并显示出相比传统(例如基于匹配滤波和形态学处理的方法(Nguyen等,2013;Singh和Srivastava,2016))的压倒性性能。大多数现有工作致力于设计复杂的网络架构(Peng等,2021;Mou等,2021;He等,2022)和部署策略,通过使用生成对抗网络(GAN)(Lin等,2021c;Son等,2019)或拓扑保持损失函数(Cheng等,2021b;Shit等,2021)来保持曲线结构的拓扑结构。这些方法通常是全监督的,需要大规模精细标注的数据集。然而,收集和标注具有完整标注的大规模数据集非常昂贵且耗时,特别是对于医学图像,因为其标注需要专业知识和临床经验。此外,鉴于曲线结构细长、多尺度、形状复杂且细节精细,标注曲线结构更具挑战性。
最近,许多努力致力于降低DL模型训练的标注成本。例如,半监督学习(SSL)通过结合有限数量的标注数据与大量未标注数据来训练模型(Xu等,2022;Hou等,2022;Mittal等,2019)。虽然有效,但大多数最先进(SOTA)的SSL方法仍然需要约5%–30%的精确标注数据才能达到约85%–95%的全监督性能,这在标注曲线结构时仍然不够经济高效且耗时。弱监督学习(WSL)试图从另一个角度通过执行稀疏粒度(即点、涂鸦、边界框级别)的监督来缓解标注问题,并取得了令人瞩目的性能(Liang等,2022;Lin等,2016;Tang等,2018a;Tang等,2018b;Kervadec等,2019)。与点或边界框相比,涂鸦是一种相对更灵活和通用的稀疏标注形式,可用于标注复杂结构(Luo等,2022)。现有的涂鸦监督分割方法主要分为两类。第一类研究利用结构或体积先验将涂鸦扩展为更准确的伪提案;例如,将具有相似灰度强度或位置的像素分组到同一类别中(Liang等,2022;Lin等,2016;Ji等,2019)。然而,扩展过程可能会引入噪声提案,这可能导致错误积累并降低分割模型的性能。一些工作(Huo等,2021)也指出了这些方法的固有弱点,即模型保留自己的预测从而抵抗更新。第二类利用额外的未配对但完整标注的掩码学习对抗形状先验。这种方法在某种程度上与节省标注成本的动机相矛盾,特别是对于复杂的曲线结构(Larrazabal等,2020;Valvano等,2021;Zhang等,2020c)。此外,大多数WSL方法仍然需要对整个数据集(或大部分)进行稀疏标注,并且它们主要是在相对简单的结构(例如心脏结构或腹部器官)上设计和验证的,其假设和先验可能不适用于复杂结构(例如曲线结构)。
为了解决上述挑战,我们在此提出了一种新颖的用于血管式曲线结构的WSL分割框架,即You Only Label One Noisy Skeleton for Curvilinear Structure Segmentation(YoloCurvSeg)。对于曲线结构,标签噪声/错误是不可避免的,一个好的分割方法应具有噪声容忍性。因此,YoloCurvSeg不是仅使用标注像素进行监督,而是通过图像合成巧妙地将弱监督问题转化为全监督或半监督问题。它使用训练好的修复网络作为背景生成器,该生成器接受一个(或多个,取决于可用性)带噪声的骨架(如图1所示)并将其扩张以作为修复掩码,从而获得与真实分布紧密匹配的背景。然后,提取的背景通过基于空间殖民算法的前景生成器生成的随机模拟曲线进行增强和组合,通过多层块状对比学习合成器获得合成数据集。最后,分割器使用合成数据集和未标注数据集(如果可用)执行从粗到细的两阶段分割。我们的主要贡献总结如下:
- 我们提出了一种新颖的弱监督框架,用于单次骨架/涂鸦监督的曲线结构分割,即YoloCurvSeg。据我们所知,YoloCurvSeg是利用带噪声和稀疏标注数据进行曲线结构分割的开创性弱监督分割方法。
- YoloCurvSeg通过四个步骤新颖地将WSL问题转化为全监督问题:曲线生成、图像修复、图像翻译和从粗到细的分割。所提出的框架具有噪声鲁棒性、样本不敏感性,并且易于扩展到各种曲线结构。
- 我们在四个具有挑战性的曲线结构分割数据集上评估YoloCurvSeg,即OCTA500(Li等,2020)、CORN(Zhao等,2020)、DRIVE(Staal等,2004)和CHASEDB1(Fraz等,2012)。实验结果表明,YoloCurvSeg大幅优于SOTA WSL和噪声标签学习方法。同时,我们证明仅使用一个带噪声的骨架标签(约完整标注的0.1%或1%)即可实现97%的全监督性能,这也将启发后续关于WSL和曲线数据集构建的工作。
2. 相关工作
相关工作主要涉及曲线结构分割、弱监督分割和医学图像合成,下文将逐一介绍。
2.1 曲线结构分割
现有自动曲线结构分割算法大致可分为两类。
第一类是传统无监督方法,主要包括数学形态学方法和各种滤波方法(Mou等,2021)。例如,Zana和Klein(2001)使用形态学滤波和交叉曲率分析的混合框架分割血管状图案。Passat等(2006)提出了一种初步方法,通过将高级解剖知识融入分割过程来加强脑血管分割。滤波方法包括基于Hessian矩阵的滤波器(Frangi等,1998)、匹配滤波器(Singh和Srivastava,2016;Hoover等,2000)、多方向滤波器(Soares等,2006)、对称滤波器(Zhao等,2017)等。
另一类是有监督方法,其中使用具有真实标签的数据基于预定义或模型提取的特征训练分割器。传统基于机器学习的方法致力于使用手工特征进行像素级分类(Zhang等,2017;Holbura等,2012)。近年来,基于DL的方法在各种分割任务中取得显著进展。例如,Ronneberger等(2015)提出的 U-Net已被广泛用于众多医学图像分割任务。现有曲线结构分割工作通过引入多尺度(He等,2022;Wu等,2018)、多任务(Lin等,2021b;Peng等,2021;Hao等,2022)或各种注意力机制(Mou等,2021;Yu等,2022)来聚焦精心设计的网络架构,并通过引入GAN或形态/拓扑保持损失函数(Cheng等,2021a;Shit等,2021)来良好保持形态和拓扑特性。尽管如此,数据可用性和标注质量仍是这些方法的主要限制因素。
2.2 弱监督分割
弱监督分割旨在通过使用粗粒度标注数据训练分割模型来降低标注成本(Liang等,2022)。在各种稀疏标注格式中,涂鸦被认为是最灵活和通用的形式,甚至可用于标注非常复杂的结构(Luo等,2022;Valvano等,2021)。现有涂鸦监督分割方法主要分为两大类。
第一类利用结构或体积先验,通过将相同类别分配给具有相似强度或邻近位置的像素来扩展涂鸦标注(Lin等,2023;Liang等,2022;Lin等,2016;Ji等,2019)。此类方法的主要局限性在于其严重依赖伪提案且通常包含多个阶段,这既耗时又容易产生错误,并可能在模型训练过程中传播。
第二类利用额外的未配对但完整标注的掩码学习对抗形状先验。这种方法在某种程度上与节省标注成本的动机相矛盾,特别是对于复杂曲线结构(Larrazabal等,2020;Valvano等,2021;Zhang等,2020b)。此外,这些方法仍需要对整个数据集或大部分进行稀疏标注,且主要是在相对简单结构(如心脏结构或腹部器官)上设计和验证的,其假设和先验可能不适用于复杂结构(如曲线结构)。
本文中,我们利用的带噪声骨架在两个方面不同于涂鸦:(1)骨架需要更多标注工作,因为所有分支都应被覆盖;(2)带噪声骨架更可能包含错误或噪声,这在快速标注细长结构时不可避免。我们通过图像合成流程将稀疏且带噪声的骨架标注转换为准确标注,因此仅需一个带噪声骨架标签。这显著降低了标注成本。
2.3 医学图像合成
GAN(Goodfellow等,2020)已成为医学图像合成的主流技术,常见应用包括模态内增强(Zhou等,2020)、跨域图像到图像转换(Peng等,2022)、质量增强(Cheng等,2021b)、缺失模态生成(Huang等,2022a,b)等。下面我们简要回顾与本文相关的视网膜图像合成研究。Costa等(2017a)采用使用配对眼底图像和血管掩码训练的U-Net。该方法使用条件GAN(即Pix2pix(Isola等,2017))学习从血管掩码到相应眼底图像的映射。为简化框架,他们提出对抗自编码器(AAE)用于视网膜血管合成和GAN用于生成视网膜图像(Costa等,2017b)。类似地,Guibas等(2017)提出两阶段方法,包括使用DCGAN从噪声生成血管系统和使用cGAN(Pix2pix)合成相应眼底图像。需要注意的是,cGAN需要配对图像和血管掩码进行训练,这在一定程度上是严格条件。这些方法需要额外一组血管标注来训练AAE或DCGAN,且有时可能生成具有不现实形态的血管。生成图像也缺乏多样性。Zhao等(2018)开发了Tub-sGAN,将风格迁移融入GAN框架以生成更多样化输出。另一项研究中,SkrGAN(Zhang等,2019)被提出引入草图先验相关约束来指导图像生成过程。然而,所使用的草图是通过Sobel边缘算子提取的,不能用作分割掩码。
在本文中,我们采用基于多层块状对比学习的前景-背景融合GAN主要基于以下考虑。根据先前研究,训练GAN学习从曲线结构掩码到相应图像的直接映射很困难,尤其在少样本条件下(Lin等,2021a)。因此,我们向GAN提供提取的真实背景,实现隐式跳跃连接,使GAN能更专注于映射前景区域。这种设计不仅提升性能,还加速收敛。多层块状对比学习使得所提供的掩码与生成图像的前景区域在空间上对齐(通过非配对训练),这进一步有利于后续分割器。
3. 方法
YoloCurvSeg包含四个主要组件:
(1)曲线生成器(Curve Generator),生成能良好适应目标图像模态的二进制曲线掩码;
(2)修复器(Inpainter),从标注样本中提取背景;
(3)合成器(Synthesizer),根据生成的曲线掩码和图像背景合成图像;
(4)两阶段分割器(Segmenter),使用合成数据集和未标注数据集进行训练。整体框架如 图2 所示。

图2。
(A) 本文提出的YoloCurvSeg方法概览,包含四个主要组件:基于空间殖民算法的曲线生成器、
背景修复器、基于多层块状对比学习的前景-背景融合合成器,以及两阶段由粗到细的分割器。
(B) 曲线生成器的细节及四个所用数据集的曲线生成过程示意图。
3.1 曲线结构生成
空间殖民算法是计算机图形学中的一种程序化建模算法,可模拟分支网络或树状结构的生长(Runions等,2005,2007),包括血管系统、叶脉、根系等。
YoloCurvSeg采用该算法模拟曲线结构的迭代生长过程,其两个基本要素为吸引子(attractors)和节点(nodes)。核心步骤如图2 左下方面板所述(蓝色点表示吸引子,黑色点表示节点):
- a) 随机或按预定义模式放置一组吸引子,然后将节点与附近吸引子关联(若节点与吸引子间距离在吸引距离Da内);
- b) 对每个节点,计算其受所有影响吸引子作用的平均方向;
- c) 通过将平均方向归一化为单位向量并按预设分段长度Ls缩放,计算新节点位置;
- d) 将节点置于计算位置,并检查是否有节点进入吸引子的清除区域;
- e) 若节点处于清除距离Dk内,则移除该吸引子;
- f) 重复步骤b)-e直至达到最大节点数。
通过观察单张或少量可获取图像中前景/曲线的模式(包括曲线起始点、边界和曲率程度等),可相对直观地设置相应超参数,如根节点坐标Cr(例如眼底血管起始点位于视盘区域)、边界和障碍物。对于Da、Dk和Ls,常用值5、30和5可根据需要重新调整。吸引子采用网格布置策略,通过设置水平和垂直方向的网格数量来控制吸引子数量。为简化,两个方向使用相同网格数Ag。每个吸引子可在一定范围Aj内抖动以引入随机性。位于边界外或障碍物内的吸引子将被移除。表1 总结了生成四类曲线所用的参数和后处理操作,代表性示例如 图2下方面板所示。
表1. 不同种类曲线生成的参数设置。其中Da、Dk、Ls和Cr分别表示吸引距离、消除距离、线段长度和根节点坐标。r和l分别表示半径和长度。Ag和Aj是两个超参数,分别用于控制吸引子网格数量和随机抖动的范围。符号 ⊕\oplus⊕ 和 ⊙\odot⊙分别表示并集运算和逐元素相乘运算。OCTA500数据集对应两行参数,因其采用两个独立组件分别生成水平大血管和向心性小血管结构。
需注意,本文采用的设置和后处理操作仅代表经验性选择而非最优方案,用户可根据实际观察和经验进一步调整。本配置中,通过与视野(FOV)区域相乘确保曲线与对应图像背景对齐,且不超出FOV范围。采用随机裁剪(Random Crop)和随机翻转(Random Flip)增强曲线多样性,同时使用腐蚀(Erode)和膨胀(Dilate)微调曲线粗细。除曲线形态外,还需模拟各分支粗细:
Rn=R1n+R2nR^n = R_1^n + R_2^n Rn=R1n+R2n
其中:R、R1和R2分别表示父分支及其两个子分支的半径。根据Murray定律(Painter等,2006),n 设为3。该计算从分支末端(半径设为1)向树基递归进行。可通过link(https://jasonwebb.github.io/2d-space-colonization-experiments/)查看直观演示。通过预定义参数设置随机网格吸引子和根节点,我们为每个目标数据集构建了同类但形状多样的曲线库,随后用于训练合成器和分割器。
3.2 用于背景提取的修复技术
修复(Inpainting)是指重建图像中缺失或被遮蔽区域的任务。类似于去除图像中的水印或无关行人,本文采用修复模型在假设扩张后的带噪声骨架能完全覆盖前景的前提下,从目标图像中移除前景(如血管和神经纤维)。在修复任务中,常见的关注点包括网络捕捉局部与全局上下文的能力,以及向不同(尤其是更高)分辨率泛化的能力。
3.2.1 网络架构
受Suvorov等人(2022)的启发,我们采用基于近期提出的快速傅里叶卷积(FFCs)(Chi等人,2020)的修复网络,该网络具有图像级感受野、强大的泛化能力和相对较少的参数。给定掩码图像I ⊙ (1 − m)(其中I和m分别表示原始图像和修复区域的二进制掩码),前向修复网络fθ(·)以四通道输入I′ = concat(I ⊙ (1 − m),m)为基础,旨在输出修复后的图像ˆI = fθ(I′)。FFC基于通道级快速傅里叶变换(FFT)构建,其感受野可覆盖整个图像。它将通道分为两个并行分支:局部分支采用传统卷积,全局分支采用实FFT捕捉全局上下文(如图3所示)。实FFT仅适用于实值信号,而逆实FFT确保输出为实值。与FFT相比,实FFT仅使用一半频谱。在FFC中,首先对输入张量应用实FFT,并通过拼接实部与虚部执行ComplexToReal操作。随后在频域中进行卷积运算,通过RealToComplex操作执行逆实FFT将特征从频域变换到空间域。最后融合局部与全局分支。对于修复器的上采样/下采样以及对抗训练中判别器的架构,我们分别遵循He等人(2016)和Suvorov等人(2022)采用的ResNet设置。训练基于[图像,随机合成掩码]对进行。我们采用Suvorov等人(2022)中的掩码生成策略,包含多个任意纵横比的矩形和宽多边形链。
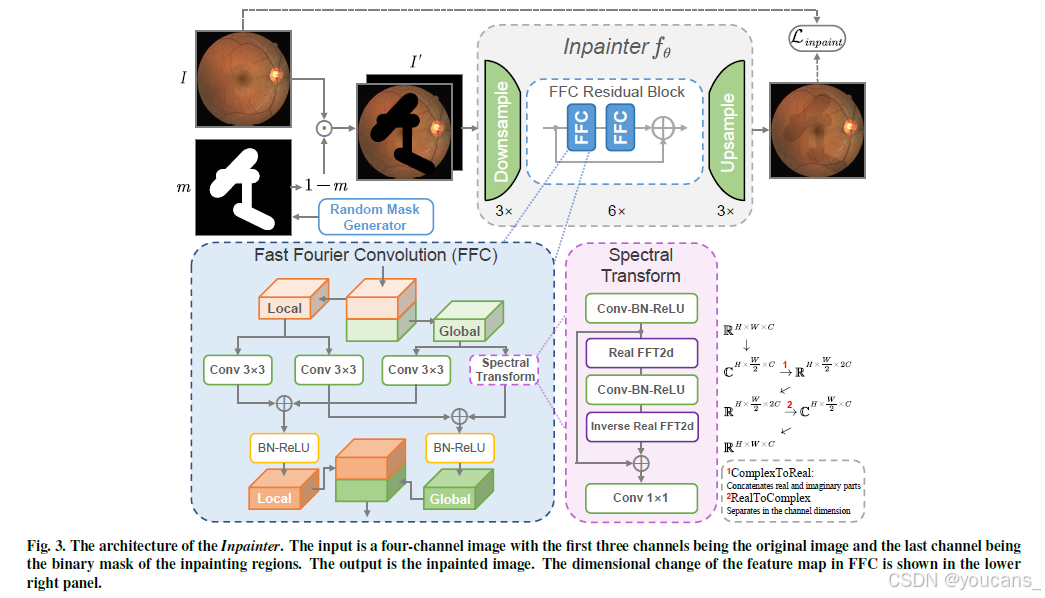
图3. Inpainter模型架构。输入为四通道图像,其中前三个通道是原始图像,最后一个通道为待修复区域的二值掩码。输出为修复后的图像。右下角面板展示了FFC中特征图的维度变化。
3.2.2 目标函数
与可能导致模糊预测的朴素监督损失相比,感知损失(Johnson等人,2016)通过预训练网络ϕ(·)评估修复图像与原始图像特征图之间的距离。它不需要精确重建,并允许重建图像存在一定变异。鉴于修复任务侧重于理解全局结构,我们通过具有扩张卷积的预训练 ResNet50 网络 ϕHRF(⋅)ϕ_{HRF}(·)ϕHRF(⋅) 引入大感受野感知损失LHRP。

其中M是一个顺序两阶段均值算子,即先计算层内均值再计算层间均值。此外,采用对抗损失Ladv以增强修复图像的真实性。具体而言,我们使用PatchGAN(Isola等,2017)判别器Dξ(·),将与掩码重叠的图像块标记为假样本,其余标记为真样本。非饱和对抗损失定义为:

其中ˆI = fθ(I′)是修复网络的输出,sgvar表示对变量var的停止梯度操作。为进一步稳定训练过程,我们使用梯度惩罚LGP = EI∥∇Dξ(I)∥22(Ross与Doshi-Velez,2018)以及基于判别器特征的感知损失LDP(Wang等,2018)。修复器的最终目标函数为:

其中λadv、λDP和λGP是平衡不同损失贡献的超参数。LHRP负责监督信号和全局结构一致性,而Ladv和LDP负责局部细节和真实感。
3.2.3 训练
鉴于修复器(Inpainter)的训练不需要标注且其通过学习上下文理解来恢复缺失区域的通用能力,我们使用Places-Challenge数据集(Zhou等,2017)的预训练参数初始化模型,并在各训练集内可获取的图像上进行微调。修复器的验证集包含可获取的训练集图像和验证集图像(每张图像配有10个预定义掩码,这些掩码采用与Suvorov等(2022)相同的生成策略)。训练批量大小为8,采用Adam优化器,学习率为10−3,共训练50个周期(epochs)。数据增强包括随机翻转、旋转和颜色抖动。对于每个训练图像,我们首先应用上述数据增强策略离线生成20张增强图像,随后在训练期间采用相同策略进行在线增强。根据经验设置λadv = 3、λDP = 10和λGP = 10−4。训练完成后,修复器以扩张后的带噪声标注作为掩码,用于从骨架标注样本中移除前景。随后通过对提取的背景进行随机水平/垂直翻转及旋转(0◦至90◦范围)增强,为每个数据集构建背景库。
3.3 基于块状对比学习的合成
4. 实验
本节将在四个具有代表性的曲线结构分割数据集上广泛评估我们提出的YoloCurvSeg框架的有效性。
4.1 数据集与预处理
我们在四个眼科数据集上全面评估YoloCurvSeg:OCTA500、CORN、DRIVE和CHASEDB1。
OCTA500用于视网膜微血管分割,仅使用包含300个样本、具有6×6 mm²视野(FOV)和400×400分辨率的子集。我们仅利用由内界膜层与外丛状层之间最大投影生成的en-face图像。
CORN包含1578张用于神经纤维分割的CCM图像,同时提供分别包含340张低质量和288张高质量图像的两个子集。所有CCM图像的分辨率为384×384,FOV为400×400 μm²。我们不采用数据集的原始划分方式,而是使用1532张图像(移除与测试集重叠的样本,验证集划分比例为0.2)进行训练和验证,并在其子集提供的60个相对准确标注的样本上进行测试。
DRIVE和CHASEDB1用于视网膜血管分割,分辨率分别为565×584和999×960。这两个眼底数据集通过提供的FOV掩码进行裁剪,并分别调整尺寸至576×576和960×960。对于DRIVE,我们采用原始划分的20个训练样本和20个测试样本。对于CHASEDB1,我们遵循Lin等(2021c)和He等(2022)的划分方式,前20张图像作为训练集,剩余8张用于测试。对于OCTA500,我们分别使用200、10和90个样本作为训练集、验证集和测试集。图像首先进行归一化处理,在线数据增强包括随机旋转、翻转和B´ezier曲线变换(Zhou等,2019)。
4.2. 实现细节
我们通过PyTorch在配备8块RTX 3090Ti GPU的工作站上实现YoloCurvSeg及其他对比方法。在合成器(Synthesizer)中,用于计算Lc的选定层索引包括{0, 4, 8, 12, 16}。对于训练分割器S_coarse和S_fine,采用power=0.9的多项式策略(Mishra和Sarawadekar,2019)在线调整学习率。其他超参数、训练细节和模型架构已在前述章节提供。
值得注意的是,OCTA500、DRIVE和CHASEDB1提供了人工描绘的血管分割标签。为这三个数据集生成带噪声骨架标注,我们使用scikit-image(Van der Walt等,2014)中的骨架化操作获取原始真实掩码的骨架,随后采用弹性变换模拟快速人工标注可能引入的抖动噪声。对于CORN数据集,仅提供带噪声骨架标签,因此直接用于所有实验。
针对该数据集,我们将每个骨架扩张至3像素宽度作为全监督学习设置中的完整掩码,此操作同样应用于测试集标注。对于其他对比WSL方法使用的稀疏标签,背景骨架也通过骨架化生成。
YoloCurvSeg中的合成过程可在线或离线进行。为更好复现性和公平比较,实验中采用离线版本——首先生成合成数据集,随后训练分割器。通过随机组合预生成曲线库和增强背景库中的样本,若所有训练样本均被标注,我们分别为OCTA500、CORN、DRIVE和CHASEDB1生成1276、5005、1240和1604个合成样本;若仅标注一个样本,则分别生成100、100、60和80个合成样本。所有对比方法和YoloCurvSeg中的分割模型均训练30000次迭代。
4.3 合成性能
在与最先进(SOTA)WSL方法比较前,我们首先定性和定量评估YoloCurvSeg的合成性能。
- 图4可视化展示了带噪声骨架标签、用于修复的扩张掩码、提取的背景、生成曲线及合成图像的代表性示例。最后一列可见生成曲线与合成图像高度匹配。
- 图5 比较合成数据集与真实数据的强度分布,显示合成图像在背景和前景方面均与真实图像具有高度强度相似性。
- 通过图6的t-SNE(Van der Maaten和Hinton,2008)可视化可见,合成数据集与真实数据总体吻合且充分混合。多数情况下,合成数据分布甚至更均匀广泛,起到类似数据增强的效果。
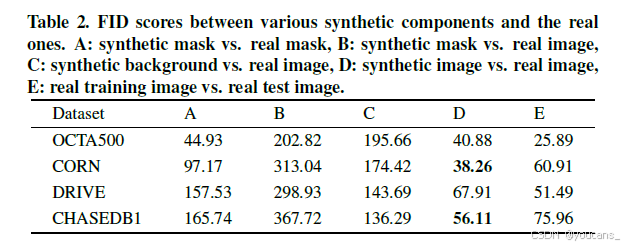
表2列出合成组件与对应真实数据之间的FID值以衡量学习两者分布间映射的难度:
- A列显示合成与真实曲线结构间的形态学差距;
- B列和C列表明从前景掩码直接转换到图像分布难度极大,而移除前景的背景图像与原始分布距离较小。
由于YoloCurvSeg引入背景信息(隐式实现跳跃连接),降低了图像转换难度,因此即使在少样本设置下也能合成接近真实的图像。我们的方法在四个数据集上均取得具有竞争力的FID分数,其中两个甚至低于真实训练集与测试集之间的FID值(见表2E列)。合成曲线与合成图像中感兴趣区域的成功对齐,以及合成图像与真实图像的高度相似性,共同构成YoloCurvSeg在后续实验中取得强大分割性能的关键因素。
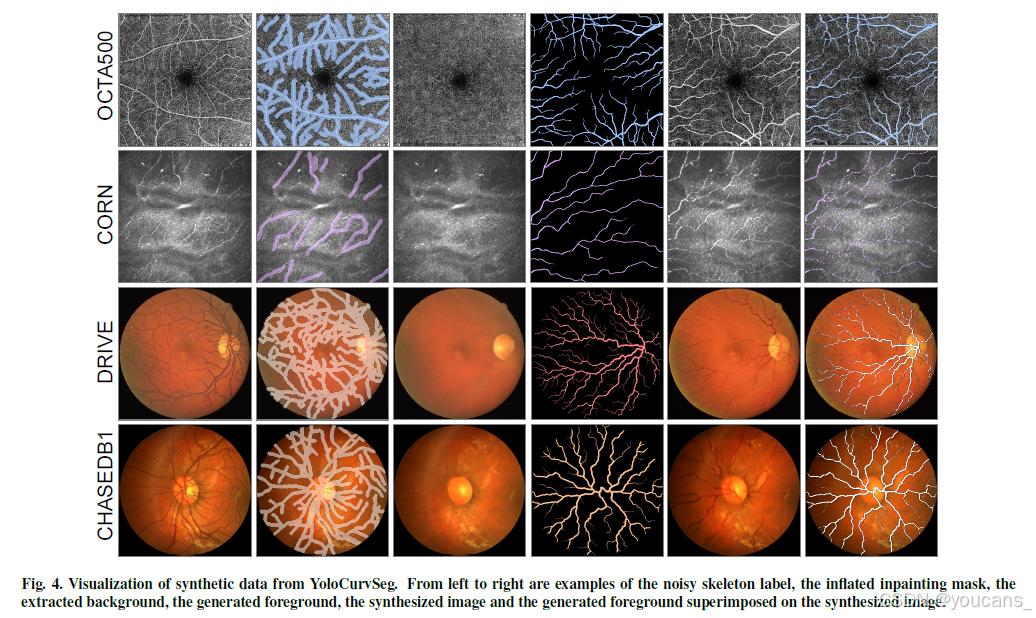
图4. YoloCurvSeg生成的合成数据可视化效果。从左至右依次为:带噪声的骨架标签、膨胀后的修复掩模、提取的背景、生成的前景、合成图像,以及叠加在合成图像上的生成前景。
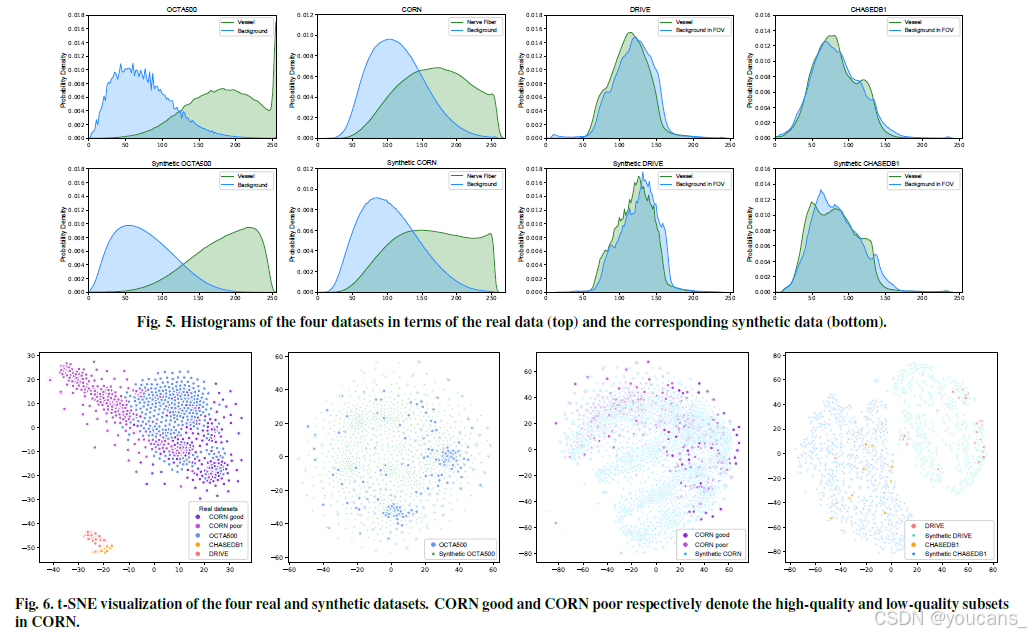
图5. 四种数据集的特征分布直方图对比(上方为真实数据,下方为对应合成数据)。
图6. 四组真实与合成数据集的t-SNE可视化结果。其中CORN_good与CORN_poor分别代表CORN数据集中的高质量与低质量子集。
表2. 各类合成组件与真实数据间的FID评分对比
A:合成掩模 vs. 真实掩模;
B:合成掩模 vs. 真实图像;
C:合成背景 vs. 真实图像;
D:合成图像 vs. 真实图像;
E:真实训练图像 vs. 真实测试图像
4.4 与最先进方法的比较
由于带噪声骨架可被视为具有一定噪声程度的稀疏标注或简单视为带噪声标签,我们将YoloCurvSeg与两类方法进行比较:(1)弱监督学习(WSL)方法;(2)噪声标签学习(NLL)方法。采用Dice相似系数(DSC[%])和平均对称表面距离(ASSD[像素])作为评估指标,同时使用灵敏度(SE)和特异度(SP)进行更全面的方法差异评估。所有作为基准的WSL方法、NLL方法以及全监督(FS)方法均采用与S_coarse和S_fine相同的分割网络架构(即原始U-Net)以确保公平比较。选择原始U-Net架构还因其在医学图像分割任务中具有多功能性和广泛应用(Ronneberger等,2015;Isensee等,2021;Antonelli等,2022)。
4.4.1 与WSL方法的比较
我们将YoloCurvSeg与11种使用相同生成骨架集的涂鸦监督分割方法进行比较:pCE(部分交叉熵损失,基线);随机游走伪标签(RW);不确定性感知自集成和变换一致性模型(USTM);Scribble2Label(S2L);Mumford-Shah损失(MLoss);熵最小化(EM);密集CRF损失;门控CRF损失;主动轮廓损失(AC);动态混合伪标签监督的双分支网络(DBDM);以及树能量损失。结果如表3 和表4 所示。为公平比较,YoloCurvSeg不经过精细阶段,表示为Ours(coarse)(即S_coarse的性能)。每个表格的上半部分表明所有训练数据均被稀疏标注,且所有训练集图像均用于比较方法中的分割器训练(对于我们的方法,这些图像用于训练修复器、合成器和分割器)。下半部分中,"One"表示仅有一个样本被标注,所有其他数据未标注且未被使用。请注意在单样本设置中,修复器和合成器仅使用稀疏标注样本的单一图像进行训练,此设置在下述所有实验流程中保持一致。此外,所有分割网络均随机初始化,未使用任何预训练参数。
表3. 现有弱监督学习方法在OCTA500与CORN数据集上的对比结果。最佳结果以粗体标出,次佳结果采用下划线标注,FS代表全监督学习(fully-supervised learning)。
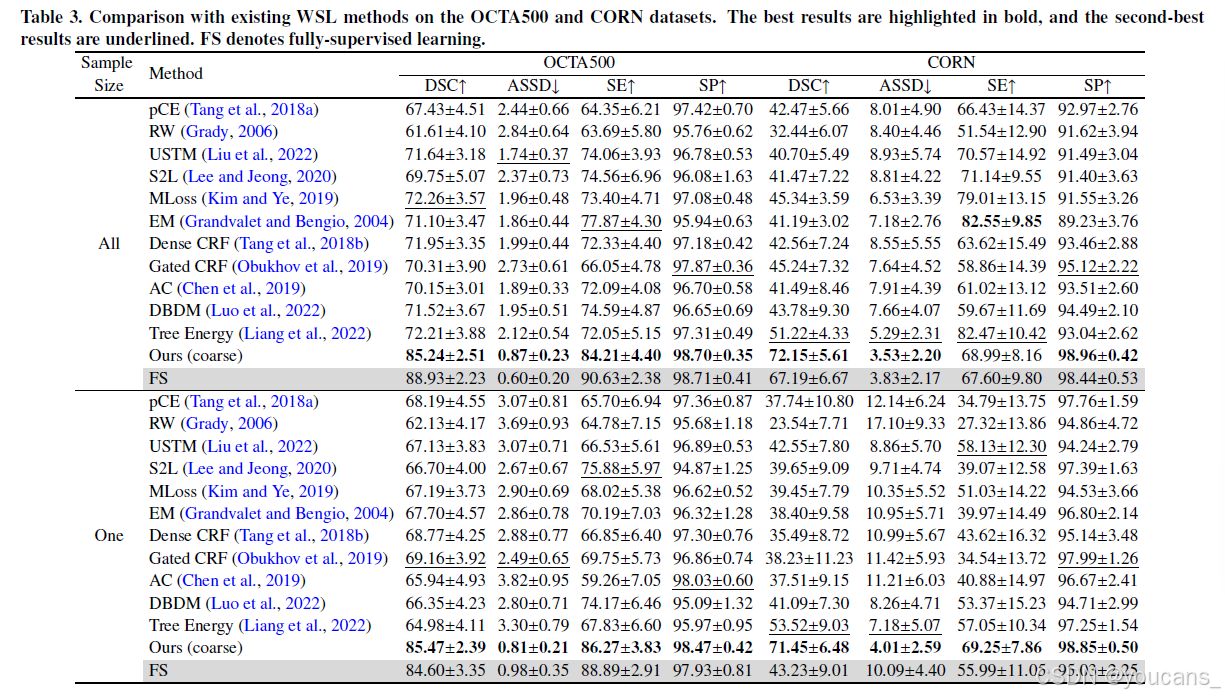
表4. 现有弱监督学习方法在DRIVE和CHASEDB1数据集上的性能对比(最优结果以粗体标示,次优结果以下划线标注)。FS表示全监督学习(fully-supervised learning)。
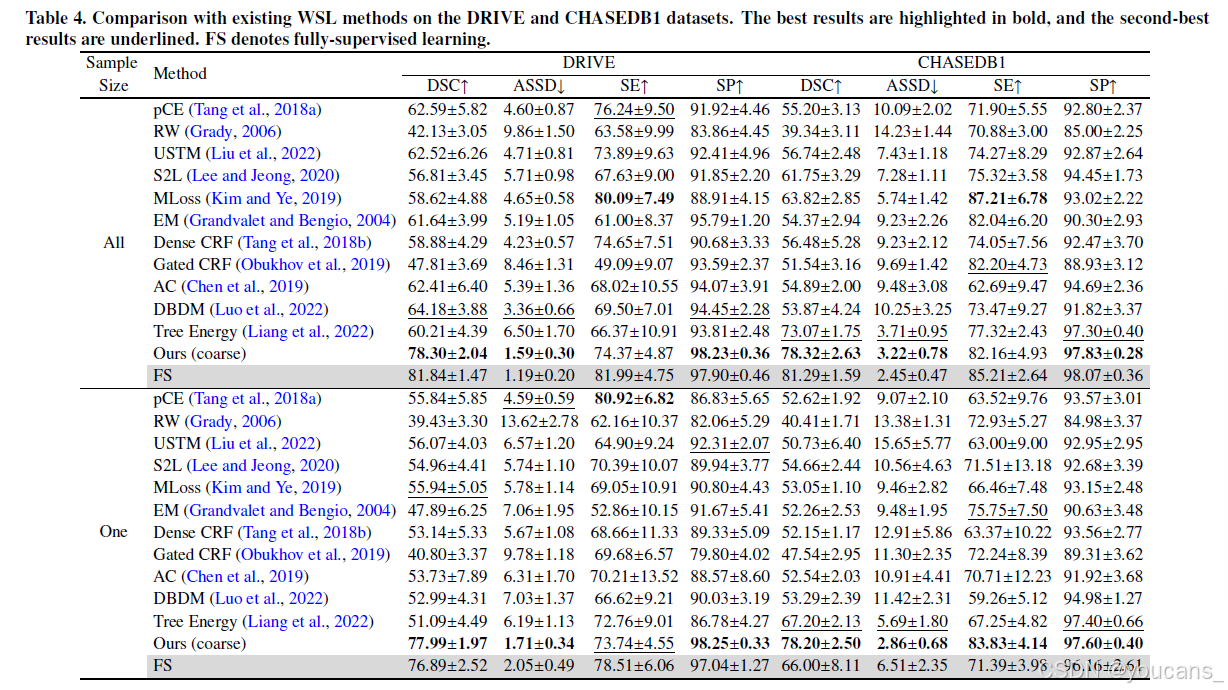
如表3 所示,pCE在大多数情况下分割性能相对较低,因其仅监督稀疏标注区域。RW显然不适用于细长曲线结构,其任意扩展在伪标签中引入大量噪声,导致性能低于基线。大多数比较方法试图通过引入各种形式的CRF损失(密集CRF和门控CRF)、结合精心设计的网络架构和一致性学习(如USTM和DBDM)或采用更先进的损失形式(如MLoss、AC和树能量损失)来生成和优化伪标签。尽管有效,这些方法仍与全监督性能存在显著差距(即使所有样本均被标注),更遑论仅标注单个样本的情况。在所有比较方法中,树能量损失在多数情况下取得第二佳性能,但仍与YoloCurvSeg存在显著差距,且高度受稀疏标注中噪声的影响。YoloCurvSeg在两种设置下的所有数据集上均取得最佳性能,大幅优于其他WSL方法。比较"All"与"One"可见,YoloCurvSeg对标注数据的样本量不敏感,在仅标注0.14%、0.03%、1.40%和0.65%像素的情况下,于四个数据集上分别达到全监督性能的96.1%、106.3%、95.3%和96.2%(以DSC计)。值得注意的是,即使仅标注单个样本,YoloCurvSeg(表格倒数第二行)的性能仍优于所有比较方法在全部样本标注时的表现(表格上半部分)。
代表性可视化结果如图7所示。
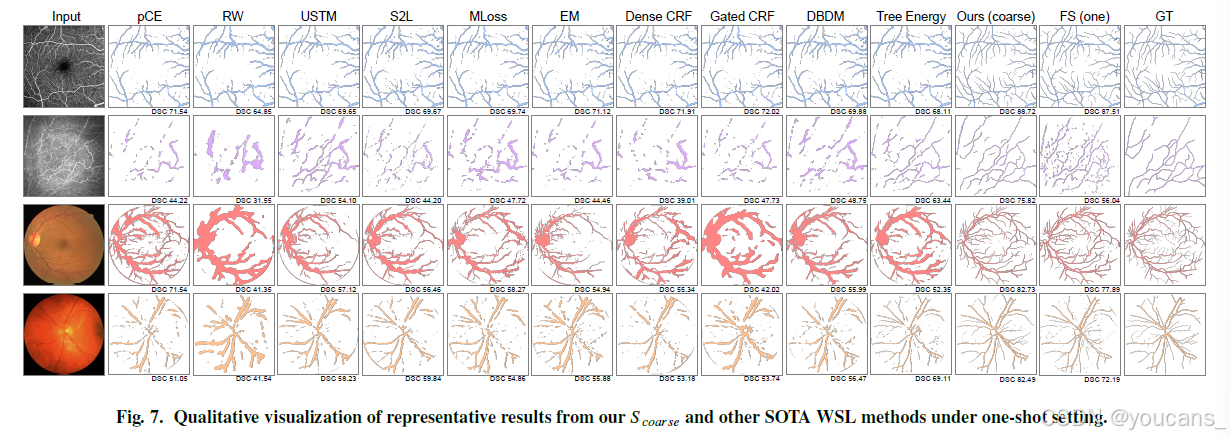
图7. 单样本设置下,本方法S-coarse与其他先进弱监督学习(WSL)方法的典型结果可视化对比
4.4.2 与NLL方法的比较
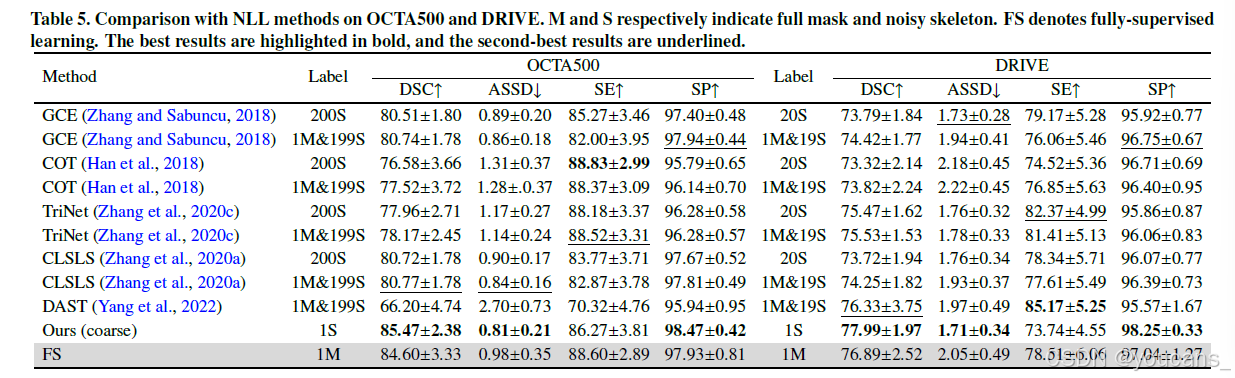
在表5中,我们还将YoloCurvSeg与几种NLL方法进行比较,包括广义交叉熵损失(GCE)、协同教学(COT)、TriNet、空间标签平滑的置信学习(CLSLS)和散度感知选择性训练(DAST),比较在OCTA500和DRIVE数据集上进行。这些方法大多允许在所有样本带噪声或混合噪声(表5中S)与全监督样本(表5中M)的条件下训练。可见当训练中包含全监督样本时,这些方法通常能取得一定程度的性能提升。然而,尽管使用一个完整掩码和多个甚至全部骨架样本,这些方法仍逊于仅使用一个骨架样本的YoloCurvSeg。我们还发现所有NLL方法的性能均不如仅使用相同单个全标注样本训练的全监督模型(表5中FS),说明在此类噪声条件下,额外带噪声标注样本对模型性能无益。
表5. NLL方法在OCTA500和DRIVE数据集上的对比结果(M表示完整掩模,S表示噪声骨架。FS代表全监督学习。最优结果以粗体显示,次优结果以下划线标注)。
4.5 鲁棒性分析与消融研究
为验证YoloCurvSeg对所选单次稀疏标注样本的鲁棒性,我们从每个数据集中随机选择10个样本,并与使用相同样本训练的全监督模型进行性能比较。如图8所示,YoloCurvSeg在几乎所有情况下均超越全监督性能,且表现出高度稳定的性能——该性能与图像/标注质量解耦,而这些质量因素会引发全监督模型的显著性能波动。除鲁棒性外,YoloCurvSeg的预测结果还具有更小的方差。这两方面表明YoloCurvSeg对样本不敏感,可降低选择错误标注样本的风险。

为研究带噪声骨架完整性对分割性能的影响,我们对骨架进行部分擦除分析实验。由于眼底图像中小血管与背景对比度低,眼底图像极易出现标注缺失。因此,我们从DRIVE数据集中选择两个样本,擦除部分小血管的带噪声骨架标签(如图11所示)。具体而言,我们分别在25号和38号样本上擦除12.55%和9.66%的标注区域。从图中可清晰观察带噪声骨架的擦除区域及其对提取背景图像和合成图像的影响。分割模型在两个完整带噪声骨架样本上的性能指标(DSC、ASSD)分别为(77.99, 1.71)和(77.74, 1.59)。擦除部分带噪声骨架并合成相应新训练集后,分割模型的性能指标变为(78.06, 1.83)和(78.11, 1.40),擦除前后仅出现微小波动,证明所提方法具有鲁棒性。这些轻微性能波动可能归因于:对带噪声骨架施加的显著扩张操作可能导致扩张掩码覆盖标注时可能遗漏的小血管;随机生成的前景曲线也可能覆盖部分小血管,进一步降低因标注遗漏引起的标签噪声。另一方面,如图8 所示,尽管样本中不可避免地存在不同程度的标注遗漏,YoloCurvSeg在所有四个数据集上均表现出稳定的单次性能——这在CORN数据集(本身已存在部分标注缺失)中尤为明显,表明我们的方法即使存在一定程度的标注缺失仍保持鲁棒性。
关于消融研究,我们首先移除修复器提取的背景库,直接进行曲线到图像的转换。如图9(a)所示,由于转换前后分布存在巨大差距(这也体现在表2的B列),合成图像呈现不真实的背景纹理。对于高分辨率图像数据集,合成图像的前景出现扭曲且未能与相应曲线掩码空间对齐——当我们移除合成器的Lc(并使用CycleGAN(Zhu等,2017)替代)时同样出现此现象(图9(b))。这表明对比合成器(尤其是Lc)对于保持相同空间位置的对应局部上下文至关重要。

为更全面证明修复器的重要性,我们尝试通过其他无监督方法生成背景图像用于后续图像合成。我们研究高斯模糊、低通滤波和中值滤波,目标是通过精细调整参数选择性移除前景血管同时最大限度保留背景细节。对于高斯模糊,我们使用9×9核大小,在OCTA500和DRIVE数据集样本上执行25次迭代。对于低通滤波,我们基于图像尺寸生成标准差为0.05的二维高斯掩码,应用这些掩码去除低频分量。对于中值滤波,我们分别对OCTA500和DRIVE数据集应用29×29和31×31的核大小。可视化结果表明中值滤波在消除前景方面相对更有效,但以牺牲细节和引入模糊为代价(图10)。如图所示最后一列,使用中值滤波背景的图像合成结果优于从曲线到真实图像的直接转换结果。但由于提供的背景相对模糊,仍存在大量伪影。随后我们从OCTA500和DRIVE中各选择十个样本进行单次粗阶段性能比较,结果如表6所示。显然,从OCTA500和DRIVE数据集中随机选择的十个样本总体性能表明:使用修复器提取的背景相比中值滤波背景在单次(仅一个样本)分割中带来约2%和4%的整体性能提升。使用带噪声骨架标注提升分割性能具有成本效益,后文所述的标注时间测量结果证明了这一点。


需指出的是,前文报告的结果代表S_coarse的性能。我们还探索了使用与不使用Dsyn时S_fine的性能。请注意"不使用Dsyn"意味着仅使用Dori(以S_coarse的预测作为伪标签)训练S_fine。为全面评估拓扑连通性和小血管分割性能,还计算了clDice(Shit等,2021)指标。从图12可见,训练数据中使用Dsyn得到的S_fine的DSC和ASSD指标略优于未使用Dsyn的S_fine,这可能归因于合成曲线具有高度连续性且能减少模型的异常预测。但clDice指标略低,这可能是因为合成数据相比真实数据不可避免存在一定强度差距(尤其在小血管区域)。此外,我们还比较了全监督模型在使用与不使用Dsyn预训练时的性能。结果表明YoloCurvSeg的合成图像作为预训练图像也具有巨大潜力:在Dsyn上预训练后在全监督数据集上微调,可进一步提升全监督模型的性能。具体而言,在OCTA500、DRIVE和CHASEDB1上,原始U-Net模型的DSC分别提高0.55、0.63、0.81,ASSD分别降低0.061、0.041、0.148。最终,通过进一步利用额外未标注数据集Dori,YoloCurvSeg(S_fine)在四个数据集上仅使用一个带噪声骨架标注即达到全监督性能(所有可用样本完整掩码)的97.00%、110.01%、97.49%和97.63%。
为更好说明本方法在真实临床场景中的省时优势,我们从四个数据集中随机选择样本(OCTA500和CORN各30样本,DRIVE和CHASEDB1各10样本),邀请两位眼科医生以带噪声骨架和完整掩码两种格式进行标注。我们发现:为6mm×6mm OCTA图像中的视网膜血管标注带噪声骨架式标签约需4.5分钟,而标注完整掩码因需仔细检查修改边缘和细节约需48分钟;类似地,为CCM图像中的角膜神经纤维、DRIVE和CHASEDB1风格视网膜眼底图像中的视网膜血管进行单样本标注分别需约1分钟、3.5分钟、3分钟(带噪声骨架)和14分钟、62分钟、55分钟(完整掩码)。图13绘制了所有评估方法(包括所有WSL方法、NLL方法和YoloCurvSeg)在单样本和全样本条件下的分割性能与标注耗时。我们的方法在所有四个任务中均以最低标注耗时(<0.3% FS)达到最高分割性能(≥97% FS)。

4.6 与最先进全监督方法的比较及讨论
在之前的实验与分析中,为公平比较,我们在各对比方法中均采用原始U-Net作为分割网络架构。为进一步证明YoloCurvSeg的实用性与可扩展性,我们结合更先进的分割网络和框架(特别是专为曲线结构分割设计的方案)进行定量与定性比较分析,并探讨未来可能的改进方向。具体而言,我们探索了两种更先进的CNN U-Net变体(EfficientUNet(Tan和Le,2019)和CS2-Net(Mou等,2021))以及四种基于Vision Transformer的分割网络(SwinUNet(Cao等,2023)、TransUNet(Chen等,2021)、UTNet(Gao等,2021)和MedFormer(Gao等,2022))在OCTA500、CORN和DRIVE数据集上的全监督性能。
所有网络均采用相同超参数(包括初始学习率、学习率策略、优化器、批量大小等)进行训练,遵循前文所述原始U-Net分割器的配置以确保公平比较。定量结果列于表7。可以观察到,由于在所有比较中保持了优越的训练范式(如优化器和学习率调度策略),原始U-Net与所有三个数据集的最佳方法相比并未表现出显著差距,甚至在部分情况下超越更先进的网络——这与nnU-Net论文(Isensee等,2021)的报告一致。在所有比较中,CS2-Net和TransUNet分别取得最佳和次佳总体性能。因此我们探索用这两种性能更优的网络替代YoloCurvSeg中的两阶段分割器,实验结果表明使用更先进的架构能进一步提升分割性能。

为阐明YoloCurvSeg与最先进全监督方法间的差距,我们在图14 中对部分示例进行定性比较。可以观察到,对于图像对比度低的结构或细小外周血管/神经(如红圈标注区域),我们的方法在准确性和结构连贯性方面与表现良好的全监督方法存在一定差距。这主要源于合成图像与真实图像间的形态学差距和强度差距。潜在解决方案包括:微调YoloCurvSeg第一组件的超参数以生成更匹配真实形状的曲线;引入图像转换/合成的新范式(如扩散模型(Ho等,2020;Cheng等,2023))以进一步提升合成图像的真实感;通过引入各种注意力机制(特别是自注意力机制)或定义保持拓扑的目标函数(Cheng等,2021a;Shit等,2021)改进网络结构——前者已在表7的实验中得到验证。另一个值得探索的未来方向是采用噪声标签学习方法(Zhang和Sabuncu,2018;Yang等,2022)训练精细阶段的分割器,因为生成的伪标签不可避免地包含噪声。

通过大量实验,我们证明 YoloCurvSeg 可应用于三种不同模态的两种最常见2D曲线结构分割任务(神经纤维和视网膜血管分割),并具有良好的泛化能力。为进一步证明所提流程的可扩展性,我们在X射线冠状动脉造影数据集DCA1(Cervantes-Sanchez等,2019)上进行了额外的合成与分割验证分析。该数据集前100个样本作为训练验证集,剩余34个样本作为测试集。所有图像调整尺寸至320×320。我们任意选择DCA1训练集中的三个样本进行全监督训练,并与YoloCurvSeg的单次性能进行比较,同时提供精细分割器的性能(实验结果详见表8)。与使用所有样本和完整掩码的全监督设置相比,YoloCurvSeg在未精确调整曲线生成器的情况下仍取得极高的单次性能。图15展示了该数据集的三个样本及YoloCurvSeg生成的对应合成图像。其他类似且可迁移的场景包括细胞膜、裂缝、航空图像中的道路及叶脉分割等。尽管如此,我们承认将YoloCurvSeg应用于3D场景(如脑血管分割和心脏血管分割)的特定曲线结构分割任务存在挑战,如Vessel-CAPTCHA(Dang等,2022)和Examinee-Examiner Network(Qi等,2021)工作中所示。这是未来探索的主要方向,当前挑战主要在于将YoloCurvSeg第二和第三组件(修复器和多层块状合成器)迁移至3D场景,这需要精心修改和设计网络的输入格式与大小以平衡性能与计算成本,此类探索一定程度上超出了本文范围。

关于YoloCurvSeg流程的一个关切点是:性能优势是源于整体框架参数还是相比其他方法存在不公平性。表9提供了YoloCurvSeg流程与对比方法在训练和测试阶段的总参数量对比。我们承认比较中可能存在一定程度的不公平性,但需强调:这种参数比较仅具说明性,因为某些方法虽未增加模型可训练参数,却需要额外非训练参数和数据处理(如树能量损失中的树滤波器和最小生成树计算、门控CRF中的能量场计算等)。此外,YoloCurvSeg的前三个组件主要贡献于图像合成过程,与分割任务具有一定解耦性,可视为不直接参与分割器训练的预处理步骤——这区别于其他在分割过程中引入额外可训练或非训练参数的方法。我们尽可能保证分割模型及其训练范式的公平性,且在YoloCurvSeg的测试或预测阶段仅需分割器参与,这与大多数对比方法一致。

5. 结论
本文提出了一种新型的稀疏标注曲线结构分割框架YoloCurvSeg。该框架是基于图像合成的流程,包含曲线生成器、修复器、合成器和两阶段分割器。通过在四个公开数据集上的大量实验,成功证明了所提框架的优越性。未来的潜在方向包括将YoloCurvSeg迁移至3D场景,以及探索更优流程以进一步缩小合成图像与真实图像之间的域差距。
利益冲突声明
作者声明不存在任何可能影响本文报道成果的已知竞争性经济利益或个人关系。
数据可用性声明
本研究使用的原始数据集均为公开数据。源代码、生成的骨架标注及合成数据集将在论文录用后发布于:https://github.com/llmir/YoloCurvSeg
参考文献
Allon, R., Aronov, M., Belkin, M., Maor, E., Shechter, M., Fabian, I.D., 2021.
Retinal microvascular signs as screening and prognostic factors for cardiac
disease: a systematic review of current evidence. Am. J. Med. 134, 36–47.
Antonelli, M., Reinke, A., Bakas, S., Farahani, K., Kopp-Schneider, A., Landman,
B.A., Litjens, G., Menze, B., Ronneberger, O., Summers, R.M., et al.,
2022. The medical segmentation decathlon. Nature communications 13,
4128.
Cao, H., Wang, Y., Chen, J., Jiang, D., Zhang, X., Tian, Q., Wang, M., 2023.
Swin-unet: Unet-like pure transformer for medical image segmentation, in:
Computer Vision–ECCV 2022 Workshops: Tel Aviv, Israel, October 23–27,
2022, Proceedings, Part III, Springer. pp. 205–218.
Cervantes-Sanchez, F., Cruz-Aceves, I., Hernandez-Aguirre, A., Hernandez-
Gonzalez, M.A., Solorio-Meza, S.E., 2019. Automatic segmentation of
coronary arteries in x-ray angiograms using multiscale analysis and artificial
neural networks. Applied Sciences 9, 5507.
Chen, J., Lu, Y., Yu, Q., Luo, X., Adeli, E., Wang, Y., Lu, L., Yuille, A.L.,
Zhou, Y., 2021. Transunet: Transformers make strong encoders for medical
image segmentation. arXiv preprint arXiv:2102.04306 .
Chen, T., Kornblith, S., Norouzi, M., Hinton, G., 2020. A simple framework for
contrastive learning of visual representations, in: International Conference
on Machine Learning (ICLR), PMLR. pp. 1597–1607.
Chen, X., Williams, B.M., Vallabhaneni, S.R., Czanner, G., Williams, R.,
Zheng, Y., 2019. Learning active contour models for medical image segmentation,
in: Proceedings of the IEEE/CVF Conference on Computer Vision
and Pattern Recognition (CVPR), pp. 11632–11640.
Cheng, M., Zhao, K., Guo, X., Xu, Y., Guo, J., 2021a. Joint topologypreserving
and feature-refinement network for curvilinear structure segmentation,
in: Proceedings of the IEEE/CVF International Conference on Computer
Vision (ICCV), pp. 7147–7156.
Cheng, P., Lin, L., Huang, Y., He, H., Luo, W., Tang, X., 2023. Learning enhancement
from degradation: A diffusion model for fundus image enhancement.
arXiv preprint arXiv:2303.04603 .
Cheng, P., Lin, L., Huang, Y., Lyu, J., Tang, X., 2021b. I-secret: Importanceguided
fundus image enhancement via semi-supervised contrastive constraining,
in: International Conference on Medical Image Computing and
Computer-Assisted Intervention (MICCAI), Springer. pp. 87–96.
Chi, L., Jiang, B., Mu, Y., 2020. Fast fourier convolution. Advances in Neural
Information Processing Systems (NeurIPS) 33, 4479–4488.
Costa, P., Galdran, A., Meyer, M.I., Abramoff, M.D., Niemeijer, M., Mendonc¸a,
A.M., Campilho, A., 2017a. Towards adversarial retinal image synthesis.
arXiv preprint arXiv:1701.08974 .
Costa, P., Galdran, A., Meyer, M.I., Niemeijer, M., Abramoff, M., Mendonc¸a,
A.M., Campilho, A., 2017b. End-to-end adversarial retinal image synthesis.
IEEE Trans. Med. Imaging 37, 781–791.
Dang, V.N., Galati, F., Cortese, R., Di Giacomo, G., Marconetto, V., Mathur,
P., Lekadir, K., Lorenzi, M., Prados, F., Zuluaga, M.A., 2022. Vesselcaptcha:
an efficient learning framework for vessel annotation and segmentation.
Medical Image Analysis 75, 102263.
Frangi, A.F., Niessen, W.J., Vincken, K.L., Viergever, M.A., 1998. Multiscale
vessel enhancement filtering, in: International Conference on Medical Image
Computing and Computer-Assisted Intervention (MICCAI), Springer.
pp. 130–137.
Fraz, M.M., Remagnino, P., Hoppe, A., Uyyanonvara, B., Rudnicka, A.R.,
Owen, C.G., Barman, S.A., 2012. An ensemble classification-based approach
applied to retinal blood vessel segmentation. IEEE Trans. Biomed.
Eng. 59, 2538–2548.
Gao, Y., Zhou, M., Liu, D., Yan, Z., Zhang, S., Metaxas, D.N., 2022. A datascalable
transformer for medical image segmentation: architecture, model
efficiency, and benchmark. arXiv preprint arXiv:2203.00131 .
Gao, Y., Zhou, M., Metaxas, D.N., 2021. Utnet: a hybrid transformer architecture
for medical image segmentation, in: International Conference on
Medical Image Computing and Computer-Assisted Intervention (MICCAI),
Springer. pp. 61–71.
Goodfellow, I., Pouget-Abadie, J., Mirza, M., Xu, B., Warde-Farley, D., Ozair,
S., Courville, A., Bengio, Y., 2020. Generative adversarial networks. Commun.
ACM 63, 139–144.
Grady, L., 2006. Random walks for image segmentation. IEEE Trans. Pattern
Anal. Mach. Intell. 28, 1768–1783.
Grandvalet, Y., Bengio, Y., 2004. Semi-supervised learning by entropy minimization.
Advances in Neural Information Processing Systems (NeurIPS)
17.
Guibas, J.T., Virdi, T.S., Li, P.S., 2017. Synthetic medical images from dual
generative adversarial networks. arXiv preprint arXiv:1709.01872 .
Han, B., Yao, Q., Yu, X., Niu, G., Xu, M., Hu, W., Tsang, I., Sugiyama, M.,
2018. Co-teaching: Robust training of deep neural networks with extremely
noisy labels. Advances in Neural Information Processing Systems (NeurIPS)
31.
Hao, J., Shen, T., Zhu, X., Liu, Y., Behera, A., Zhang, D., Chen, B., Liu, J.,
Zhang, J., Zhao, Y., 2022. Retinal structure detection in octa image via
voting-based multitask learning. IEEE Trans. Med. Imaging 41, 3969–3980.
He, K., Zhang, X., Ren, S., Sun, J., 2016. Deep residual learning for image
recognition, in: Proceedings of the IEEE/CVF Conference on Computer Vision
and Pattern Recognition (CVPR), pp. 770–778.
He, Y., Sun, H., Yi, Y., Chen, W., Kong, J., Zheng, C., 2022. Curv-net: Curvilinear
structure segmentation network based on selective kernel and multibi-
convlstm. Med. Phys. 49, 3144–3158.
Heusel, M., Ramsauer, H., Unterthiner, T., Nessler, B., Hochreiter, S., 2017.
Gans trained by a two time-scale update rule converge to a local nash equilibrium.
Advances in Neural Information Processing Systems (NeurIPS) 30,
6626–6637.
Ho, J., Jain, A., Abbeel, P., 2020. Denoising diffusion probabilistic models.
Advances in Neural Information Processing Systems 33, 6840–6851.
Holbura, C., Gordan, M., Vlaicu, A., Stoian, I., Capatana, D., 2012. Retinal
vessels segmentation using supervised classifiers decisions fusion, in: Proceedings
of 2012 IEEE International Conference on Automation, Quality
and Testing, Robotics (AQTR), IEEE. pp. 185–190.
Hoover, A., Kouznetsova, V., Goldbaum, M., 2000. Locating blood vessels in
retinal images by piecewise threshold probing of a matched filter response.
IEEE Trans. Med. Imaging 19, 203–210.
Hou, J., Ding, X., Deng, J.D., 2022. Semi-supervised semantic segmentation of
vessel images using leaking perturbations, in: Proceedings of the IEEE/CVF
Winter Conference on Applications of Computer Vision (WACV), pp. 2625–
2634.
Huang, Z., Lin, L., Cheng, P., Pan, K., Tang, X., 2022a. Ds3-net: Difficultyperceived
common-to-t1ce semi-supervised multimodal mri synthesis network,
in: International Conference on Medical Image Computing and
Computer-Assisted Intervention (MICCAI), Springer. pp. 571–581.
Huang, Z., Lin, L., Cheng, P., Peng, L., Tang, X., 2022b. Multi-modal brain
tumor segmentation via missing modality synthesis and modality-level attention
fusion. arXiv preprint arXiv:2203.04586 .
Huo, X., Xie, L., He, J., Yang, Z., Zhou,W., Li, H., Tian, Q., 2021. Atso: Asynchronous
teacher-student optimization for semi-supervised image segmentation,
in: Proceedings of the IEEE/CVF Conference on Computer Vision and
Pattern Recognition (CVPR), pp. 1235–1244.
Isensee, F., Jaeger, P.F., Kohl, S.A., Petersen, J., Maier-Hein, K.H., 2021. nnunet:
a self-configuring method for deep learning-based biomedical image
segmentation. Nature methods 18, 203–211.
Isola, P., Zhu, J.Y., Zhou, T., Efros, A.A., 2017. Image-to-image translation
with conditional adversarial networks, in: Proceedings of the IEEE/CVF
Conference on Computer Vision and Pattern Recognition (CVPR), pp.
1125–1134.
Ji, Z., Shen, Y., Ma, C., Gao, M., 2019. Scribble-based hierarchical weakly
supervised learning for brain tumor segmentation, in: International Conference
on Medical Image Computing and Computer-Assisted Intervention
(MICCAI), Springer. pp. 175–183.
Johnson, J., Alahi, A., Fei-Fei, L., 2016. Perceptual losses for real-time style
transfer and super-resolution, in: Proceedings of the European Conference
on Computer Vision (ECCV), Springer. pp. 694–711.
Kawasaki, R., Cheung, N., Wang, J.J., Klein, R., Klein, B.E., Cotch, M.F.,
Sharrett, A.R., Shea, S., Islam, F.A.,Wong, T.Y., 2009. Retinal vessel diameters
and risk of hypertension: the multiethnic study of atherosclerosis. J.
Hypertens. 27, 2386–2393.
Kervadec, H., Dolz, J., Tang, M., Granger, E., Boykov, Y., Ayed, I.B., 2019.
Constrained-cnn losses for weakly supervised segmentation. Med. Image
Anal. 54, 88–99.
Kim, B., Ye, J.C., 2019. Mumford–shah loss functional for image segmentation
with deep learning. IEEE Trans. Image Process. 29, 1856–1866.
Larrazabal, A.J., Mart´ınez, C., Glocker, B., Ferrante, E., 2020. Post-dae:
anatomically plausible segmentation via post-processing with denoising autoencoders.
IEEE Trans. Med. Imaging 39, 3813–3820.
Lee, H., Jeong, W.K., 2020. Scribble2label: Scribble-supervised cell segmentation
via self-generating pseudo-labels with consistency, in: International
Conference on Medical Image Computing and Computer-Assisted Intervention
(MICCAI), Springer. pp. 14–23.
Li, M., Zhang, Y., Ji, Z., Xie, K., Yuan, S., Liu, Q., Chen, Q., 2020. Ipn-v2 and
octa-500: Methodology and dataset for retinal image segmentation. arXiv
preprint arXiv:2012.07261 .
Liang, Z., Wang, T., Zhang, X., Sun, J., Shen, J., 2022. Tree energy loss:
Towards sparsely annotated semantic segmentation, in: Proceedings of
the IEEE/CVF Conference on Computer Vision and Pattern Recognition
(CVPR), pp. 16907–16916.
Lin, D., Dai, J., Jia, J., He, K., Sun, J., 2016. Scribblesup: Scribblesupervised
convolutional networks for semantic segmentation, in: Proceedings
of the IEEE/CVF Conference on Computer Vision and Pattern Recognition
(CVPR), pp. 3159–3167.
Lin, L., Cheng, P., Wang, Z., Li, M., Wang, K., Tang, X., 2021a. Automated
segmentation of corneal nerves in confocal microscopy via contrastive learning
based synthesis and quality enhancement, in: IEEE 18th International
Symposium on Biomedical Imaging (ISBI), IEEE. pp. 1314–1318.
Lin, L., Li, M., Huang, Y., Cheng, P., Xia, H., Wang, K., Yuan, J., Tang, X.,
2020. The sustech-sysu dataset for automated exudate detection and diabetic
retinopathy grading. Sci. Data 7, 1–10.
Lin, L.,Wang, Z.,Wu, J., Huang, Y., Lyu, J., Cheng, P.,Wu, J., Tang, X., 2021b.
Bsda-net: A boundary shape and distance aware joint learning framework
for segmenting and classifying octa images, in: International Conference on
Medical Image Computing and Computer-Assisted Intervention (MICCAI),
Springer. pp. 65–75.
Lin, L., Wu, J., Cheng, P., Wang, K., Tang, X., 2021c. Blu-gan: Bi-directional
convlstm u-net with generative adversarial training for retinal vessel segmentation,
in: BenchCouncil International Federated Intelligent Computing
and Block Chain Conferences (FICC), Springer. pp. 3–13.
Lin, L., Wu, J., Liu, Y., Wong, K.K., Tang, X., 2023. Unifying and personalizing
weakly-supervised federated medical image segmentation via adaptive
representation and aggregation. arXiv preprint arXiv:2304.05635 .
Liu, X., Yuan, Q., Gao, Y., He, K., Wang, S., Tang, X., Tang, J., Shen, D.,
2022. Weakly supervised segmentation of covid19 infection with scribble
annotation on ct images. Pattern Recognit. 122, 108341.
Luo, X., Hu, M., Liao, W., Zhai, S., Song, T., Wang, G., Zhang, S., 2022.
Scribble-supervised medical image segmentation via dual-branch network
and dynamically mixed pseudo labels supervision, in: International Conference
on Medical Image Computing and Computer-Assisted Intervention
(MICCAI), Springer. pp. 528–538.
Van der Maaten, L., Hinton, G., 2008. Visualizing data using t-sne. J. Mach.
Learn. Res. 9.
Mao, X., Li, Q., Xie, H., Lau, R.Y., Wang, Z., Paul Smolley, S., 2017. Least
squares generative adversarial networks, in: Proceedings of the IEEE/CVF
International Conference on Computer Vision (ICCV), pp. 2794–2802.
Mishra, P., Sarawadekar, K., 2019. Polynomial learning rate policy with warm
restart for deep neural network, in: TENCON IEEE Region 10 Conference
(TENCON), IEEE. pp. 2087–2092.
Mittal, S., Tatarchenko, M., Brox, T., 2019. Semi-supervised semantic segmentation
with high-and low-level consistency. IEEE Trans. Pattern Anal. Mach.
Intell. 43, 1369–1379.
Mou, L., Zhao, Y., Fu, H., Liu, Y., Cheng, J., Zheng, Y., Su, P., Yang, J.,
Chen, L., Frangi, A.F., et al., 2021. Cs2-net: Deep learning segmentation of
curvilinear structures in medical imaging. Med. Image Anal. 67, 101874.
Nguyen, U.T., Bhuiyan, A., Park, L.A., Ramamohanarao, K., 2013. An effective
retinal blood vessel segmentation method using multi-scale line detection.
Pattern Recognit. 46, 703–715.
Obukhov, A., Georgoulis, S., Dai, D., Van Gool, L., 2019. Gated crf
loss for weakly supervised semantic image segmentation. arXiv preprint
arXiv:1906.04651 .
Oord, A.v.d., Li, Y., Vinyals, O., 2018. Representation learning with contrastive
predictive coding. arXiv preprint arXiv:1807.03748 .
Painter, P.R., Ed´en, P., Bengtsson, H.U., 2006. Pulsatile blood flow, shear force,
energy dissipation and murray’s law. Theor. Biol. Medical Model. 3, 1–10.
Park, T., Efros, A.A., Zhang, R., Zhu, J.Y., 2020. Contrastive learning for
unpaired image-to-image translation, in: Proceedings of the European Conference
on Computer Vision (ECCV), Springer. pp. 319–345.
Passat, N., Ronse, C., Baruthio, J., Armspach, J.P., Maillot, C., 2006. Magnetic
resonance angiography: From anatomical knowledge modeling to vessel
segmentation. Med. Image Anal. 10, 259–274.
Peng, L., Lin, L., Cheng, P., Huang, Z., Tang, X., 2022. Unsupervised domain
adaptation for cross-modality retinal vessel segmentation via disentangling
representation style transfer and collaborative consistency learning, in: IEEE
19th International Symposium on Biomedical Imaging, IEEE. pp. 1–5.
Peng, L., Lin, L., Cheng, P., Wang, Z., Tang, X., 2021. Fargo: A joint framework
for faz and rv segmentation from octa images, in: International Workshop
on Ophthalmic Medical Image Analysis (OMIA), Springer. pp. 42–51.
Pritchard, N., Edwards, K., Dehghani, C., Fadavi, H., Jeziorska, M., Marshall,
A., Petropoulos, I.N., Ponirakis, G., Russell, A.W., Sampson, G.P., et al.,
2014. Longitudinal assessment of neuropathy in type 1 diabetes using novel
ophthalmic markers (landmark): study design and baseline characteristics.
Diabetes Res. Clin. Pract. 104, 248–256.
Qi, Y., Xu, H., He, Y., Li, G., Li, Z., Kong, Y., Coatrieux, J.L., Shu, H., Yang,
G., Tu, S., 2021. Examinee-examiner network: weakly supervised accurate
coronary lumen segmentation using centerline constraint. IEEE Transactions
on Image Processing 30, 9429–9441.
Ronneberger, O., Fischer, P., Brox, T., 2015. U-net: Convolutional networks
for biomedical image segmentation, in: International Conference on
Medical Image Computing and Computer-Assisted Intervention (MICCAI),
Springer. pp. 234–241.
Ross, A., Doshi-Velez, F., 2018. Improving the adversarial robustness and interpretability
of deep neural networks by regularizing their input gradients,
in: Proceedings of the AAAI Conference on Artificial Intelligence (AAAI),
pp. 1660–1669.
Runions, A., Fuhrer, M., Lane, B., Federl, P., Rolland-Lagan, A.G.,
Prusinkiewicz, P., 2005. Modeling and visualization of leaf venation patterns.
ACM Trans. Graph. 24, 702–711.
Runions, A., Lane, B., Prusinkiewicz, P., 2007. Modeling trees with a space
colonization algorithm, in: Proceedings of the Third Eurographics conference
on Natural Phenomena (NPH), pp. 63–70.
Shit, S., Paetzold, J.C., Sekuboyina, A., Ezhov, I., Unger, A., Zhylka, A.,
Pluim, J.P., Bauer, U., Menze, B.H., 2021. cldice-a novel topologypreserving
loss function for tubular structure segmentation, in: Proceedings
of the IEEE/CVF Conference on Computer Vision and Pattern Recognition
(CVPR), pp. 16560–16569.
Singh, N.P., Srivastava, R., 2016. Retinal blood vessels segmentation by using
gumbel probability distribution function based matched filter. Comput.
Methods Programs Biomed. 129, 40–50.
Soares, J.V., Leandro, J.J., Cesar, R.M., Jelinek, H.F., Cree, M.J., 2006. Retinal
vessel segmentation using the 2-d gabor wavelet and supervised classification.
IEEE Trans. Med. Imaging 25, 1214–1222.
Son, J., Park, S.J., Jung, K.H., 2019. Towards accurate segmentation of retinal
vessels and the optic disc in fundoscopic images with generative adversarial
networks. J. Digit. Imaging 32, 499–512.
Staal, J., Abramoff, M.D., Niemeijer, M., Viergever, M.A., Van Ginneken, B.,
2004. Ridge-based vessel segmentation in color images of the retina. IEEE
Trans. Med. Imaging 23, 501–509.
Suvorov, R., Logacheva, E., Mashikhin, A., Remizova, A., Ashukha, A., Silvestrov,
A., Kong, N., Goka, H., Park, K., Lempitsky, V., 2022. Resolutionrobust
large mask inpainting with fourier convolutions, in: Proceedings
of the IEEE/CVF Winter Conference on Applications of Computer Vision
(WACV), pp. 2149–2159.
Tan, M., Le, Q., 2019. Efficientnet: Rethinking model scaling for convolutional
neural networks, in: International conference on machine learning, PMLR.
pp. 6105–6114.
Tang, M., Djelouah, A., Perazzi, F., Boykov, Y., Schroers, C., 2018a. Normalized
cut loss for weakly-supervised cnn segmentation, in: Proceedings
of the IEEE/CVF Conference on Computer Vision and Pattern Recognition
(CVPR), pp. 1818–1827.
Tang, M., Perazzi, F., Djelouah, A., Ben Ayed, I., Schroers, C., Boykov, Y.,
2018b. On regularized losses for weakly-supervised cnn segmentation, in:
Proceedings of the European Conference on Computer Vision (ECCV), pp.
507–522.
Valvano, G., Leo, A., Tsaftaris, S.A., 2021. Learning to segment from scribbles
using multi-scale adversarial attention gates. IEEE Trans. Med. Imaging 40,
1990–2001.
Van der Walt, S., Sch¨onberger, J.L., Nunez-Iglesias, J., Boulogne, F., Warner,
J.D., Yager, N., Gouillart, E., Yu, T., 2014. scikit-image: image processing
in python. PeerJ 2, e453.
Wang, T.C., Liu, M.Y., Zhu, J.Y., Tao, A., Kautz, J., Catanzaro, B., 2018. Highresolution
image synthesis and semantic manipulation with conditional gans,
in: Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern
Recognition (CVPR), pp. 8798–8807.
Wu, Y., Xia, Y., Song, Y., Zhang, Y., Cai, W., 2018. Multiscale network followed
network model for retinal vessel segmentation, in: International Conference
on Medical Image Computing and Computer-Assisted Intervention
(MICCAI), Springer. pp. 119–126.
Xu, X., Nguyen, M.C., Yazici, Y., Lu, K., Min, H., Foo, C.S., 2022. Semicurv:
Semi-supervised curvilinear structure segmentation. IEEE Trans. on Image
Process. 31, 5109–5120.
Yang, S., Wang, G., Sun, H., Luo, X., Sun, P., Li, K., Wang, Q., Zhang, S.,
2022. Learning covid-19 pneumonia lesion segmentation from imperfect
annotations via divergence-aware selective training. IEEE J. Biomed. Health
Inform. 26, 3673–3684.
Yu, H., Shim, J.h., Kwak, J., Song, J.W., Kang, S.J., 2022. Vision transformerbased
retina vessel segmentation with deep adaptive gamma correction, in:
ICASSP 2022-2022 IEEE International Conference on Acoustics, Speech
and Signal Processing (ICASSP), IEEE. pp. 1456–1460.
Zana, F., Klein, J.C., 2001. Segmentation of vessel-like patterns using mathematical
morphology and curvature evaluation. IEEE Trans. Med. Imaging
10, 1010–1019.
Zhang, J., Chen, Y., Bekkers, E.,Wang, M., Dashtbozorg, B., ter Haar Romeny,
B.M., 2017. Retinal vessel delineation using a brain-inspired wavelet transform
and random forest. Pattern Recognit. 69, 107–123.
Zhang, M., Gao, J., Lyu, Z., Zhao, W., Wang, Q., Ding, W., Wang, S., Li,
Z., Cui, S., 2020a. Characterizing label errors: confident learning for noisylabeled
image segmentation, in: International Conference on Medical Image
Computing and Computer-Assisted Intervention (MICCAI), Springer. pp.
721–730.
Zhang, P., Zhong, Y., Li, X., 2020b. Accl: Adversarial constrained-cnn
loss for weakly supervised medical image segmentation. arXiv preprint
arXiv:2005.00328 .
Zhang, T., Fu, H., Zhao, Y., Cheng, J., Guo, M., Gu, Z., Yang, B., Xiao, Y.,
Gao, S., Liu, J., 2019. Skrgan: Sketching-rendering unconditional generative
adversarial networks for medical image synthesis, in: International Conference
on Medical Image Computing and Computer-Assisted Intervention
(MICCAI), Springer. pp. 777–785.
Zhang, T., Yu, L., Hu, N., Lv, S., Gu, S., 2020c. Robust medical image segmentation
from non-expert annotations with tri-network, in: International
Conference on Medical Image Computing and Computer-Assisted Intervention
(MICCAI), Springer. pp. 249–258.
Zhang, Z., Sabuncu, M., 2018. Generalized cross entropy loss for training
deep neural networks with noisy labels. Advances in Neural Information
Processing Systems (NeurIPS) 31.
Zhao, H., Li, H., Maurer-Stroh, S., Cheng, L., 2018. Synthesizing retinal and
neuronal images with generative adversarial nets. Med. Image Anal. 49,
14–26.
Zhao, Y., Zhang, J., Pereira, E., Zheng, Y., Su, P., Xie, J., Zhao, Y., Shi, Y.,
Qi, H., Liu, J., et al., 2020. Automated tortuosity analysis of nerve fibers in
corneal confocal microscopy. IEEE Trans. Med. Imaging 39, 2725–2737.
Zhao, Y., Zheng, Y., Liu, Y., Zhao, Y., Luo, L., Yang, S., Na, T., Wang, Y.,
Liu, J., 2017. Automatic 2-d/3-d vessel enhancement in multiple modality
images using a weighted symmetry filter. IEEE Trans. Med. Imaging 37,
438–450.
Zhou, B., Lapedriza, A., Khosla, A., Oliva, A., Torralba, A., 2017. Places: A
10 million image database for scene recognition. IEEE Trans. Pattern Anal.
Mach. Intell. 40, 1452–1464.
Zhou, Y., Wang, B., He, X., Cui, S., Shao, L., 2020. Dr-gan: conditional
generative adversarial network for fine-grained lesion synthesis on diabetic
retinopathy images. IEEE Journal of Biomedical and Health Informatics 26,
56–66.
Zhou, Z., Sodha, V., Rahman Siddiquee, M.M., Feng, R., Tajbakhsh, N., Gotway,
M.B., Liang, J., 2019. Models genesis: Generic autodidactic models
for 3d medical image analysis, in: International Conference on Medical Image
Computing and Computer-Assisted Intervention (MICCAI), Springer.
pp. 384–393.
Zhu, J.Y., Park, T., Isola, P., Efros, A.A., 2017. Unpaired image-to-image
translation using cycle-consistent adversarial networks, in: Proceedings of
the IEEE/CVF International Conference on Computer Vision (ICCV), pp.
2223–2232.
版权说明:
本文由 youcans@xidian 对论文 YoloCurvSeg: You Only Label One Noisy Skeleton for Vessel-style Curvilinear Structure Segmentation 进行摘编和翻译。该论文版权属于原文期刊和作者,本译文只供研究学习使用。
youcans@xidian 作品,转载必须标注原文链接:
【医学影像 AI】YoloCurvSeg:仅需标注一个带噪骨架即可实现血管状曲线结构分割(https://youcans.blog.csdn.net/article/details/151262171)
Crated:2025-09
