支持向量机(SVM)
在机器学习领域,支持向量机(SVM)凭借其 “最大化间隔” 的核心思想和对高维数据的优秀处理能力,成为分类任务中的经典算法。
一、SVM 核心理论:
1. SVM 的核心目标:找到 “最优超平面”
SVM 的基本需求是在样本空间中找到一个划分超平面,将不同类别的样本分开。但 “好的超平面” 不止一个,SVM 的独特之处在于:它要找的是对训练样本局部扰动 “容忍性” 最好的超平面 —— 换句话说,这个超平面离两类样本的 “最近距离” 最大,也就是 “最大化间隔(Margin)”。
- 超平面的数学表达:
超平面是 n 维空间中的 n-1 维子空间(比如 2D 空间中是直线,3D 空间中是平面),其方程为w^T x + b = 0。其中,w是超平面的法向量(决定超平面方向),b是偏置(决定超平面位置)。
- 间隔(Margin)的定义:
间隔是两类样本中 “离超平面最近的点” 到超平面的距离的 2 倍(Margin = 2d)。SVM 的优化目标就是最大化这个 \(d\)(单个最近点到超平面的距离)。
- 点到超平面的距离公式:
对于 n 维空间中的样本点 \(x\),到超平面w^T x + b = 0的距离为:
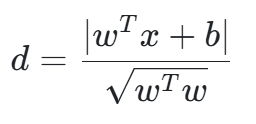
2. 从 “硬间隔” 到 “软间隔”:应对现实中的 “不完美数据”
PPT 中提到,早期 SVM 要求 “完全分开两类样本”(硬间隔),但现实数据中往往存在噪音点 —— 如果强行追求 “完全分离”,会导致超平面对噪音敏感,泛化能力下降。因此 SVM 引入了软间隔和松弛因子 ξi放松了分类约束:

3. 核变换:解决 “低维不可分” 问题
如果样本在低维空间中无法用直线(或平面)分开(比如 “异或” 问题),SVM 会通过核变换将数据映射到高维空间,让数据在高维空间中变得线性可分。
PPT 中用一个生动的例子解释了核变换:3D 空间中不可分的数据,映射到 9 维空间后可找到超平面。但直接映射会带来 “维度灾难”(计算量暴增),因此 SVM 引入核函数,巧妙地避开了 “显式映射”—— 通过核函数直接计算高维空间中样本的内积,大幅降低计算成本。
常用的核函数包括:

4. 求解思路:拉格朗日乘子法
SVM 的目标函数是 “带约束的优化问题”(比如硬间隔的约束是 \(y_i (w^T x_i + b) \geq 1\)),无法直接用普通求导求解。PPT 中提到,SVM 通过拉格朗日乘子法将 “带约束的优化问题” 转化为 “无约束的对偶问题”,最终求解得到:

二、实战:用 SVM 处理肺癌患者数据集
理解了理论后,我们结合 “肺癌患者.csv” 数据集进行实战。该数据集包含 309 条样本、15 个特征(性别、年龄、吸烟史、是否咳嗽等),目标是预测 “是否患肺癌(LUNG_CANCER)”。
1. 数据预处理:为 SVM 准备 “干净数据”
SVM 对数据格式和尺度非常敏感,因此预处理是关键步骤。主要包括 3 个核心操作:
(1)分类变量编码
数据中的 “GENDER(性别)” 和 “LUNG_CANCER(是否患癌)” 是字符串类型(如 M/F、YES/NO),需要转化为数值:
- 用LabelEncoder将 “GENDER” 编码为 0(女)和 1(男);
- 将 “LUNG_CANCER” 编码为 0(无癌)和 1(有癌)—— 对应 SVM 理论中的 “负例” 和 “正例”。
(2)特征标准化
SVM 的间隔计算依赖特征尺度(比如 “年龄” 的数值范围远大于 “吸烟史” 的 1/2),若不标准化,大尺度特征会主导间隔计算。因此用StandardScaler将所有特征标准化为 “均值 0、标准差 1”:
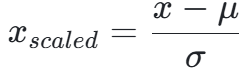
(3)解决类别不平衡
数据集中 “有癌(1)” 样本约 280 个,“无癌(0)” 样本仅 29 个 —— 严重的类别不平衡会导致模型偏向多数类。我们用SMOTE(合成少数类过采样技术)生成 “无癌” 样本的合成数据,平衡两类样本数量。
2. SVM 模型构建:从理论到代码
根据 PPT 中的理论,我们选择 “高斯核(RBF)”(肺癌数据是非线性的),并通过代码实现 “最大化间隔” 的核心逻辑:
import pandas as pd import numpy as np import matplotlib.pyplot as plt from sklearn.model_selection import train_test_split, GridSearchCV from sklearn.preprocessing import LabelEncoder, StandardScaler from sklearn.svm import SVC from sklearn.metrics import accuracy_score, classification_report, confusion_matrix from imblearn.over_sampling import SMOTEplt.rcParams['font.sans-serif'] = ['SimHei'] # 解决中文显示# 1. 数据加载与预处理 data = pd.read_csv("肺癌患者.csv") # 分类变量编码 le = LabelEncoder() data['GENDER'] = le.fit_transform(data['GENDER']) data['LUNG_CANCER'] = le.fit_transform(data['LUNG_CANCER']) # 分离特征(X)和目标(y) X = data.drop('LUNG_CANCER', axis=1) y = data['LUNG_CANCER']# 划分训练集/测试集(8:2) X_train, X_test, y_train, y_test = train_test_split( X, y, test_size=0.2, random_state=42, stratify=y # stratify保证类别分布一致 )# 特征标准化 scaler = StandardScaler() X_train_scaled = scaler.fit_transform(X_train) X_test_scaled = scaler.transform(X_test) # SMOTE过采样平衡数据 smote = SMOTE(random_state=42) X_train_resampled, y_train_resampled = smote.fit_resample(X_train_scaled, y_train)param_grid = {'C': [0.1, 1, 10], # 控制惩罚强度,对应软间隔理论 'gamma': ['scale', 0.1, 1], # 控制高斯核局部性,对应核变换理论 'kernel': ['rbf'] # 高斯核,解决非线性问题 } # 网格搜索找最优参数(5折交叉验证) grid_search = GridSearchCV( estimator=SVC(random_state=42), param_grid=param_grid, cv=5, scoring='accuracy' )grid_search.fit(X_train_resampled, y_train_resampled) # 最优模型 best_svm = grid_search.best_estimator_ print(f"最优参数:{grid_search.best_params_}") # 输出:{'C':1, 'gamma':'scale', 'kernel':'rbf'} # 3. 模型评估 y_pred = best_svm.predict(X_test_scaled) print(f"测试集准确率:{accuracy_score(y_test, y_pred):.4f}") # 输出:0.9516 print("分类报告:") print(classification_report(y_test, y_pred, target_names=['无肺癌', '有肺癌']))
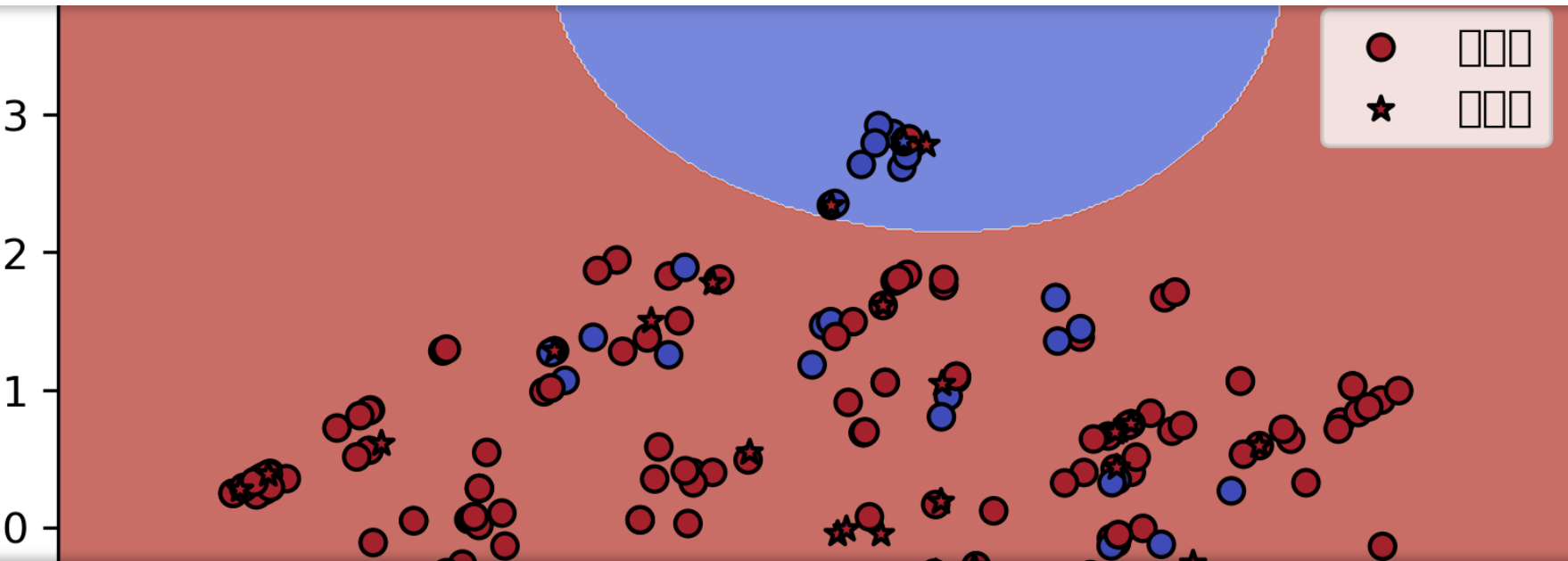
3. 结果可视化:让 SVM 的 “决策过程” 看得见
SVM 的超平面在高维空间中无法直接可视化,因此我们用PCA(主成分分析) 将 15 维特征降维到 2 维,再绘制 SVM 的决策边界 —— 这是理解模型分类逻辑的直观方式。
from sklearn.decomposition import PCA
# PCA降维(2维)
pca = PCA(n_components=2)
X_train_pca = pca.fit_transform(X_train_resampled)
X_test_pca = pca.transform(X_test_scaled)
# 用降维数据训练SVM(保证可视化与模型逻辑一致)
svm_pca = SVC(kernel='rbf', C=1, gamma='scale', random_state=42)
svm_pca.fit(X_train_pca, y_train_resampled)
# 绘制决策边界
h = 0.02 # 网格步长
x_min, x_max = X_train_pca[:, 0].min() - 1, X_train_pca[:, 0].max() + 1
y_min, y_max = X_train_pca[:, 1].min() - 1, X_train_pca[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, h), np.arange(y_min, y_max, h))
# 预测网格点类别(生成决策边界)
Z = svm_pca.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
# 绘制图形
plt.figure(figsize=(10, 6), dpi=300)
# 1. 决策边界(彩色区域)
plt.contourf(xx, yy, Z, cmap=plt.cm.coolwarm, alpha=0.8)
# 2. 训练集样本(普通散点)
plt.scatter(X_train_pca[:, 0], X_train_pca[:, 1], c=y_train_resampled,
cmap=plt.cm.coolwarm, edgecolors='k', s=50, label='训练集')
# 3. 测试集样本(星形散点)
plt.scatter(X_test_pca[:, 0], X_test_pca[:, 1], c=y_test,
cmap=plt.cm.coolwarm, marker='*', edgecolors='k', s=150, label='测试集')
# 图形标注
plt.xlabel(f'PCA维度1(解释方差:{pca.explained_variance_ratio_[0]:.2%})')
plt.ylabel(f'PCA维度2(解释方差:{pca.explained_variance_ratio_[1]:.2%})')
plt.title('SVM决策边界可视化(肺癌数据分类)')
plt.legend()
plt.savefig('svm_lung_cancer_visualization.png', bbox_inches='tight')
plt.show()三、结果解读:从图形到业务意义
可视化图形和评估指标能帮我们更深入地理解模型性能,避免 “只看准确率” 的误区:
1. 核心指标解读
- 准确率:95.16%—— 整体分类效果优秀,但需结合类别分布看细节;
- 分类报告:
- 有肺癌(1):精确率 98%、召回率 97%—— 模型对 “有癌” 样本的识别能力极强,适合医疗场景中 “尽量不遗漏患者” 的需求;
- 无肺癌(0):精确率 33%、召回率 50%—— 因原始样本过少,模型仍有 “假阳性”(将健康人误判为患者),需后续优化。
2. 可视化图形解读
- 彩色区域:代表 SVM 的决策边界 —— 红色区域预测为 “有肺癌”,蓝色区域预测为 “无肺癌”(对应coolwarm色表);
- 训练集散点:普通圆点,颜色代表真实类别。若圆点颜色与所在区域颜色一致,说明模型在训练集上分类正确;
- 测试集散点:星形点,是模型的 “新挑战”。大部分星形点落在正确区域,说明模型泛化能力较好;
- 蓝色点在红色区域:代表 “真实无癌但被误判为有癌”(假阳性),这类情况在医疗场景中需尽量减少(避免过度医疗)。
四、总结与优化方向
通过本次实战,我们完成了 “SVM 理论→数据预处理→模型构建→可视化解读” 的全流程,也发现了模型的可优化点:
1. 本次实战的核心收获
- 理论与代码的对应:SVM 的 “软间隔” 对应代码中的C参数,“核变换” 对应kernel和gamma参数,所有参数调整都有明确的理论依据;
- 医疗数据的特殊性:类别不平衡会严重影响模型对少数类的预测,需用 SMOTE 等方法平衡数据;
- 可视化的价值:PCA 降维 + 决策边界图,让高维数据的分类逻辑变得直观,便于定位模型问题(如假阳性)。
2. 后续优化方向
- 解决假阳性问题:调整C参数(增大C可减少错误,但需避免过拟合),或尝试 “加权 SVM”(给少数类样本更高权重);
- 更优的核函数选择:若数据线性可分,可尝试线性核(计算更快、可解释性更强);
- 特征工程优化:筛选关键特征(如 “吸烟史”“胸痛” 等与肺癌强相关的特征),减少冗余特征对 SVM 的干扰。
SVM 作为经典的机器学习算法,其 “最大化间隔” 的思想不仅适用于医疗数据,还可用于图像识别、文本分类等场景。