从0开始学习R语言--Day49--Lasso-Cox 回归
对于正常的医疗数据来说(指的是每个变量都有对应的结局数,不出现某类情况病人完全死亡或完全存活),我们对其做单因素回归确定变量的显著性,再进一步做多因素回归是我们正常的分析思路。但往往我们都会看到存在完全分离的变量,而且由于医疗数据往往都是指标或者是否患某种共病、是否用某种药物,不好直接作变量的删除。
而Lasso-Cox 回归可以直接处理这类数据,这个方法会给每个变量一个惩罚项系数,根据系数给予权重,自动筛选出对生存时间影响最大的预测变量,而且对于高度相关,其能够处理这个许多变量共线性大的问题,且在过程中是正则化了变量的值,能很好地防止过拟合。
以下是一个例子:
# 加载必要的包
library(glmnet)
library(survival)
library(ggplot2)# 设置随机种子保证可重复性
set.seed(123)# 生成模拟数据
n <- 200 # 样本量
p <- 30 # 变量数(其中10个是有真实效应的)# 生成预测变量矩阵 (包含一些相关变量)
X <- matrix(rnorm(n * p), n, p)
colnames(X) <- paste0("Gene", 1:p)# 使前10个变量有真实效应
true_beta <- c(rep(0.5, 5), rep(-0.3, 5), rep(0, p-10))# 生成生存时间 (指数分布)
hazard <- exp(X %*% true_beta)
surv_time <- rexp(n, rate = hazard)# 生成删失时间 (约30%的删失)
cens_time <- runif(n, 0, quantile(surv_time, 0.8))
status <- ifelse(surv_time <= cens_time, 1, 0)
obs_time <- pmin(surv_time, cens_time)# 创建生存数据框
surv_data <- data.frame(time = obs_time, status = status, X)# 传统Cox回归 (只使用前5个变量,因为变量多时会报错)
cox_fit <- coxph(Surv(time, status) ~ Gene1 + Gene2 + Gene3 + Gene4 + Gene5, data = surv_data)
summary(cox_fit)# 准备数据
x <- as.matrix(surv_data[, 3:ncol(surv_data)]) # 预测变量
y <- Surv(surv_data$time, surv_data$status) # 生存时间与状态# 拟合Lasso-Cox模型
lasso_fit <- glmnet(x, y, family = "cox", alpha = 1) # alpha=1表示Lasso# 交叉验证选择最优lambda
cv_fit <- cv.glmnet(x, y, family = "cox", alpha = 1)# 绘制交叉验证结果
plot(cv_fit)# 最优lambda值
best_lambda <- cv_fit$lambda.min# 使用最优lambda重新拟合模型
final_lasso <- glmnet(x, y, family = "cox", alpha = 1, lambda = best_lambda)# 查看选择的变量及其系数
coef(final_lasso)# 提取非零系数
selected_vars <- coef(final_lasso)@Dimnames[[1]][which(coef(final_lasso) != 0)]
selected_coef <- coef(final_lasso)[which(coef(final_lasso) != 0)]
results <- data.frame(Variable = selected_vars, Coefficient = selected_coef)# 绘制系数图
ggplot(results, aes(x = Variable, y = Coefficient)) +geom_bar(stat = "identity") +coord_flip() +ggtitle("Selected Variables in Lasso-Cox Model") +theme_minimal()输出:
Call:
coxph(formula = Surv(time, status) ~ Gene1 + Gene2 + Gene3 + Gene4 + Gene5, data = surv_data)n= 200, number of events= 121 coef exp(coef) se(coef) z Pr(>|z|)
Gene1 0.56683 1.76267 0.10197 5.559 2.71e-08 ***
Gene2 0.25203 1.28664 0.09467 2.662 0.007759 **
Gene3 0.33068 1.39192 0.10333 3.200 0.001374 **
Gene4 0.33642 1.39993 0.09460 3.556 0.000376 ***
Gene5 0.27406 1.31529 0.09266 2.958 0.003098 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1exp(coef) exp(-coef) lower .95 upper .95
Gene1 1.763 0.5673 1.443 2.153
Gene2 1.287 0.7772 1.069 1.549
Gene3 1.392 0.7184 1.137 1.704
Gene4 1.400 0.7143 1.163 1.685
Gene5 1.315 0.7603 1.097 1.577Concordance= 0.709 (se = 0.026 )
Likelihood ratio test= 63.3 on 5 df, p=3e-12
Wald test = 64.66 on 5 df, p=1e-12
Score (logrank) test = 65.3 on 5 df, p=1e-1230 x 1 sparse Matrix of class "dgCMatrix"s0
Gene1 6.201753e-01
Gene2 2.915195e-01
Gene3 3.549350e-01
Gene4 3.213577e-01
Gene5 3.288011e-01
Gene6 -9.586332e-02
Gene7 -2.326214e-01
Gene8 -2.277405e-01
Gene9 -2.113369e-01
Gene10 -2.877383e-01
Gene11 .
Gene12 -4.402928e-02
Gene13 .
Gene14 .
Gene15 .
Gene16 .
Gene17 6.981656e-05
Gene18 .
Gene19 .
Gene20 -5.708374e-02
Gene21 .
Gene22 4.294571e-02
Gene23 2.453387e-02
Gene24 .
Gene25 .
Gene26 .
Gene27 1.483148e-01
Gene28 .
Gene29 .
Gene30 . 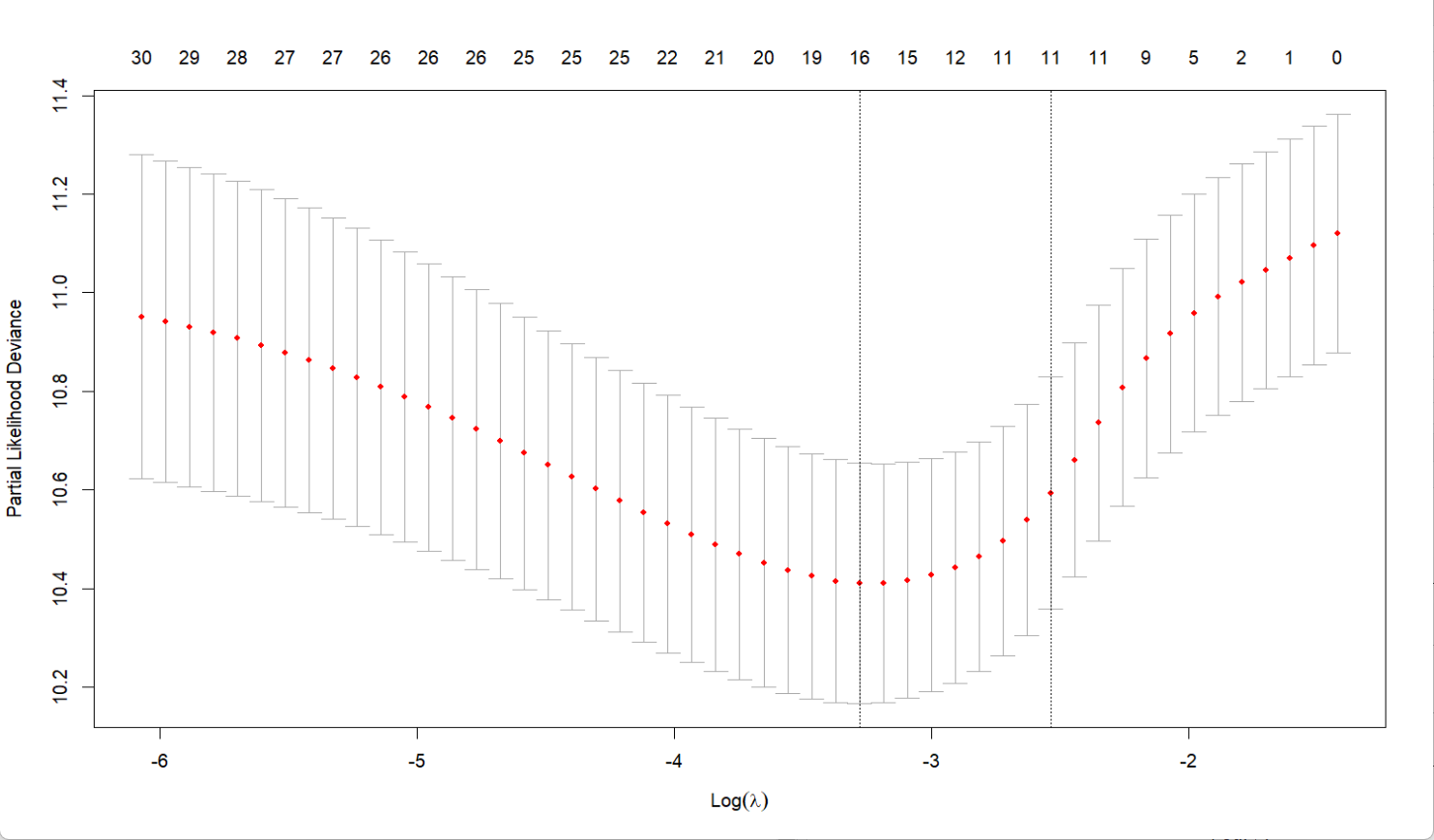
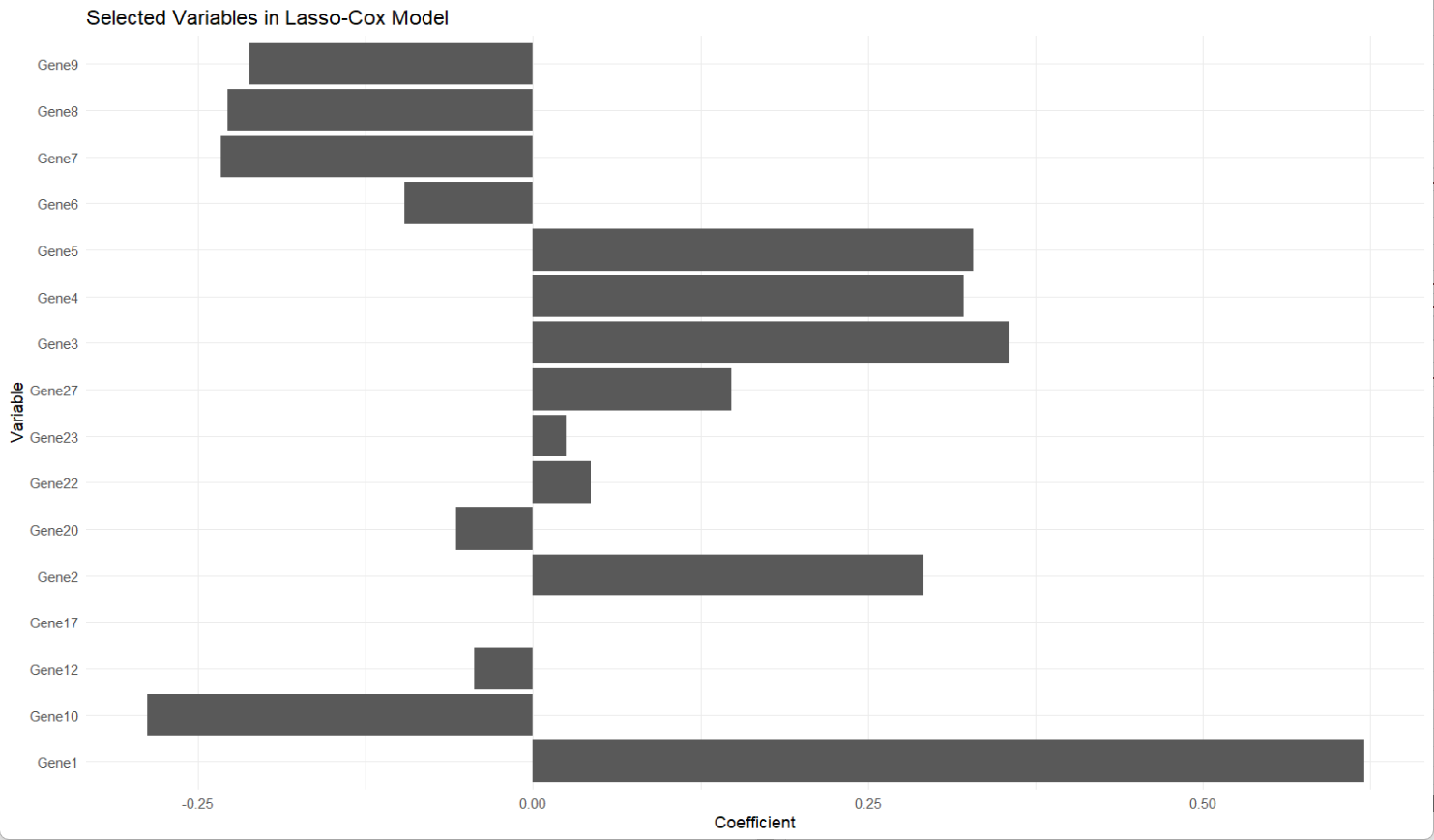
从输出中可以看到,模型的c-index值为0.7,p值小于0.05,可解释性较好;结合第一张图,我们会选择中间偏右一些的lambda参数,使得保持较好的拟合度的同时,维持变量数量不要太少;而第二张图显示Gene1、Gene3、Gene5、Gene10的系数较大,说明它们对生存结局的影响更显著。
