DNA与蛋白相互作用检测技术ChIP-qPCR
DNA与蛋白质作为生命活动的核心分子,DNA不仅承载遗传信息,更通过与蛋白质的动态互作调控基因表达、维持染色质结构,并在疾病发生发展中起关键作用。二者的功能异常常导致疾病发生,因此解析其相互作用机制对揭示疾病本质具有重要意义。为助力科研突破,伯远生物提供高效的蛋白与DNA、蛋白与RNA、蛋白与蛋白互作研究解决方案(详见【业务介绍】蛋白互作技术大全)
其中染色质免疫共沉淀技术(Chromatin Immunoprecipitation,ChIP)是研究体内蛋白质与DNA相互作用的“黄金标准”,通过特异性抗体免疫共沉淀目的蛋白和与其互作结合的DNA区段。利用ChIP,我们能精准定位基因组特定区域的组蛋白修饰或转录因子结合情况,为表观遗传学和基因调控研究提供核心数据。在ChIP-seq分析后,一般常需对筛选出的靶基因进行结合验证。此时,ChIP-qPCR凭借其体内富集优势(相比体外实验,ChIP-qPCR体内富集能够更真实地反映结合状态的特性)成为首选方法。目前ChIP定量主要采用两种策略:
双标准曲线法: 需同时绘制目的基因和看家基因的标准曲线,若标准品与待测样品性质差异较大,可能引入扩增效率误差;
双ΔCT法: 通过做校正,避免标准曲线偏差,现已成为主流分析方案。
一、ChIP-qPCR实验流程
(一)ChIP免疫共沉淀富集DNA
1、实验材料的交联处理:甲醛交联将DNA结合蛋白和DNA紧密固定;
2、实验材料的染色质抽提;
3、染色质的超声波片段化:通过超声破碎将交联后的染色质打断成小片段,片段范围在200-700bp左右;
4、染色质前处理和免疫共沉淀:利用抗原抗体的特异性识别反应,将与目的蛋白相结合的DNA片段沉淀下来。Protein A/G磁珠拉取抗体-蛋白-DNA复合物;
5、洗脱蛋白质-DNA复合物;
6、纯化 DNA。
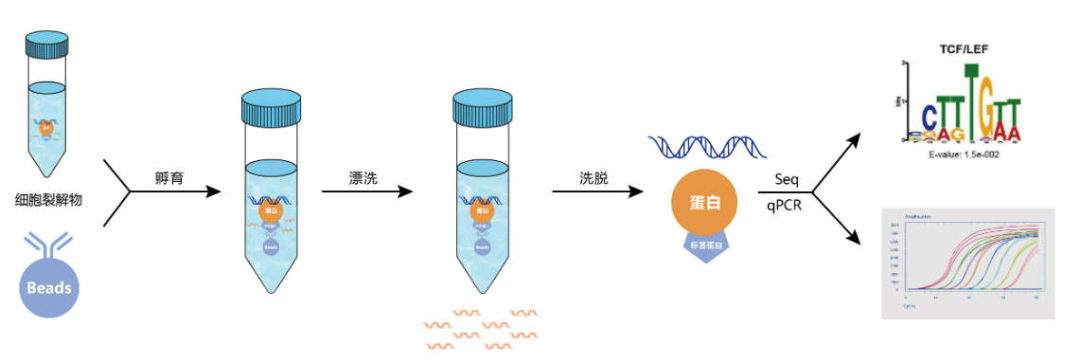
图 ChIP-seq /qPCR流程图。
(二)引物设计
1、潜在靶点选择
(1)基于前期测序结果:若已有ChIP-seq数据,可直接针对预测的motif关联peak区域设计引物,或通过IGV等可视化工具精确定位peak核心区序列;
(2)借助公共数据库:若无测序数据,可检索Cistrome DB等ChIP数据库,参考同类研究中目标蛋白的结合位点信息进行验证性设计;
(3)利用保守性生信预测:对于人、小鼠等常规模型物种,还可通过Jaspar等数据库预测转录因子结合位点,并在靶基因序列中筛选潜在互作区域。
2、引物设计建议
一般ChIP-qPCR的引物长度不宜过长,扩增片段大小需在200bp以内。引物设计时与普通荧光定量引物设计原理一致(可使用引物设计软件或常用网站,注意区分cDNA和genomic DNA),需满足多项常规原则,包括引物长度、Tm退火温度及GC含量等关键参数。
Tips:
(1)ChIP-qPCR引物设计直接影响实验的灵敏度和特异性,建议在设计完后对引物的特异性进行验证后使用,有效避免非特异性扩增对实验数据的干扰。
(2)ChIPprimersDB数据库网站收录了部分ChIP经过验证的qPCR引物可供参考。
(3)如果需特定序列位置的引物设计,您也可提供详细序列信息委托我们完成。
(三)qPCR及数据分析
1、PCR 反应
对每个样本(单个样本通常做3个技术重复)的Input、IP、IgG-DNA进行qPCR实验,统计每个复孔的CT值(即荧光信号达到设定阈值时的循环数),随后利用2-ΔΔ CT法进行富集倍率的计算。2-ΔΔ CT法是一种基于实时荧光定量PCR循环阈值的标准化方法。该方法首先通过Input对照(片段化并解交联的纯化DNA,用于消除样本背景差异)对免疫沉淀(IP)产物进行均一化处理,得到ΔCT(IP CT值 - Input CT值);同时,阴性对照抗体IgG的富集产物也需用相同Input进行均一化处理。随后,计算IP与IgG均一化后的CT值差异(ΔΔCT = ΔCT_IP - ΔCT_IgG),最终通过公式2-ΔΔ CT将循环数差异转换为目标位点的富集倍数,从而定量分析特定蛋白质与DNA的结合强度。这一方法通过双重标准化(Input和IgG对照)有效减少了实验系统误差,提高了数据可比性。
2、数据计算
Input通常为总量的1/10,即稀释了10倍,Log2 (Input Dilution Factor)≈3.3
ΔCt [normalized IP] = (Ct [IP] - (Ct [Input] -Log2 (Input Dilution Factor))
Input Dilution Factor(IDF)= (fraction of the Input chromatin saved) -1
ΔΔCt [IP/NIS] = ΔCt [normalized IP] - ΔCt [ IgG ]
Fold Enrichment = 2^ (-ΔΔCt [IP/NIS])
二、ChIP-qPCR技术优势
1、更真实地反映结合状态:能够直接反映细胞内原位的蛋白质-DNA互作状态,相较于EMSA或Dual-LUC等体外实验,其数据更贴近生理真实环境,具有更高的生物学说服力。
2、高灵敏性与特异性:通过抗体特异性富集目标蛋白-DNA复合物,结合qPCR的精准扩增检测,可准确测定特定基因或区域(如启动子、增强子)的结合情况,显著降低假阳性风险。
3、直接定量能力:无需依赖间接信号转换,通过标准曲线直接计算DNA结合位点的拷贝数,提供靶标区域结合的定量化数据(如富集倍数)。
4、靶向分析灵活性:可针对特定基因调控元件(如转录因子结合位点、组蛋白修饰区域)进行定制化检测,支持高分辨率的功能机制研究。
5、多技术联用潜力:可与RNA-seq(验证转录调控效应)、CRISPR编辑(验证功能位点)或ChIP-seq(先导筛选)等技术协同,形成多维研究体系。
三、ChIP-qPCR应用场景
1、转录因子与DNA互作研究:鉴定转录因子在特定基因组位点(如启动子、增强子)的结合情况。
验证已知转录因子是否结合到目标基因的调控区域。
研究转录因子在疾病或刺激条件下的动态结合变化。
2. 组蛋白修饰分析:检测组蛋白修饰(如H3K27ac激活标记、H3K27me3抑制标记)在基因区域的富集程度。
关联修饰与基因表达状态(如高H3K4me3水平常出现在活跃转录的启动子区)。
比较不同细胞类型或发育阶段中修饰模式的差异(如干细胞分化过程中修饰重编程)。
3、疾病机制与生物标志物探索
癌症中异常转录因子结合。
神经退行性疾病中组蛋白修饰异常。
4. 药物开发与表观遗传治疗
确认药物是否通过改变转录因子结合或修饰发挥作用。
定量评估表观遗传药物处理后目标位点的修饰变化。
四、ChIP-qPCR交付内容
1、前期质检WB验证结果;
2、包括实验过程、实验试剂与设备等的详细实验报告;
3、所有实验的原始数据,包含原始下机EDS文件、结果导出文件等;
4、验证所用的靶基因引物序列;
5、相对表达量检测分析结果;
6、结题后剩余的富集DNA(如有剩余可选择返样)。
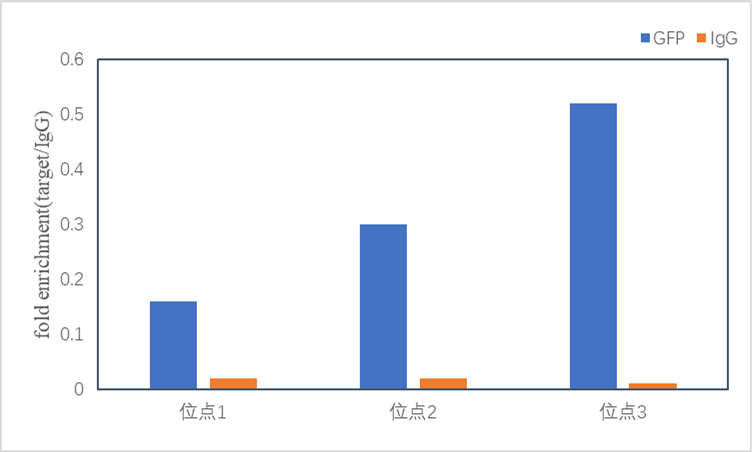
图 ChIP-qPCR部分结果示例。
五、常见问题
1、什么是Input、IgG样本?
Input样本是实验片段化之后取出来的对照,只是不做后续的IP过程,相当于Input含有样本的所有蛋白和DNA等物质,经纯化后作为计算中的对照样本。而IgG是作为阴性对照样本,理论上不具备特异性结合DNA片段的能力,其CT值在富集过程中可作为阴性参照基线,通过与目标抗体(IP组)的CT值对比计算富集倍率(Fold Enrichment)。当IP组与IgG组的CT值差异越大时,表明目标DNA片段的富集特异性越显著。
2、ChIP-qPCR是否需要做生物学重复?
一般是建议2-3个生物学重复,确保结果可靠性和科学严谨性,复杂样本或高变异情况:可能需要更多重复(如临床样本或异质性组织)。
(1)减少个体差异影响:不同样本(如细胞、动物)之间存在天然变异,生物学重复可评估这种变异对结果的干扰。
(2)提高统计效力:通过重复实验能区分真实信号与随机误差,满足统计分析的基本要求。
(3)大多数期刊和领域的会有要求。
六、案例展示
题目:Thiostrepton induces ferroptosis in pancreatic cancer cells through STAT3/GPX4 signalling.
期刊:Cell death & disease(IF=8.1)
研究背景:铁死亡的关键调控因子谷胱甘肽过氧化物酶4(Glutathione Peroxidase 4,GPX4)通过催化脂质过氧化物转化为无毒的脂质醇,从而有效抑制铁死亡。尽管已报道硫链丝菌肽(Thiostrepton,TST)具有抗肿瘤作用,但其在胰腺癌中的作用及其潜在机制仍不清楚。本研究揭示了TST通过STAT3/GPX4信号通路诱导胰腺癌细胞铁死亡的作用机制。
研究结果:
实验结果表明,TST能显著抑制胰腺癌细胞系的活力与克隆形成能力,同时引发细胞内铁过载、活性氧(ROS)累积、丙二醛(MDA)水平升高以及谷胱甘肽过氧化物酶(GSH-PX)耗竭等铁死亡特征性改变。机制研究发现,STAT3可直接结合GPX4启动子区域并激活其转录,而TST通过调控STAT3显著抑制GPX4表达。动物实验进一步证实,TST能有效抑制皮下移植瘤的生长且表现出良好的生物安全性。该研究不仅阐明了TST抗胰腺癌的作用靶点,也为铁死亡相关肿瘤治疗策略提供了新的理论依据。
作者首先使用 JASPAR网站预测了STAT3在GPX4启动子上的潜在结合位点。随后通过 ChIP-qPCR实验来确认这些位点。ChIP实验的结果证实了STAT3可以直接与GPX4的启动子区域(P2)结合,而非其他预测的结合位点(P1、P3 和 P4)。
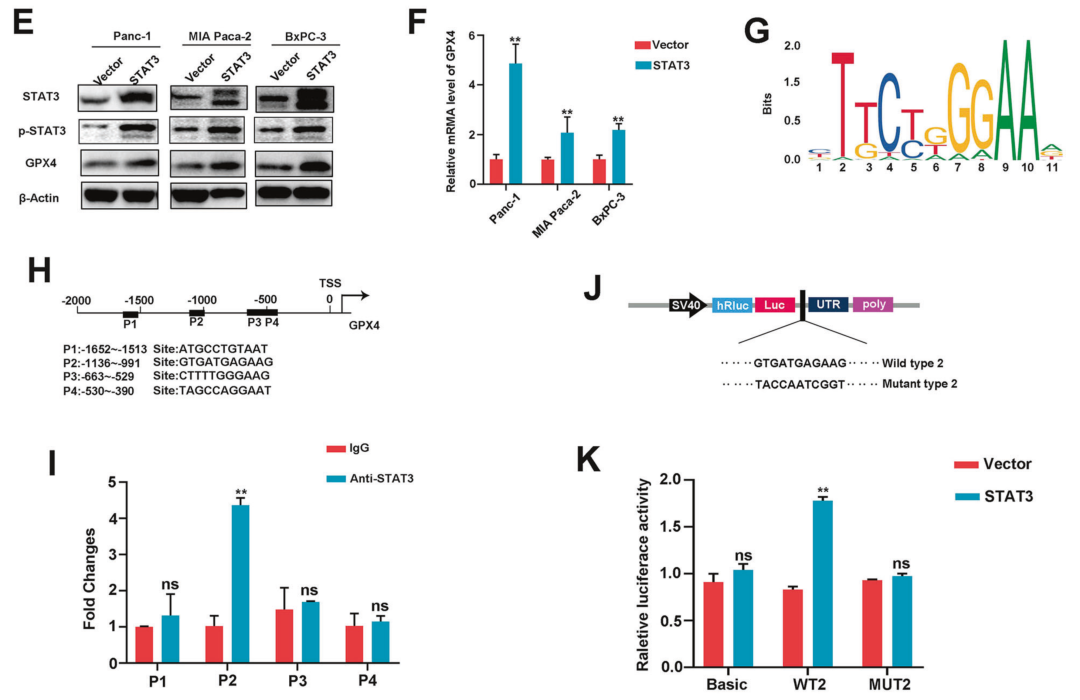
图 TST 激活STAT3–GPX4信号通路(Zhang et al., 2022)。
E-F:蛋白质印迹和 qRT-PCR分析,胰腺癌中STAT3过表达引起的GPX4表达变化。G-H:JASPAR 预测了一个保守的 STAT3 结合基序,并显示了GPX4启动子中潜在STAT3结合位点的示意图。I:ChIP分析细胞中GPX4启动子上STAT3的占有率。J:在GPX4启动子区含有STAT3结合位点(WT和MUT)的荧光素酶报告质粒的示意图。K:荧光素酶报告基因分析。
小医叨叨
作为表观遗传学研究的关键技术,ChIP-qPCR验证实验对操作规范与经验积累有着极高要求。伯远生物配备专业技术团队,累计为上千课题组提供ChIP富集实验服务,我们的技术团队不仅掌握超声破碎效率优化、抗体特异性验证等核心环节的标准化质控标准,更建立了覆盖植物、动物、微生物的跨物种实验体系。若您正面临染色质免疫共沉淀的验证难题,欢迎通过通过400-027-0273联系我们,期待与您的沟通交流!
参考文献
[1] Zhang W, Gong M, Zhang W, et al. Thiostrepton induces ferroptosis in pancreatic cancer cells through STAT3/GPX4 signalling[J]. Cell death & disease, 2022, 13(7): 630.
