基于深度学习的胸部 X 光图像肺炎分类系统(五)
改进版,前面训练出来的指标不尽人意,这里搞个改进版。重点说明各类优化措施。
同时 也进行了下列优化:
使用迁移学习:采用预训练的ResNet50模型作为特征提取器
优化数据处理:
合并训练集和验证集后重新划分(解决验证集过小问题)
增强数据预处理(更丰富的图像增强)
改进模型架构:
使用全局平均池化代替展平层
添加更多正则化
训练优化:
添加学习率调度器
增加模型检查点保存
调整早停策略
解决类别不平衡:
结合过采样和类别权重
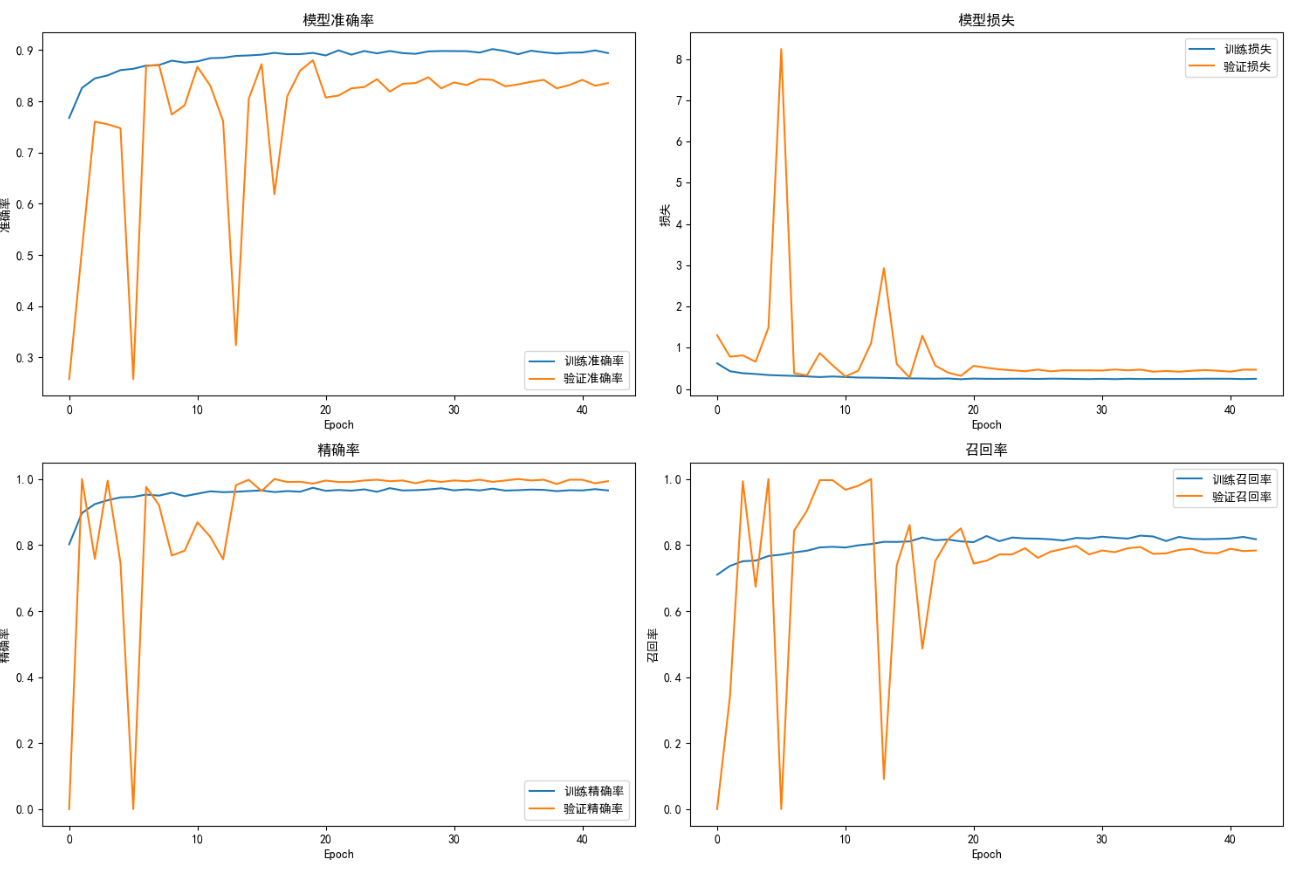
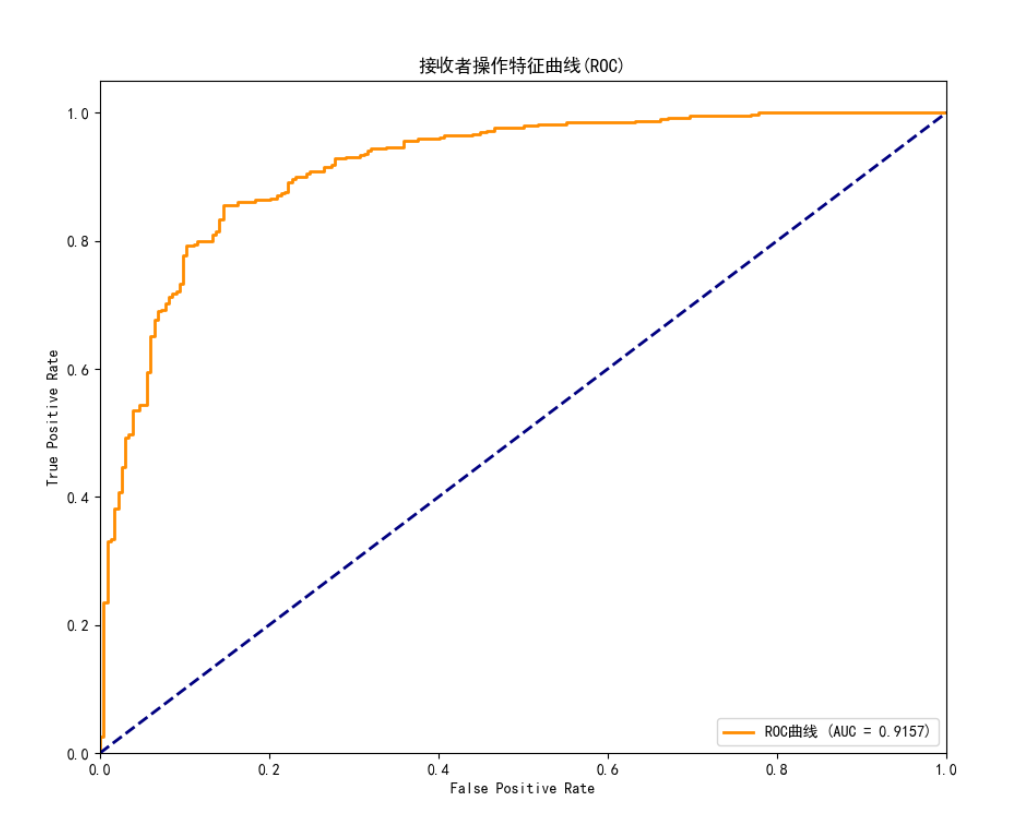
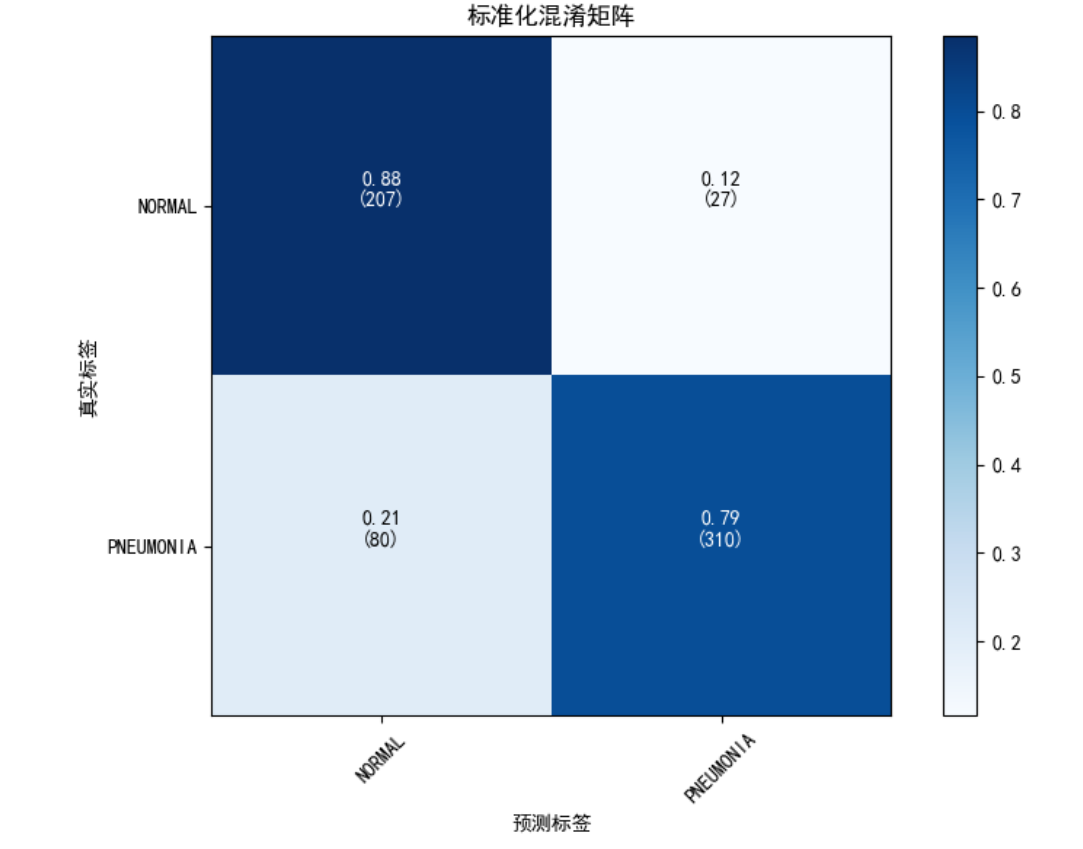
改进后运行效果如图:
medical_image_classification_fixed2.py :
import numpy as np
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.metrics import f1_score, roc_auc_score, roc_curve, confusion_matrix, classification_report
from imblearn.over_sampling import RandomOverSampler
import tensorflow as tf
from keras import layers
from keras import models
from keras.models import Sequential
from keras.preprocessing.image import ImageDataGenerator
import os
import shutil
from keras.callbacks import EarlyStopping, ModelCheckpoint, ReduceLROnPlateau
from keras.applications import ResNet50
from keras.optimizers import Adam
import warnings
import requests# 忽略警告
warnings.filterwarnings('ignore')plt.rcParams['font.sans-serif'] = ['SimHei'] # 使用 SimHei 字体
plt.rcParams['axes.unicode_minus'] = False # 解决负号显示问题
plt.rcParams['font.size'] = 10 # 设置全局字体大小def prepare_data_directories(base_dir):"""准备数据目录,合并训练集和验证集"""# 创建临时目录用于合并数据temp_dir = os.path.join(base_dir, 'temp_combined')combined_train_dir = os.path.join(temp_dir, 'train')os.makedirs(combined_train_dir, exist_ok=True)# 原始目录original_train_dir = os.path.join(base_dir, 'train')original_val_dir = os.path.join(base_dir, 'val')# 合并训练集和验证集for class_name in ['NORMAL', 'PNEUMONIA']:# 创建类别目录class_dir = os.path.join(combined_train_dir, class_name)os.makedirs(class_dir, exist_ok=True)# 复制原始训练集src_train = os.path.join(original_train_dir, class_name)for file in os.listdir(src_train):src_file = os.path.join(src_train, file)dst_file = os.path.join(class_dir, file)if not os.path.exists(dst_file):shutil.copy(src_file, dst_file)# 复制验证集src_val = os.path.join(original_val_dir, class_name)if os.path.exists(src_val):for file in os.listdir(src_val):src_file = os.path.join(src_val, file)dst_file = os.path.join(class_dir, file)if not os.path.exists(dst_file):shutil.copy(src_file, dst_file)return temp_dir, combined_train_dirdef load_data(base_dir, img_size=(224, 224), batch_size=32, validation_split=0.2):"""加载并预处理数据"""# 准备数据目录temp_dir, combined_train_dir = prepare_data_directories(base_dir)# 训练数据生成器(包含增强)train_datagen = ImageDataGenerator(rescale=1. / 255,rotation_range=15,width_shift_range=0.15,height_shift_range=0.15,shear_range=0.15,zoom_range=0.15,horizontal_flip=True,brightness_range=[0.8, 1.2],fill_mode='nearest',validation_split=validation_split)# 验证和测试数据生成器(只进行缩放)test_val_datagen = ImageDataGenerator(rescale=1. / 255)# 训练集train_generator = train_datagen.flow_from_directory(combined_train_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=True,subset='training')# 验证集val_generator = train_datagen.flow_from_directory(combined_train_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=False,subset='validation')# 测试集test_dir = os.path.join(base_dir, 'test')test_generator = test_val_datagen.flow_from_directory(test_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=False)return train_generator, val_generator, test_generator, temp_dirdef handle_imbalance(generator):"""处理样本不平衡问题"""# 获取所有数据X, y = [], []generator.reset()for _ in range(len(generator)):batch_x, batch_y = generator.next()X.append(batch_x)y.append(batch_y)X = np.concatenate(X)y = np.concatenate(y)# 打印原始分布print(f"原始样本分布: 正常={np.sum(y == 0)}, 肺炎={np.sum(y == 1)}")# 过采样X_flat = X.reshape(X.shape[0], -1)ros = RandomOverSampler(random_state=42)X_resampled, y_resampled = ros.fit_resample(X_flat, y)X_resampled = X_resampled.reshape(-1, *X.shape[1:])print(f"过采样后分布: 正常={np.sum(y_resampled == 0)}, 肺炎={np.sum(y_resampled == 1)}")return X_resampled, y_resampled, ydef download_resnet_weights():"""下载ResNet50权重文件"""weights_url = "https://github.com/keras-team/keras-applications/releases/download/resnet/resnet50_weights_tf_dim_ordering_tf_kernels_notop.h5"local_path = "models/resnet50_weights_tf_dim_ordering_tf_kernels_notop.h5"os.makedirs("models", exist_ok=True)if not os.path.exists(local_path):print("正在下载ResNet50权重文件...")try:response = requests.get(weights_url, stream=True)response.raise_for_status()with open(local_path, 'wb') as f:for chunk in response.iter_content(chunk_size=8192):f.write(chunk)print("权重文件下载完成")except Exception as e:print(f"下载失败: {e}")print("请手动下载权重文件并放在 models/ 目录下:")print("https://github.com/keras-team/keras-applications/releases/download/resnet/resnet50_weights_tf_dim_ordering_tf_kernels_notop.h5")return Nonereturn local_pathdef build_model(input_shape):"""构建基于ResNet50的迁移学习模型"""# 获取本地权重文件路径weights_path = download_resnet_weights()if weights_path is None:print("使用随机初始化的权重(性能会降低)")base_model = ResNet50(weights=None,include_top=False,input_shape=input_shape)else:print(f"使用本地权重文件: {weights_path}")base_model = ResNet50(weights=weights_path,include_top=False,input_shape=input_shape)# 冻结基础模型base_model.trainable = False# 创建新模型model = models.Sequential([base_model,layers.GlobalAveragePooling2D(),layers.Dense(512, activation='relu'),layers.BatchNormalization(),layers.Dropout(0.5),layers.Dense(256, activation='relu'),layers.BatchNormalization(),layers.Dropout(0.3),layers.Dense(1, activation='sigmoid')])# 编译模型optimizer = Adam(learning_rate=0.0001)model.compile(optimizer=optimizer,loss='binary_crossentropy',metrics=['accuracy',tf.keras.metrics.Precision(name='precision'),tf.keras.metrics.Recall(name='recall'),tf.keras.metrics.AUC(name='auc')])return modeldef plot_training_history(history):"""绘制训练历史图表"""plt.figure(figsize=(15, 10))# 准确率plt.subplot(2, 2, 1)plt.plot(history.history['accuracy'], label='训练准确率')plt.plot(history.history['val_accuracy'], label='验证准确率')plt.title('模型准确率')plt.ylabel('准确率')plt.xlabel('Epoch')plt.legend()# 损失plt.subplot(2, 2, 2)plt.plot(history.history['loss'], label='训练损失')plt.plot(history.history['val_loss'], label='验证损失')plt.title('模型损失')plt.ylabel('损失')plt.xlabel('Epoch')plt.legend()# 精确率plt.subplot(2, 2, 3)plt.plot(history.history['precision'], label='训练精确率')plt.plot(history.history['val_precision'], label='验证精确率')plt.title('精确率')plt.ylabel('精确率')plt.xlabel('Epoch')plt.legend()# 召回率plt.subplot(2, 2, 4)plt.plot(history.history['recall'], label='训练召回率')plt.plot(history.history['val_recall'], label='验证召回率')plt.title('召回率')plt.ylabel('召回率')plt.xlabel('Epoch')plt.legend()plt.tight_layout()plt.savefig('training_history_improved.png', dpi=300)plt.show()def plot_roc_curve(y_true, y_pred_prob):"""绘制ROC曲线"""fpr, tpr, _ = roc_curve(y_true, y_pred_prob)auc_score = roc_auc_score(y_true, y_pred_prob)plt.figure(figsize=(10, 8))plt.plot(fpr, tpr, color='darkorange', lw=2, label=f'ROC曲线 (AUC = {auc_score:.4f})')plt.plot([0, 1], [0, 1], color='navy', lw=2, linestyle='--')plt.xlim([0.0, 1.0])plt.ylim([0.0, 1.05])plt.xlabel('False Positive Rate')plt.ylabel('True Positive Rate')plt.title('接收者操作特征曲线(ROC)')plt.legend(loc="lower right")plt.savefig('roc_curve_improved.png', dpi=300)plt.show()return auc_scoredef plot_confusion_matrix(y_true, y_pred):"""绘制混淆矩阵"""cm = confusion_matrix(y_true, y_pred)cm_normalized = cm.astype('float') / cm.sum(axis=1)[:, np.newaxis]plt.figure(figsize=(8, 6))plt.imshow(cm_normalized, interpolation='nearest', cmap=plt.cm.Blues)plt.title('标准化混淆矩阵')plt.colorbar()classes = ['NORMAL', 'PNEUMONIA']tick_marks = np.arange(len(classes))plt.xticks(tick_marks, classes, rotation=45)plt.yticks(tick_marks, classes)fmt = '.2f'thresh = cm_normalized.max() / 2.for i in range(cm_normalized.shape[0]):for j in range(cm_normalized.shape[1]):plt.text(j, i, f"{cm_normalized[i, j]:.2f}\n({cm[i, j]})",horizontalalignment="center",color="white" if cm_normalized[i, j] > thresh else "black")plt.ylabel('真实标签')plt.xlabel('预测标签')plt.tight_layout()plt.savefig('confusion_matrix_improved.png', dpi=300)plt.show()# 主函数
def main():# 数据集路径base_dir = "chest_xray"# 加载数据img_size = (224, 224) # ResNet50的标准输入尺寸batch_size = 32validation_split = 0.15 # 验证集比例train_generator, val_generator, test_generator, temp_dir = load_data(base_dir, img_size, batch_size, validation_split)# 处理样本不平衡X_train, y_train_resampled, y_train_original = handle_imbalance(train_generator)# 计算类别权重n_normal = np.sum(y_train_original == 0)n_pneumonia = np.sum(y_train_original == 1)total = n_normal + n_pneumoniaweight_for_normal = (1 / n_normal) * (total / 2.0)weight_for_pneumonia = (1 / n_pneumonia) * (total / 2.0)class_weights = {0: weight_for_normal, 1: weight_for_pneumonia}print(f"类别权重: 正常={weight_for_normal:.2f}, 肺炎={weight_for_pneumonia:.2f}")# 构建模型model = build_model((*img_size, 3))model.summary()# 回调函数early_stopping = EarlyStopping(monitor='val_auc',patience=8,verbose=1,mode='max',restore_best_weights=True)reduce_lr = ReduceLROnPlateau(monitor='val_loss',factor=0.2,patience=3,min_lr=1e-7,verbose=1)model_checkpoint = ModelCheckpoint('best_pneumonia_model.h5',monitor='val_auc',save_best_only=True,mode='max',verbose=1)# 训练模型history = model.fit(X_train, y_train_resampled,epochs=50,batch_size=32,validation_data=val_generator,class_weight=class_weights,callbacks=[early_stopping, reduce_lr, model_checkpoint],verbose=1)# 加载最佳模型model.load_weights('best_pneumonia_model.h5')# 评估测试集test_generator.reset()test_steps = len(test_generator)test_results = model.evaluate(test_generator, steps=test_steps, verbose=1)print("\n测试集评估结果:")print(f"准确率: {test_results[1]:.4f}")print(f"精确率: {test_results[2]:.4f}")print(f"召回率: {test_results[3]:.4f}")print(f"AUC: {test_results[4]:.4f}")# 获取预测结果test_generator.reset()y_true = []y_pred_prob = []for i in range(test_steps):batch_x, batch_y = test_generator.next()y_true.extend(batch_y)batch_pred = model.predict(batch_x, verbose=0).ravel()y_pred_prob.extend(batch_pred)y_true = np.array(y_true)y_pred_prob = np.array(y_pred_prob)y_pred = (y_pred_prob > 0.5).astype(int)# 计算额外指标f1 = f1_score(y_true, y_pred)auc = roc_auc_score(y_true, y_pred_prob)print(f"\nF1-score: {f1:.4f}")print(f"AUC-ROC: {auc:.4f}")# 分类报告print("\n分类报告:")print(classification_report(y_true, y_pred, target_names=['NORMAL', 'PNEUMONIA']))# 混淆矩阵cm = confusion_matrix(y_true, y_pred)print("混淆矩阵:")print(cm)# 可视化plot_training_history(history)plot_roc_curve(y_true, y_pred_prob)plot_confusion_matrix(y_true, y_pred)# 清理临时目录shutil.rmtree(temp_dir)if __name__ == "__main__":main()
D:\ProgramData\anaconda3\envs\tf_env\python.exe D:\workspace_py\deeplean\medical_image_classification_fixed2.py
Found 4448 images belonging to 2 classes.
Found 784 images belonging to 2 classes.
Found 624 images belonging to 2 classes.
原始样本分布: 正常=1147, 肺炎=3301
过采样后分布: 正常=3301, 肺炎=3301
类别权重: 正常=1.94, 肺炎=0.67
使用本地权重文件: models/resnet50_weights_tf_dim_ordering_tf_kernels_notop.h5
2025-07-25 14:47:12.955043: I tensorflow/core/platform/cpu_feature_guard.cc:182] This TensorFlow binary is optimized to use available CPU instructions in performance-critical operations.
To enable the following instructions: SSE SSE2 SSE3 SSE4.1 SSE4.2 AVX AVX2 FMA, in other operations, rebuild TensorFlow with the appropriate compiler flags.
Model: "sequential"
_________________________________________________________________Layer (type) Output Shape Param #
=================================================================resnet50 (Functional) (None, 7, 7, 2048) 23587712 global_average_pooling2d ( (None, 2048) 0 GlobalAveragePooling2D) dense (Dense) (None, 512) 1049088 batch_normalization (Batch (None, 512) 2048 Normalization) dropout (Dropout) (None, 512) 0 dense_1 (Dense) (None, 256) 131328 batch_normalization_1 (Bat (None, 256) 1024 chNormalization) dropout_1 (Dropout) (None, 256) 0 dense_2 (Dense) (None, 1) 257 =================================================================
Total params: 24771457 (94.50 MB)
Trainable params: 1182209 (4.51 MB)
Non-trainable params: 23589248 (89.99 MB)
_________________________________________________________________
Epoch 1/50
207/207 [==============================] - ETA: 0s - loss: 0.6233 - accuracy: 0.7675 - precision: 0.8022 - recall: 0.7101 - auc: 0.8398
Epoch 1: val_auc improved from -inf to 0.87968, saving model to best_pneumonia_model.h5
207/207 [==============================] - 245s 1s/step - loss: 0.6233 - accuracy: 0.7675 - precision: 0.8022 - recall: 0.7101 - auc: 0.8398 - val_loss: 1.3033 - val_accuracy: 0.2577 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.8797 - lr: 1.0000e-04
Epoch 2/50
207/207 [==============================] - ETA: 0s - loss: 0.4313 - accuracy: 0.8261 - precision: 0.8971 - recall: 0.7367 - auc: 0.9035
Epoch 2: val_auc improved from 0.87968 to 0.92927, saving model to best_pneumonia_model.h5
207/207 [==============================] - 257s 1s/step - loss: 0.4313 - accuracy: 0.8261 - precision: 0.8971 - recall: 0.7367 - auc: 0.9035 - val_loss: 0.7843 - val_accuracy: 0.5115 - val_precision: 1.0000 - val_recall: 0.3419 - val_auc: 0.9293 - lr: 1.0000e-04
Epoch 3/50
207/207 [==============================] - ETA: 0s - loss: 0.3833 - accuracy: 0.8446 - precision: 0.9240 - recall: 0.7510 - auc: 0.9174
Epoch 3: val_auc did not improve from 0.92927
207/207 [==============================] - 242s 1s/step - loss: 0.3833 - accuracy: 0.8446 - precision: 0.9240 - recall: 0.7510 - auc: 0.9174 - val_loss: 0.8157 - val_accuracy: 0.7602 - val_precision: 0.7585 - val_recall: 0.9931 - val_auc: 0.8778 - lr: 1.0000e-04
Epoch 4/50
207/207 [==============================] - ETA: 0s - loss: 0.3648 - accuracy: 0.8505 - precision: 0.9356 - recall: 0.7528 - auc: 0.9254
Epoch 4: val_auc improved from 0.92927 to 0.94531, saving model to best_pneumonia_model.h5
207/207 [==============================] - 242s 1s/step - loss: 0.3648 - accuracy: 0.8505 - precision: 0.9356 - recall: 0.7528 - auc: 0.9254 - val_loss: 0.6605 - val_accuracy: 0.7551 - val_precision: 0.9949 - val_recall: 0.6735 - val_auc: 0.9453 - lr: 1.0000e-04
Epoch 5/50
207/207 [==============================] - ETA: 0s - loss: 0.3387 - accuracy: 0.8608 - precision: 0.9444 - recall: 0.7667 - auc: 0.9325
Epoch 5: val_auc did not improve from 0.94531
207/207 [==============================] - 239s 1s/step - loss: 0.3387 - accuracy: 0.8608 - precision: 0.9444 - recall: 0.7667 - auc: 0.9325 - val_loss: 1.4892 - val_accuracy: 0.7474 - val_precision: 0.7462 - val_recall: 1.0000 - val_auc: 0.7024 - lr: 1.0000e-04
Epoch 6/50
207/207 [==============================] - ETA: 0s - loss: 0.3275 - accuracy: 0.8634 - precision: 0.9457 - recall: 0.7710 - auc: 0.9403
Epoch 6: val_auc did not improve from 0.94531
207/207 [==============================] - 240s 1s/step - loss: 0.3275 - accuracy: 0.8634 - precision: 0.9457 - recall: 0.7710 - auc: 0.9403 - val_loss: 8.2440 - val_accuracy: 0.2577 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.5095 - lr: 1.0000e-04
Epoch 7/50
207/207 [==============================] - ETA: 0s - loss: 0.3184 - accuracy: 0.8696 - precision: 0.9529 - recall: 0.7776 - auc: 0.9406
Epoch 7: val_auc improved from 0.94531 to 0.94572, saving model to best_pneumonia_model.h5
207/207 [==============================] - 240s 1s/step - loss: 0.3184 - accuracy: 0.8696 - precision: 0.9529 - recall: 0.7776 - auc: 0.9406 - val_loss: 0.3840 - val_accuracy: 0.8686 - val_precision: 0.9761 - val_recall: 0.8436 - val_auc: 0.9457 - lr: 1.0000e-04
Epoch 8/50
207/207 [==============================] - ETA: 0s - loss: 0.3061 - accuracy: 0.8706 - precision: 0.9497 - recall: 0.7828 - auc: 0.9458
Epoch 8: val_auc did not improve from 0.94572
207/207 [==============================] - 238s 1s/step - loss: 0.3061 - accuracy: 0.8706 - precision: 0.9497 - recall: 0.7828 - auc: 0.9458 - val_loss: 0.3327 - val_accuracy: 0.8712 - val_precision: 0.9212 - val_recall: 0.9038 - val_auc: 0.9368 - lr: 1.0000e-04
Epoch 9/50
207/207 [==============================] - ETA: 0s - loss: 0.2896 - accuracy: 0.8793 - precision: 0.9586 - recall: 0.7928 - auc: 0.9498
Epoch 9: val_auc did not improve from 0.94572
207/207 [==============================] - 236s 1s/step - loss: 0.2896 - accuracy: 0.8793 - precision: 0.9586 - recall: 0.7928 - auc: 0.9498 - val_loss: 0.8718 - val_accuracy: 0.7742 - val_precision: 0.7682 - val_recall: 0.9966 - val_auc: 0.8534 - lr: 1.0000e-04
Epoch 10/50
207/207 [==============================] - ETA: 0s - loss: 0.3042 - accuracy: 0.8755 - precision: 0.9480 - recall: 0.7946 - auc: 0.9467
Epoch 10: val_auc did not improve from 0.94572
207/207 [==============================] - 237s 1s/step - loss: 0.3042 - accuracy: 0.8755 - precision: 0.9480 - recall: 0.7946 - auc: 0.9467 - val_loss: 0.5748 - val_accuracy: 0.7921 - val_precision: 0.7827 - val_recall: 0.9966 - val_auc: 0.9386 - lr: 1.0000e-04
Epoch 11/50
207/207 [==============================] - ETA: 0s - loss: 0.2909 - accuracy: 0.8778 - precision: 0.9554 - recall: 0.7925 - auc: 0.9501
Epoch 11: val_auc improved from 0.94572 to 0.95169, saving model to best_pneumonia_model.h5
207/207 [==============================] - 237s 1s/step - loss: 0.2909 - accuracy: 0.8778 - precision: 0.9554 - recall: 0.7925 - auc: 0.9501 - val_loss: 0.3041 - val_accuracy: 0.8673 - val_precision: 0.8688 - val_recall: 0.9674 - val_auc: 0.9517 - lr: 1.0000e-04
Epoch 12/50
207/207 [==============================] - ETA: 0s - loss: 0.2779 - accuracy: 0.8841 - precision: 0.9628 - recall: 0.7992 - auc: 0.9537
Epoch 12: val_auc did not improve from 0.95169
207/207 [==============================] - 236s 1s/step - loss: 0.2779 - accuracy: 0.8841 - precision: 0.9628 - recall: 0.7992 - auc: 0.9537 - val_loss: 0.4429 - val_accuracy: 0.8304 - val_precision: 0.8249 - val_recall: 0.9794 - val_auc: 0.9420 - lr: 1.0000e-04
Epoch 13/50
207/207 [==============================] - ETA: 0s - loss: 0.2761 - accuracy: 0.8849 - precision: 0.9602 - recall: 0.8031 - auc: 0.9530
Epoch 13: val_auc did not improve from 0.95169
207/207 [==============================] - 236s 1s/step - loss: 0.2761 - accuracy: 0.8849 - precision: 0.9602 - recall: 0.8031 - auc: 0.9530 - val_loss: 1.1105 - val_accuracy: 0.7615 - val_precision: 0.7568 - val_recall: 1.0000 - val_auc: 0.8080 - lr: 1.0000e-04
Epoch 14/50
207/207 [==============================] - ETA: 0s - loss: 0.2710 - accuracy: 0.8885 - precision: 0.9612 - recall: 0.8098 - auc: 0.9555
Epoch 14: ReduceLROnPlateau reducing learning rate to 1.9999999494757503e-05.Epoch 14: val_auc did not improve from 0.95169
207/207 [==============================] - 236s 1s/step - loss: 0.2710 - accuracy: 0.8885 - precision: 0.9612 - recall: 0.8098 - auc: 0.9555 - val_loss: 2.9286 - val_accuracy: 0.3240 - val_precision: 0.9815 - val_recall: 0.0911 - val_auc: 0.8606 - lr: 1.0000e-04
Epoch 15/50
207/207 [==============================] - ETA: 0s - loss: 0.2634 - accuracy: 0.8894 - precision: 0.9636 - recall: 0.8095 - auc: 0.9567
Epoch 15: val_auc improved from 0.95169 to 0.95699, saving model to best_pneumonia_model.h5
207/207 [==============================] - 237s 1s/step - loss: 0.2634 - accuracy: 0.8894 - precision: 0.9636 - recall: 0.8095 - auc: 0.9567 - val_loss: 0.6077 - val_accuracy: 0.8048 - val_precision: 0.9977 - val_recall: 0.7388 - val_auc: 0.9570 - lr: 2.0000e-05
Epoch 16/50
207/207 [==============================] - ETA: 0s - loss: 0.2576 - accuracy: 0.8909 - precision: 0.9654 - recall: 0.8110 - auc: 0.9600
Epoch 16: val_auc did not improve from 0.95699
207/207 [==============================] - 236s 1s/step - loss: 0.2576 - accuracy: 0.8909 - precision: 0.9654 - recall: 0.8110 - auc: 0.9600 - val_loss: 0.2776 - val_accuracy: 0.8724 - val_precision: 0.9635 - val_recall: 0.8608 - val_auc: 0.9566 - lr: 2.0000e-05
Epoch 17/50
207/207 [==============================] - ETA: 0s - loss: 0.2553 - accuracy: 0.8944 - precision: 0.9604 - recall: 0.8228 - auc: 0.9601
Epoch 17: val_auc did not improve from 0.95699
207/207 [==============================] - 238s 1s/step - loss: 0.2553 - accuracy: 0.8944 - precision: 0.9604 - recall: 0.8228 - auc: 0.9601 - val_loss: 1.2915 - val_accuracy: 0.6186 - val_precision: 1.0000 - val_recall: 0.4863 - val_auc: 0.9440 - lr: 2.0000e-05
Epoch 18/50
207/207 [==============================] - ETA: 0s - loss: 0.2505 - accuracy: 0.8919 - precision: 0.9635 - recall: 0.8146 - auc: 0.9608
Epoch 18: val_auc did not improve from 0.95699
207/207 [==============================] - 236s 1s/step - loss: 0.2505 - accuracy: 0.8919 - precision: 0.9635 - recall: 0.8146 - auc: 0.9608 - val_loss: 0.5704 - val_accuracy: 0.8099 - val_precision: 0.9909 - val_recall: 0.7509 - val_auc: 0.9460 - lr: 2.0000e-05
Epoch 19/50
207/207 [==============================] - ETA: 0s - loss: 0.2557 - accuracy: 0.8920 - precision: 0.9615 - recall: 0.8167 - auc: 0.9593
Epoch 19: ReduceLROnPlateau reducing learning rate to 3.999999898951501e-06.Epoch 19: val_auc did not improve from 0.95699
207/207 [==============================] - 236s 1s/step - loss: 0.2557 - accuracy: 0.8920 - precision: 0.9615 - recall: 0.8167 - auc: 0.9593 - val_loss: 0.3977 - val_accuracy: 0.8597 - val_precision: 0.9917 - val_recall: 0.8179 - val_auc: 0.9558 - lr: 2.0000e-05
Epoch 20/50
207/207 [==============================] - ETA: 0s - loss: 0.2383 - accuracy: 0.8944 - precision: 0.9735 - recall: 0.8110 - auc: 0.9643
Epoch 20: val_auc improved from 0.95699 to 0.96287, saving model to best_pneumonia_model.h5
207/207 [==============================] - 236s 1s/step - loss: 0.2383 - accuracy: 0.8944 - precision: 0.9735 - recall: 0.8110 - auc: 0.9643 - val_loss: 0.3187 - val_accuracy: 0.8801 - val_precision: 0.9861 - val_recall: 0.8505 - val_auc: 0.9629 - lr: 4.0000e-06
Epoch 21/50
207/207 [==============================] - ETA: 0s - loss: 0.2539 - accuracy: 0.8894 - precision: 0.9642 - recall: 0.8088 - auc: 0.9606
Epoch 21: val_auc did not improve from 0.96287
207/207 [==============================] - 236s 1s/step - loss: 0.2539 - accuracy: 0.8894 - precision: 0.9642 - recall: 0.8088 - auc: 0.9606 - val_loss: 0.5630 - val_accuracy: 0.8074 - val_precision: 0.9954 - val_recall: 0.7440 - val_auc: 0.9522 - lr: 4.0000e-06
Epoch 22/50
207/207 [==============================] - ETA: 0s - loss: 0.2480 - accuracy: 0.8994 - precision: 0.9667 - recall: 0.8273 - auc: 0.9616
Epoch 22: ReduceLROnPlateau reducing learning rate to 7.999999979801942e-07.Epoch 22: val_auc did not improve from 0.96287
207/207 [==============================] - 236s 1s/step - loss: 0.2480 - accuracy: 0.8994 - precision: 0.9667 - recall: 0.8273 - auc: 0.9616 - val_loss: 0.5155 - val_accuracy: 0.8112 - val_precision: 0.9910 - val_recall: 0.7526 - val_auc: 0.9588 - lr: 4.0000e-06
Epoch 23/50
207/207 [==============================] - ETA: 0s - loss: 0.2474 - accuracy: 0.8909 - precision: 0.9647 - recall: 0.8116 - auc: 0.9634
Epoch 23: val_auc improved from 0.96287 to 0.96317, saving model to best_pneumonia_model.h5
207/207 [==============================] - 238s 1s/step - loss: 0.2474 - accuracy: 0.8909 - precision: 0.9647 - recall: 0.8116 - auc: 0.9634 - val_loss: 0.4768 - val_accuracy: 0.8253 - val_precision: 0.9912 - val_recall: 0.7715 - val_auc: 0.9632 - lr: 8.0000e-07
Epoch 24/50
207/207 [==============================] - ETA: 0s - loss: 0.2496 - accuracy: 0.8981 - precision: 0.9686 - recall: 0.8228 - auc: 0.9612
Epoch 24: val_auc improved from 0.96317 to 0.96399, saving model to best_pneumonia_model.h5
207/207 [==============================] - 236s 1s/step - loss: 0.2496 - accuracy: 0.8981 - precision: 0.9686 - recall: 0.8228 - auc: 0.9612 - val_loss: 0.4538 - val_accuracy: 0.8278 - val_precision: 0.9956 - val_recall: 0.7715 - val_auc: 0.9640 - lr: 8.0000e-07
Epoch 25/50
207/207 [==============================] - ETA: 0s - loss: 0.2504 - accuracy: 0.8935 - precision: 0.9613 - recall: 0.8201 - auc: 0.9618
Epoch 25: ReduceLROnPlateau reducing learning rate to 1.600000018697756e-07.Epoch 25: val_auc did not improve from 0.96399
207/207 [==============================] - 237s 1s/step - loss: 0.2504 - accuracy: 0.8935 - precision: 0.9613 - recall: 0.8201 - auc: 0.9618 - val_loss: 0.4323 - val_accuracy: 0.8431 - val_precision: 0.9978 - val_recall: 0.7904 - val_auc: 0.9596 - lr: 8.0000e-07
Epoch 26/50
207/207 [==============================] - ETA: 0s - loss: 0.2448 - accuracy: 0.8981 - precision: 0.9723 - recall: 0.8194 - auc: 0.9617
Epoch 26: val_auc did not improve from 0.96399
207/207 [==============================] - 236s 1s/step - loss: 0.2448 - accuracy: 0.8981 - precision: 0.9723 - recall: 0.8194 - auc: 0.9617 - val_loss: 0.4714 - val_accuracy: 0.8189 - val_precision: 0.9933 - val_recall: 0.7612 - val_auc: 0.9618 - lr: 1.6000e-07
Epoch 27/50
207/207 [==============================] - ETA: 0s - loss: 0.2514 - accuracy: 0.8940 - precision: 0.9653 - recall: 0.8173 - auc: 0.9611
Epoch 27: val_auc improved from 0.96399 to 0.96583, saving model to best_pneumonia_model.h5
207/207 [==============================] - 236s 1s/step - loss: 0.2514 - accuracy: 0.8940 - precision: 0.9653 - recall: 0.8173 - auc: 0.9611 - val_loss: 0.4280 - val_accuracy: 0.8342 - val_precision: 0.9956 - val_recall: 0.7801 - val_auc: 0.9658 - lr: 1.6000e-07
Epoch 28/50
207/207 [==============================] - ETA: 0s - loss: 0.2496 - accuracy: 0.8926 - precision: 0.9662 - recall: 0.8137 - auc: 0.9627
Epoch 28: ReduceLROnPlateau reducing learning rate to 1e-07.Epoch 28: val_auc did not improve from 0.96583
207/207 [==============================] - 235s 1s/step - loss: 0.2496 - accuracy: 0.8926 - precision: 0.9662 - recall: 0.8137 - auc: 0.9627 - val_loss: 0.4548 - val_accuracy: 0.8355 - val_precision: 0.9871 - val_recall: 0.7887 - val_auc: 0.9576 - lr: 1.6000e-07
Epoch 29/50
207/207 [==============================] - ETA: 0s - loss: 0.2448 - accuracy: 0.8973 - precision: 0.9682 - recall: 0.8216 - auc: 0.9635
Epoch 29: val_auc did not improve from 0.96583
207/207 [==============================] - 236s 1s/step - loss: 0.2448 - accuracy: 0.8973 - precision: 0.9682 - recall: 0.8216 - auc: 0.9635 - val_loss: 0.4514 - val_accuracy: 0.8469 - val_precision: 0.9957 - val_recall: 0.7973 - val_auc: 0.9533 - lr: 1.0000e-07
Epoch 30/50
207/207 [==============================] - ETA: 0s - loss: 0.2409 - accuracy: 0.8981 - precision: 0.9720 - recall: 0.8198 - auc: 0.9636
Epoch 30: val_auc did not improve from 0.96583
207/207 [==============================] - 236s 1s/step - loss: 0.2409 - accuracy: 0.8981 - precision: 0.9720 - recall: 0.8198 - auc: 0.9636 - val_loss: 0.4526 - val_accuracy: 0.8253 - val_precision: 0.9912 - val_recall: 0.7715 - val_auc: 0.9612 - lr: 1.0000e-07
Epoch 31/50
207/207 [==============================] - ETA: 0s - loss: 0.2476 - accuracy: 0.8979 - precision: 0.9656 - recall: 0.8252 - auc: 0.9615
Epoch 31: val_auc did not improve from 0.96583
207/207 [==============================] - 236s 1s/step - loss: 0.2476 - accuracy: 0.8979 - precision: 0.9656 - recall: 0.8252 - auc: 0.9615 - val_loss: 0.4479 - val_accuracy: 0.8367 - val_precision: 0.9956 - val_recall: 0.7835 - val_auc: 0.9585 - lr: 1.0000e-07
Epoch 32/50
207/207 [==============================] - ETA: 0s - loss: 0.2393 - accuracy: 0.8978 - precision: 0.9686 - recall: 0.8222 - auc: 0.9645
Epoch 32: val_auc did not improve from 0.96583
207/207 [==============================] - 236s 1s/step - loss: 0.2393 - accuracy: 0.8978 - precision: 0.9686 - recall: 0.8222 - auc: 0.9645 - val_loss: 0.4738 - val_accuracy: 0.8316 - val_precision: 0.9934 - val_recall: 0.7784 - val_auc: 0.9583 - lr: 1.0000e-07
Epoch 33/50
207/207 [==============================] - ETA: 0s - loss: 0.2492 - accuracy: 0.8950 - precision: 0.9654 - recall: 0.8194 - auc: 0.9614
Epoch 33: val_auc did not improve from 0.96583
207/207 [==============================] - 236s 1s/step - loss: 0.2492 - accuracy: 0.8950 - precision: 0.9654 - recall: 0.8194 - auc: 0.9614 - val_loss: 0.4522 - val_accuracy: 0.8431 - val_precision: 0.9978 - val_recall: 0.7904 - val_auc: 0.9589 - lr: 1.0000e-07
Epoch 34/50
207/207 [==============================] - ETA: 0s - loss: 0.2429 - accuracy: 0.9018 - precision: 0.9709 - recall: 0.8285 - auc: 0.9618
Epoch 34: val_auc did not improve from 0.96583
207/207 [==============================] - 239s 1s/step - loss: 0.2429 - accuracy: 0.9018 - precision: 0.9709 - recall: 0.8285 - auc: 0.9618 - val_loss: 0.4706 - val_accuracy: 0.8418 - val_precision: 0.9914 - val_recall: 0.7938 - val_auc: 0.9541 - lr: 1.0000e-07
Epoch 35/50
207/207 [==============================] - ETA: 0s - loss: 0.2455 - accuracy: 0.8979 - precision: 0.9650 - recall: 0.8258 - auc: 0.9627
Epoch 35: val_auc improved from 0.96583 to 0.97260, saving model to best_pneumonia_model.h5
207/207 [==============================] - 237s 1s/step - loss: 0.2455 - accuracy: 0.8979 - precision: 0.9650 - recall: 0.8258 - auc: 0.9627 - val_loss: 0.4193 - val_accuracy: 0.8291 - val_precision: 0.9956 - val_recall: 0.7732 - val_auc: 0.9726 - lr: 1.0000e-07
Epoch 36/50
207/207 [==============================] - ETA: 0s - loss: 0.2442 - accuracy: 0.8919 - precision: 0.9661 - recall: 0.8122 - auc: 0.9631
Epoch 36: val_auc did not improve from 0.97260
207/207 [==============================] - 234s 1s/step - loss: 0.2442 - accuracy: 0.8919 - precision: 0.9661 - recall: 0.8122 - auc: 0.9631 - val_loss: 0.4375 - val_accuracy: 0.8329 - val_precision: 1.0000 - val_recall: 0.7749 - val_auc: 0.9634 - lr: 1.0000e-07
Epoch 37/50
207/207 [==============================] - ETA: 0s - loss: 0.2447 - accuracy: 0.8987 - precision: 0.9680 - recall: 0.8246 - auc: 0.9628
Epoch 37: val_auc did not improve from 0.97260
207/207 [==============================] - 235s 1s/step - loss: 0.2447 - accuracy: 0.8987 - precision: 0.9680 - recall: 0.8246 - auc: 0.9628 - val_loss: 0.4201 - val_accuracy: 0.8380 - val_precision: 0.9956 - val_recall: 0.7852 - val_auc: 0.9673 - lr: 1.0000e-07
Epoch 38/50
207/207 [==============================] - ETA: 0s - loss: 0.2460 - accuracy: 0.8955 - precision: 0.9671 - recall: 0.8188 - auc: 0.9621
Epoch 38: val_auc did not improve from 0.97260
207/207 [==============================] - 236s 1s/step - loss: 0.2460 - accuracy: 0.8955 - precision: 0.9671 - recall: 0.8188 - auc: 0.9621 - val_loss: 0.4425 - val_accuracy: 0.8418 - val_precision: 0.9978 - val_recall: 0.7887 - val_auc: 0.9652 - lr: 1.0000e-07
Epoch 39/50
207/207 [==============================] - ETA: 0s - loss: 0.2503 - accuracy: 0.8932 - precision: 0.9632 - recall: 0.8176 - auc: 0.9622
Epoch 39: val_auc did not improve from 0.97260
207/207 [==============================] - 234s 1s/step - loss: 0.2503 - accuracy: 0.8932 - precision: 0.9632 - recall: 0.8176 - auc: 0.9622 - val_loss: 0.4590 - val_accuracy: 0.8253 - val_precision: 0.9847 - val_recall: 0.7766 - val_auc: 0.9571 - lr: 1.0000e-07
Epoch 40/50
207/207 [==============================] - ETA: 0s - loss: 0.2499 - accuracy: 0.8949 - precision: 0.9660 - recall: 0.8185 - auc: 0.9611
Epoch 40: val_auc did not improve from 0.97260
207/207 [==============================] - 235s 1s/step - loss: 0.2499 - accuracy: 0.8949 - precision: 0.9660 - recall: 0.8185 - auc: 0.9611 - val_loss: 0.4443 - val_accuracy: 0.8316 - val_precision: 0.9978 - val_recall: 0.7749 - val_auc: 0.9639 - lr: 1.0000e-07
Epoch 41/50
207/207 [==============================] - ETA: 0s - loss: 0.2489 - accuracy: 0.8952 - precision: 0.9654 - recall: 0.8198 - auc: 0.9612
Epoch 41: val_auc did not improve from 0.97260
207/207 [==============================] - 240s 1s/step - loss: 0.2489 - accuracy: 0.8952 - precision: 0.9654 - recall: 0.8198 - auc: 0.9612 - val_loss: 0.4213 - val_accuracy: 0.8418 - val_precision: 0.9978 - val_recall: 0.7887 - val_auc: 0.9677 - lr: 1.0000e-07
Epoch 42/50
207/207 [==============================] - ETA: 0s - loss: 0.2420 - accuracy: 0.8993 - precision: 0.9694 - recall: 0.8246 - auc: 0.9637
Epoch 42: val_auc did not improve from 0.97260
207/207 [==============================] - 234s 1s/step - loss: 0.2420 - accuracy: 0.8993 - precision: 0.9694 - recall: 0.8246 - auc: 0.9637 - val_loss: 0.4687 - val_accuracy: 0.8304 - val_precision: 0.9870 - val_recall: 0.7818 - val_auc: 0.9554 - lr: 1.0000e-07
Epoch 43/50
207/207 [==============================] - ETA: 0s - loss: 0.2483 - accuracy: 0.8941 - precision: 0.9653 - recall: 0.8176 - auc: 0.9614Restoring model weights from the end of the best epoch: 35.Epoch 43: val_auc did not improve from 0.97260
207/207 [==============================] - 234s 1s/step - loss: 0.2483 - accuracy: 0.8941 - precision: 0.9653 - recall: 0.8176 - auc: 0.9614 - val_loss: 0.4686 - val_accuracy: 0.8355 - val_precision: 0.9935 - val_recall: 0.7835 - val_auc: 0.9582 - lr: 1.0000e-07
Epoch 43: early stopping
20/20 [==============================] - 20s 998ms/step - loss: 0.4608 - accuracy: 0.8285 - precision: 0.9199 - recall: 0.7949 - auc: 0.9151测试集评估结果:
准确率: 0.8285
精确率: 0.9199
召回率: 0.7949
AUC: 0.9151F1-score: 0.8528
AUC-ROC: 0.9157分类报告:precision recall f1-score supportNORMAL 0.72 0.88 0.79 234PNEUMONIA 0.92 0.79 0.85 390accuracy 0.83 624macro avg 0.82 0.84 0.82 624
weighted avg 0.85 0.83 0.83 624混淆矩阵:
[[207 27][ 80 310]]Process finished with exit code 0
肺炎检测 AI 程序
这段代码是一个肺炎检测的 AI 程序,简单说就是用电脑自动看胸部 X 光片,判断是正常还是有肺炎。整个过程就像教电脑 “看图识病”:
先说说这个程序的目的
医生看胸部 X 光片能判断有没有肺炎,但人可能会累、会出错。这个程序想让电脑学这件事 —— 给它一堆标好 “正常” 或 “肺炎” 的 X 光片,让它学规律,之后就能自动判断新的 X 光片了。
分步骤解释代码在做什么
1. 准备数据:把图片 “整理好”
- prepare_data_directories 函数:
原始数据分散在 “训练集”“验证集” 两个文件夹里,这一步把它们合并到一个临时文件夹,方便统一处理。就像把散落在不同抽屉的文件放进一个盒子里。
- load_data 函数:
把图片变成电脑能看懂的格式(比如缩放成 224×224 大小,把颜色数值缩放到 0-1 之间)。同时还会 “折腾” 图片(比如旋转、轻微缩放),让电脑见更多样的图片,学得更扎实(这叫 “数据增强”)。
最后把数据分成 3 份:给电脑学的 “训练集”、学中间检查效果的 “验证集”、最后考试的 “测试集”。
2. 处理 “数据不平衡”:让电脑公平学习
- handle_imbalance 函数:
假设数据里有 1000 张肺炎图片,但只有 200 张正常图片。电脑可能会偷懒,大部分时候猜 “肺炎”,正确率也会高,但这样不好。
这一步会复制正常图片,让两种图片数量差不多(比如都变成 1000 张),保证电脑对两种情况都认真学。
3. 建模型:搭一个 “会学习的程序”
- download_resnet_weights 函数:
直接从零开始教电脑看图片很难,所以借用一个别人已经训练好的 “看图高手”(ResNet50 模型)的经验(叫 “权重文件”)。这就像学画画时先模仿大师的作品,不用从零开始。
- build_model 函数:
在 “看图高手” 的基础上,加几层简单的 “判断层”,让它专门学 “肺炎和正常的区别”。就像给通用的相机加一个 “肺炎模式”,专门优化这个任务。
4. 训练模型:让电脑 “反复练习”
- 用准备好的训练集让模型学,学的时候用 “验证集” 随时检查效果。
- 加了几个 “助手”(回调函数):
- 早停(early_stopping):如果学了很久没进步,就停下,避免白费劲;
- 降学习率(reduce_lr):如果学不动了,就放慢节奏慢慢学;
- 存最好模型(model_checkpoint):把学得最好的时候的状态存下来。
5. 考试和展示:看电脑学得多好
- 用 “测试集” 给模型考试,算 “正确率”(猜对的比例)、“精确率”(说有肺炎时真有的比例)、“召回率”(所有肺炎都能查出来的比例)等。
- 画图表展示:
- 训练过程图:看学习过程中正确率是不是越来越高,错误是不是越来越少;
- ROC 曲线:看模型区分正常和肺炎的能力强不强;
- 混淆矩阵:具体看多少张正常被错判成肺炎,多少肺炎被漏判。
- 整理图片数据,让电脑方便学;
- 调整数据,让电脑公平学两种情况;
- 借用现成的 “看图经验”,搭一个专门判断肺炎的程序;
- 让程序反复学习,随时调整避免白费力气;
- 考试并画图展示,看看程序学得多好。
最后,这个程序就能帮医生初步判断胸部 X 光片了,提高效率
优化数据处理:
合并训练集和验证集后重新划分(解决验证集过小问题)
增强数据预处理(更丰富的图像增强)
合并训练集和验证集重新划分
在代码中,load_data函数通过两个关键步骤体现了 “合并训练集和验证集后重新划分”,从而解决原始验证集可能过小的问题:
第一步:先合并原始训练集和验证集
在load_data函数开头,调用了prepare_data_directories函数:
temp_dir, combined_train_dir = prepare_data_directories(base_dir) |
这个函数的作用是:把原始的train(训练集)和val(验证集)文件夹里的所有图片,合并到一个新的临时文件夹temp_combined/train中。
比如原来train有 3000 张图、val有 500 张图,合并后就有 3500 张图放在一起。
第二步:用新的比例重新划分训练集和验证集
合并后,通过ImageDataGenerator的validation_split参数,按自定义比例重新划分:
# 定义训练数据生成器时,设置验证集比例(比如validation_split=0.15) train_datagen = ImageDataGenerator( ..., validation_split=validation_split # 这里的validation_split就是重新划分的比例 ) # 从合并后的数据集里,按比例拆分出“新训练集” train_generator = train_datagen.flow_from_directory( combined_train_dir, ..., subset='training' # 取大部分(比如85%)作为新训练集 ) # 从合并后的数据集里,按比例拆分出“新验证集” val_generator = train_datagen.flow_from_directory( combined_train_dir, ..., subset='validation' # 取小部分(比如15%)作为新验证集 ) |
为什么能解决 “验证集过小” 的问题?
假设原始数据中,val(验证集)只有很少的图片(比如 50 张),不足以准确评估模型。
合并后,所有图片(比如 3500 张)放在一起,再按validation_split=0.15划分,新验证集就有 3500×15%≈525 张,数量更多,评估结果更可靠。
简单说,就是 “把两杯水倒进一个大杯子,再按需要的比例重新分成两杯”,避免了原来小杯子(原始验证集)容量不够的问题。
X光片数据增强
load_data 的时候:
在 train_datagen = ImageDataGenerator(...) 里,这些参数属于增强数据预处理(让图片更多样化的操作):
- rotation_range=15
随机旋转图片(范围 0-15 度),比如把 X 光片轻微转个角度,模拟不同拍摄时的倾斜情况。
- width_shift_range=0.15
随机左右平移图片(最多移图片宽度的 15%),比如把肺部位置稍微左移或右移一点。
- height_shift_range=0.15
随机上下平移图片(最多移图片高度的 15%),类似左右平移,增加位置多样性。
- shear_range=0.15
随机轻微 “扭曲” 图片(剪切变换),比如让图片边缘稍微倾斜,模拟拍摄时的微小变形。
- zoom_range=0.15
随机缩放图片(范围 0.85-1.15 倍),比如放大一点或缩小一点,让模型适应不同大小的肺部区域。
- horizontal_flip=True
随机水平翻转图片(左右反过来),因为左右肺结构对称,翻转后不影响判断,还能增加数据量。
- brightness_range=[0.8, 1.2]
随机调整亮度(在原图的 80%-120% 之间),模拟不同设备拍摄的明暗差异。
- fill_mode='nearest'
图片经过旋转、平移等操作后,边缘可能出现空白,用旁边的像素颜色填充这些空白(避免出现黑边影响模型学习)。
这些操作的目的是:让同一张 X 光片产生多种 “变体”,相当于给模型提供更多样的训练样本,避免模型只认 “标准姿势” 的图片,从而提高对真实世界中各种情况的适应能力(比如不同医院、不同设备拍的 X 光片可能有差异)。
而 rescale=1. / 255 和 validation_split=validation_split 不属于增强,前者是把像素值从 0-255 缩放到 0-1(模型更容易处理),后者是划分验证集的比例。
合并数据目录
这个 prepare_data_directories 函数的主要作用是整理训练集和验证集的数据目录,让后续的模型训练更方便。
函数整体目标
原始数据通常会分开存放在 train(训练集)和 val(验证集)两个文件夹里,这个函数会把这两个文件夹里的图片合并到一个临时文件夹中,方便后续统一处理(比如重新划分训练集和验证集)。
逐行代码解释
1. 创建临时目录
temp_dir = os.path.join(base_dir, 'temp_combined') combined_train_dir = os.path.join(temp_dir, 'train') os.makedirs(combined_train_dir, exist_ok=True) |
- 首先在原始数据的根目录(base_dir)下创建一个叫 temp_combined 的临时文件夹(比如 chest_xray/temp_combined)。
- 然后在这个临时文件夹里再创建一个 train 文件夹(chest_xray/temp_combined/train),用来存放合并后的数据。
- os.makedirs(..., exist_ok=True) 表示:如果这个文件夹已经存在,就不重复创建,避免报错。
2. 定义原始数据目录
original_train_dir = os.path.join(base_dir, 'train') # 原始训练集目录 original_val_dir = os.path.join(base_dir, 'val') # 原始验证集目录 |
- 找到原始数据中存放训练集和验证集的文件夹(比如 chest_xray/train 和 chest_xray/val)。
3. 合并两个目录的图片(按类别)
for class_name in ['NORMAL', 'PNEUMONIA']: # 遍历两个类别:正常、肺炎 # 创建类别子目录 class_dir = os.path.join(combined_train_dir, class_name) # 比如 temp_combined/train/NORMAL os.makedirs(class_dir, exist_ok=True) # 确保类别文件夹存在 # 复制原始训练集的图片到合并目录 src_train = os.path.join(original_train_dir, class_name) # 原始训练集的类别目录 for file in os.listdir(src_train): # 遍历该类别下的所有图片文件 src_file = os.path.join(src_train, file) # 原始图片路径 dst_file = os.path.join(class_dir, file) # 目标路径(合并目录) if not os.path.exists(dst_file): # 如果目标路径没有这张图,才复制 shutil.copy(src_file, dst_file) # 复制图片 # 复制验证集的图片到合并目录(和上面逻辑一样) src_val = os.path.join(original_val_dir, class_name) # 原始验证集的类别目录 if os.path.exists(src_val): # 防止验证集目录不存在报错 for file in os.listdir(src_val): src_file = os.path.join(src_val, file) dst_file = os.path.join(class_dir, file) if not os.path.exists(dst_file): # 避免重复复制 shutil.copy(src_file, dst_file) |
- 因为数据是按类别分的(NORMAL 正常、PNEUMONIA 肺炎),所以要分别处理每个类别的图片。
- 先把原始训练集里的 NORMAL 图片复制到临时目录的 NORMAL 文件夹,再把原始验证集里的 NORMAL 图片也复制过去,肺炎图片同理。
- if not os.path.exists(dst_file) 是为了避免重复复制同一张图片(比如训练集和验证集可能有重名文件)。
4. 返回结果
return temp_dir, combined_train_dir |
- 函数最后返回两个路径:临时文件夹的路径(temp_dir)和合并后的数据目录(combined_train_dir),方便后续代码使用。
为什么要这么做?
原始数据可能是别人划分好的 train 和 val,但我们可能想按自己的比例重新划分(比如用 85% 做训练、15% 做验证)。这个函数先把所有数据合并,后续就可以用 ImageDataGenerator 的 validation_split 参数灵活划分了,更方便调整。
简单说,就是 “先把所有食材倒进一个大碗,再按自己的口味重新分配”。
数据生成器generator:高效助力电脑学习X光片(前文已描述)
数据生成器(generator)你可以理解成一个 “自动上菜的服务员”,专门给电脑 “喂” 数据的。
为啥需要这个 “服务员”?
如果你的电脑要学 10000 张 X 光片,这些片子加起来可能有几个 G 大。如果一下子全塞进电脑内存(相当于 “一口气把所有菜都端上桌”),内存可能装不下,电脑会变慢甚至卡死。
这时候就需要 “数据生成器” 这个服务员:它不一次性把所有片子都拿出来,而是一批一批地给(比如一次给 32 张),电脑学完这 32 张,再给下 32 张,循环往复,直到学完所有。
数据生成器具体干了啥?
- 按批次取数据:比如你设定 “一批 32 张”,它就每次从文件夹里挑 32 张 X 光片。
- 顺便做预处理:拿片子的时候,自动把它们改成统一大小(比如 150x150),或者旋转、缩放一下(增加数据多样性,让电脑学得更灵活)。
- 给片子贴标签:每张片子对应的 “正常” 或 “肺炎” 标签,它也会一起拿给电脑,不用你手动对应。
迁移学习在医疗中的应用
比如你想学会 “骑自行车”,但你已经会 “骑电动车” 了 —— 电动车的平衡感、转向技巧这些能力,其实可以直接用到骑自行车上,不用完全从零开始学。
我们代码中用了ResNet50。
- ResNet50 这个 “学霸” 已经通过海量图片(比如日常照片、自然场景等)学会了通用的 “看图能力”(比如识别边缘、纹理、颜色变化等基础特征),这些能力就像 “骑电动车的平衡感”。
- 我们的模型要做的是 “看 X 光片判断肺炎”,这是一个更专业的任务,但它可以先把 ResNet50 的 “基础看图能力” 拿过来用(通过加载权重文件),再在此基础上专门学习 “肺炎特征”(比如肺部阴影的形状、位置等)。
这就是迁移学习的本质:把一个任务中学到的知识,迁移到另一个相关任务上,让新任务学得更快、更好。
如果不用迁移学习,我们的模型就得从 “怎么识别像素点” 开始学起,效率会很低,尤其是在医疗数据有限的情况下,效果可能很差。而借助 ResNet50 的 “已有知识”,相当于站在巨人的肩膀上,能快速聚焦到专业任务上。
下载 ResNet50 权重
这个 download_resnet_weights 函数的作用很简单:下载一个叫 “ResNet50 权重” 的文件,给我们的肺炎检测模型当 “预习资料”。
先理解什么是 “权重文件”
你可以把 ResNet50 想象成一个 “已经学过很多图片的学霸”,它通过大量图片训练后,记住了很多通用的看图规律(比如如何识别边缘、颜色、纹理等基础特征)。
“权重文件” 就是这个学霸的 “笔记”,里面记录了它学到的所有规律(以数字形式存储)。我们直接用这份笔记,就不用让模型从零开始学看图了,能省很多时间和精力。
函数具体做了什么
1. 定义文件的 “来源” 和 “存放位置”
weights_url = "..." # 权重文件在网上的地址(就像一个下载链接) local_path = "models/..." # 下载后存在电脑上的位置(models文件夹里) |
2. 准备存放文件夹
os.makedirs("models", exist_ok=True) |
- 在当前文件夹下创建一个叫 models 的文件夹(如果已经有了就不重复创建),专门用来放这个权重文件。
3. 检查是否已经下载过
if not os.path.exists(local_path): # 如果本地没有这个文件,就执行下载操作 else: # 如果已经有了,就直接返回文件路径 |
- 避免重复下载,省流量和时间。
4. 下载文件(如果没下载过)
print("正在下载ResNet50权重文件...") response = requests.get(weights_url, stream=True) # 用链接下载文件 response.raise_for_status() # 检查下载是否成功(比如有没有网络问题) # 把下载的内容存到本地文件里 with open(local_path, 'wb') as f: for chunk in response.iter_content(chunk_size=8192): f.write(chunk) # 分小块下载,避免一次性占太多内存 print("权重文件下载完成") |
5. 处理下载失败的情况
except Exception as e: print(f"下载失败: {e}") # 告诉我们失败原因(比如网络断开) print("请手动下载...") # 提供手动下载的链接,避免程序卡在这里 return None |
6. 返回结果
return local_path # 告诉程序“权重文件存在这里了,可以用了” |
为什么要做这个?
我们的模型是用来 “看 X 光片判断肺炎” 的,但它首先得学会 “怎么看图片”(比如识别图片里的各种细节)。
ResNet50 的权重文件就像一本 “看图入门教材”,我们的模型先 “读” 这本教材,掌握基础的看图能力,再去学更专业的 “肺炎判断”,会学得更快、更好。
简单说,这个函数就是帮我们把 “学霸的笔记” 下载到本地,让我们的模型能站在别人的肩膀上学习,不用从零开始。
改进模型架构:
使用全局平均池化代替展平层
添加更多正则化
与展平层的比较肺炎检测中全局平均池化
咱们用最通俗的例子解释 “全局平均池化” 和 “展平层” 的原理,以及为什么前者更适合这个肺炎检测模型:
核心目的:把 “网格图” 变成 “一串数字”
不管是哪种层,最终都是为了同一个目标:模型中间会生成类似 “网格状的细节图”(比如 7×7 的小格子,每个格子有一个数字代表特征),但后面的计算层只能处理 “一串数字”,所以需要转换格式。
展平层(Flatten):像抄电话号码一样 “硬抄”
假设现在有一张 7×7 的网格图(类似手机键盘的布局,每个按键上有一个数字):
1 2 3 4 5 6 7 8 9 0 1 2 3 4 ...(共7行7列) |
展平层的做法是:从第一行第一个数字开始,按顺序把所有数字抄成一串,比如 “1,2,3,4,5,6,7,8,9,0,...”,最后变成 49 个数字的长串。
特点:不做任何计算,原样保留所有细节,但数字太多(7×7=49 个),容易让模型记不住重点。
全局平均池化层(GlobalAveragePooling2D):像算平均分一样 “提炼重点”
还是那张 7×7 的网格图,全局平均池化的做法是:
把这 7×7=49 个数字加起来,再除以 49,算出一个 “平均值”(比如所有数字加起来是 245,平均值就是 5),最后只保留这 1 个数字。
如果有 2048 张这样的网格图(就像 2048 张不同角度的细节照片),就会算出 2048 个平均值,组成一串 2048 个数字的串。
特点:不算细账,只算总账,数字数量大幅减少(从 49 个→1 个 / 每张图),但保留了每张图的整体特征(比如 “这张图整体数字偏大”)。
为什么肺炎检测要用全局平均池化?
- 医疗影像更看重 “整体特征”:
判断肺炎时,医生更关注 “肺部整体有没有大片阴影”,而不是 “某个像素点亮不亮”。全局平均池化刚好能提炼这种整体特征,就像医生快速扫一眼 X 光片的整体情况。
- 避免模型 “钻牛角尖”:
如果用展平层,模型可能会纠结 “某个小格子的数字变化”(比如胶片上的一点污渍),反而忽略了真正的肺炎特征。全局平均池化能过滤这些无关细节,让模型更专注于关键信息。
- 减少计算量,更稳定:
从 100352 个数字(展平层)减少到 2048 个(全局平均池化),模型学起来更轻松,不容易 “记混”(过拟合),对新的 X 光片适应能力更强。
简单说,展平层是 “把所有细节一股脑塞给模型”,全局平均池化是 “先提炼重点再给模型”。对肺炎检测这种需要抓整体特征的任务,后者显然更合适。
全局平均池化代替展平层在医疗影像中的应用
在这段代码中,build_model函数里的layers.GlobalAveragePooling2D() 就是 “用全局平均池化代替展平层” 的具体体现。
先理解两个 “层” 的作用
不管是 “全局平均池化层” 还是 “展平层”,都是为了把模型中间输出的 “特征图”(可以想象成一张带细节的网格图)转换成 “一维向量”(一串数字),方便后面的全连接层处理。
- 展平层(Flatten):就像 “把网格图上的数字按顺序抄下来”。比如一张 7×7 的网格,会直接变成 7×7=49 个数字的一串,不做任何计算。
- 全局平均池化层(GlobalAveragePooling2D):就像 “对网格图的每一列算平均值”。比如每个特征通道是 7×7 的网格,会算出 7×7 个数字的平均值,变成 1 个数字;如果有 2048 个特征通道,就会得到 2048 个平均值,组成一串数字。
代码中哪里体现了?
看build_model函数里的模型结构:
model = models.Sequential([ base_model, # ResNet50输出的是(7,7,2048)的特征图(7×7的网格,共2048个通道) layers.GlobalAveragePooling2D(), # 全局平均池化层:把(7,7,2048)变成(2048,)的一维向量 layers.Dense(512, activation='relu'), # 后面接全连接层处理 ... # 其他层 ]) |
这里没有用layers.Flatten(),而是用了layers.GlobalAveragePooling2D(),就是明确用 “全局平均池化” 代替了 “展平”。
为什么这么做?
举个例子:
ResNet50 输出的特征图是(7,7,2048)(可以理解为 2048 张 7×7 的细节图)。
- 如果用展平层:会把每张 7×7 的图拆成 49 个数字,2048 张图就会变成 2048×49=100352 个数字,太多了,容易让模型 “记混”(过拟合)。
- 用全局平均池化:每张 7×7 的图只算 1 个平均值,2048 张图就变成 2048 个数字,数量大幅减少,模型更稳定,还能保留每张图的整体特征(比如 “这张图整体偏亮”)。
对医疗影像来说,这种方式更适合 —— 不需要纠结每个像素的细节,而是关注肺部的整体特征(比如阴影的整体分布),同时减少过拟合风险。
什么是正则化
简单说,正则化就是给模型 “加约束”“设规矩”,防止它 “学太疯”。
打个比方:
- 你教孩子认字,正常情况他会学 “猫 = 一种毛茸茸的动物”;
- 但如果孩子 “学太细”,把你教的某张特定照片里 “猫 + 蓝色背景” 当成 “猫” 的定义,那换张白色背景的猫照片,他就认不出来了 —— 这就是 “学太疯”(过拟合)。
- 正则化就像告诉你的孩子:“别只记那张照片的细节,多关注猫本身的样子”,让他学到更通用的规律。
正则化解决什么问题?
核心解决 **“过拟合”** 问题。
- 过拟合:模型在训练数据上表现极好(比如正确率 99%),但换了新数据(测试数据)就变拉胯(比如正确率 60%)。原因是模型 “死记硬背” 了训练数据的细节(包括噪音、偶然特征),而没学到真正的规律。
- 正则化的作用:通过限制模型的 “复杂度”(比如不让它的参数太大、不让它学太多冗余特征),强迫它放弃对训练数据细节的 “死记硬背”,转而学习更普遍的规律,从而在新数据上表现更好。
常见的正则化方法(结合 ResNet50 举例)
- L1/L2 正则化(权重惩罚)
- 白话:给模型的参数(比如卷积层的权重)“上枷锁”,不让它们的值太大。
- 原理:训练时,损失函数里额外加一项 “参数大小的惩罚”,参数越大,惩罚越重。
- 效果:让模型更倾向于用小参数、简单的方式拟合数据,避免 “钻牛角尖”。
- Dropout(随机 “关掉” 神经元)
- 白话:训练时随机让一部分神经元 “休息”(输出设为 0),迫使模型不能依赖某几个神经元的 “特殊能力”。
- 比如在 ResNet50 的全连接层或某些卷积层后加 Dropout,每次训练随机关掉 20% 的神经元,模型就必须学出更稳健的特征(因为不知道谁会被关掉)。
- 数据增强(变相的正则化)
- 白话:给训练数据 “做手脚”(比如随机裁剪、翻转、加噪音),让模型看到更多 “变形” 的数据,避免记住原始数据的固定细节。
- 比如训练 ResNet50 时,把图片随机旋转、缩放后再输入,模型就不会认为 “某个角度的猫才是猫”。
- Batch Normalization(批归一化)
- 白话:让每一层的输入数据 “保持稳定”(比如均值、方差差不多),防止模型被极端数据带偏,间接降低过拟合风险。
- ResNet 本身已经用了 BatchNorm,进一步优化可以调整其参数(如动量)增强稳定性。
- 早停(Early Stopping)
- 白话:模型训练到一定程度就 “喊停”,别等它开始 “死记硬背” 再停。
- 比如观察测试集效果,当测试精度不再提升甚至下降时,就停止训练,避免过拟合。
总结
正则化的本质是 **“牺牲一点训练精度,换取更好的泛化能力”**,让模型从 “只懂训练数据” 变成 “能适应新数据”。在改进 ResNet50 时,合理组合这些方法(比如加 Dropout+L2 正则),能有效提升模型在实际场景中的稳定性。
模型正则化与过拟合防范
在这段代码的模型架构中,有以下几种正则化手段(防止模型过拟合、让模型更稳健的技术):
1. Dropout(随机失活)
代码位置:build_model函数的模型结构中
layers.Dropout(0.5), # 第一个Dropout层,随机丢弃50%的神经元 ... layers.Dropout(0.3) # 第二个Dropout层,随机丢弃30%的神经元 |
- 说明:就像学生做题时,老师随机遮住 50%(或 30%)的知识点,强迫学生不能只依赖某几个知识点解题,必须掌握更通用的规律。
- 作用:训练时随机让一部分神经元 “休息”,避免模型过度依赖某些特征(比如 X 光片上的某个无关噪点),提高模型对新数据的适应能力。
2. BatchNormalization(批量归一化)
代码位置:build_model函数的模型结构中
layers.BatchNormalization(), # 第一个批量归一化层 ... layers.BatchNormalization() # 第二个批量归一化层 |
- 说明:就像把不同来源的试卷(数据)统一难度后再给学生做,避免某张试卷太难或太简单影响学习效果。
- 作用:让每一层的输入数据保持相对稳定的分布(比如均值、方差相近),防止模型因为输入数据波动太大而学偏,同时加速训练过程,让模型更稳定。
layers.BatchNormalization()
可以理解成 “数据标准化工具”,作用是让每层处理的数据 “保持稳定”。
比如:
不同 X 光片的亮度可能差很多(有的亮、有的暗),经过它处理后,会把亮度统一到一个合理范围。
就像老师批改作业时,先把不同学生的字迹大小、卷面整洁度 “标准化”,再公平打分,避免因为数据本身的差异影响学习效果。
3. 冻结预训练模型(间接正则化)
代码位置:build_model函数中
base_model.trainable = False # 冻结ResNet50的权重,不允许更新 |
- 说明:就像借用学霸的笔记时,先完全照搬核心内容,不随便修改,避免自己改乱了基础知识点。
- 作用:ResNet50 预训练模型已经学到了通用的图像特征(如边缘、纹理),冻结后不让这些基础特征被破坏,只训练新增的几层,减少模型的可训练参数,降低过拟合风险。
这些正则化手段的共同目的
防止模型 “死记硬背” 训练数据的细节(比如某个特定 X 光片的噪点),而是学会更通用的规律(比如肺炎的典型阴影特征),从而在新的 X 光片上也能准确判断。
简单说,就是通过各种方式 “约束” 模型,不让它学太细、太偏,保证学习效果更稳健。
训练优化:
添加学习率调度器
增加模型检查点保存
调整早停策略
# 编译模型optimizer = Adam(learning_rate=0.0001) # 使用较小的学习率# 使用Adam优化器model.compile( optimizer=optimizer, loss='binary_crossentropy', metrics=['accuracy',tf.keras.metrics.Precision(name='precision'), # 精确率tf.keras.metrics.Recall(name='recall'), # 召回率tf.keras.metrics.AUC(name='auc') # AUC])
# 回调函数early_stopping = EarlyStopping(monitor='val_auc', # 监控验证集AUCpatience=8, # 提前停止的耐心轮数verbose=1, # 输出日志mode='max', # 最大化AUCrestore_best_weights=True # 恢复最佳权重)reduce_lr = ReduceLROnPlateau( # 学习率调整monitor='val_loss',factor=0.2,patience=3,min_lr=1e-7,verbose=1)model_checkpoint = ModelCheckpoint( # 保存最佳模型'best_pneumonia_model.h5',monitor='val_auc',save_best_only=True,mode='max',verbose=1)# 训练模型history = model.fit(X_train, y_train_resampled,epochs=50,batch_size=32,validation_data=val_generator,class_weight=class_weights,callbacks=[early_stopping, reduce_lr, model_checkpoint], # 回调函数verbose=1)这里只介绍一下 学习率调整;
学习率调度器在肺炎诊断模型中的应用
简单说,学习率调度器就是让模型在训练时 “动态调整学习步伐”。
打个比方:
- 学习率就像你走路的 “步长”—— 刚开始对路况不熟,迈大步子(较大学习率)快速探索;
- 走了一段时间快到目的地了,再迈大步容易走过头,这时候就换成小碎步(较小学习率)慢慢调整,精准到达。
学习率调度器的作用就是:在训练初期用较大学习率加速收敛,后期自动减小学习率,让模型更精准地找到最优解,避免在最优值附近 “来回震荡”。
这段代码中哪里体现了学习率调度器?
在代码中,通过 ReduceLROnPlateau 实现了学习率调度器,具体如下:
reduce_lr = ReduceLROnPlateau( monitor='val_loss', # 监控验证集的损失值 factor=0.2, # 当触发时,学习率乘以0.2(即变为原来的1/5) patience=3, # 如果连续3个epoch,监控的指标(val_loss)没改善,就触发调整 min_lr=1e-7, # 学习率的下限,不能比这个更小 verbose=1 # 调整时打印日志 ) |
训练时盯着 “验证集损失”(val_loss):
- 如果连续 3 个回合(epoch),验证集损失都没下降(没改善),就把当前学习率 “打 2 折”(乘以 0.2);
- 比如原来学习率是 0.0001,调整后就变成 0.00002;
- 但学习率最低不会低于 0.0000001(1e-7),防止学习率过小导致训练停滞。
最后,这个调度器通过回调函数传入训练过程,在训练中自动生效:
model.fit( ..., callbacks=[..., reduce_lr, ...] # 加入学习率调度器 ) |
为什么要用这个调度器?
在肺炎诊断模型中,数据可能存在噪声(如影像模糊),训练后期如果学习率不变,可能会出现 “模型在最优解附近晃悠却达不到最佳效果” 的情况。通过动态减小学习率,能让模型更稳定地收敛到更好的性能(比如更高的准确率和召回率)。
解决类别不平衡:
结合过采样和类别权重
类别权重在模型训练中的应用
1. 计算类别权重的代码
通过公式直接计算 “正常(NORMAL)” 和 “肺炎(PNEUMONIA)” 两个类别的权重,核心代码如下:
# 计算类别权重 n_normal = np.sum(y_train_original == 0) # 正常样本的数量 n_pneumonia = np.sum(y_train_original == 1) # 肺炎样本的数量 total = n_normal + n_pneumonia # 总样本数 # 计算权重(核心公式) weight_for_normal = (1 / n_normal) * (total / 2.0) # 正常类的权重 weight_for_pneumonia = (1 / n_pneumonia) * (total / 2.0) # 肺炎类的权重 # 用字典存储权重,键为类别标签(0代表正常,1代表肺炎) class_weights = {0: weight_for_normal, 1: weight_for_pneumonia} # 打印权重,方便查看 print(f"类别权重: 正常={weight_for_normal:.2f}, 肺炎={weight_for_pneumonia:.2f}") |
- 样本少的类别会被赋予更高的权重。例如,若肺炎样本只有正常样本的 1/3,那么肺炎的权重会是正常样本的 3 倍左右。
- 公式的本质是:样本数量越少,权重越大,以此平衡模型对少数类的关注度。
2. 训练时应用类别权重
在模型训练的fit函数中,通过class_weight参数传入上述计算好的权重,让模型在训练时生效:
history = model.fit( X_train, y_train_resampled, epochs=50, batch_size=32, validation_data=val_generator, class_weight=class_weights, # 这里传入类别权重 callbacks=[early_stopping, reduce_lr, model_checkpoint], verbose=1 ) |
- 训练时,模型计算 “损失值”(判断模型预测错误的程度)时,会给权重高的类别(如肺炎)的错误 “加钱”—— 比如肺炎样本被错判时,损失值会比正常样本被错判时更大。
- 这样模型会更 “害怕” 错判少数类(肺炎),从而被迫更认真地学习肺炎样本的特征,避免因样本少而被忽略。
总结
代码通过 “计算权重→传入训练” 两个步骤实现了类别权重的应用,核心目的是让模型在肺炎样本可能较少的情况下,依然能重视肺炎的识别,减少漏诊(提高召回率)。
权重调整与模型训练
上面代码中,乘以 (total/2.0) 是为了给权重 “定一个合理的总量”,避免权重数值太大或太小,让模型训练更稳定。
举个个生活例子你就明白了:
假设你有两个苹果(正常样本)和 1 个橘子(肺炎样本):
- 1/n_normal = 1/2 = 0.5(苹果的基础权重)
- 1/n_pneumonia = 1/1 = 1(橘子的基础权重)
这时候橘子权重已经是苹果的 2 倍,确实体现了 “少即是大”。但如果不加 (total/2.0):
- 苹果权重 = 0.5,橘子权重 = 1,总权重 = 1.5
加了 (total/2.0) 之后(total=3,total/2=1.5):
- 苹果权重 = 0.5×1.5=0.75
- 橘子权重 = 1×1.5=1.5
- 总权重 = 2.25
核心作用有两个:
- 让权重总量更合理:乘以总样本的一半后,权重总和会接近总样本数的一半,避免权重数值过小(比如 0.5 和 1)导致对模型影响太弱,或过大导致训练不稳定。
- 保持权重比例不变:乘以同一个数(total/0),橘子和苹果的权重比例还是 2:1(和原来一样),既保留了 “少数类权重更高” 的特性,又让数值大小更适合模型训练。
就像给天平加砝码:1 克和 2 克的比例是 1:2,乘以 3 之后变成 3 克和 6 克,比例没变,但重量更适合天平测量了。这里的 (total/2.0) 就相当于那个 “3”,让权重数值处于模型更容易 “感知” 的范围。
