Materials Studio学习笔记(二十九)——尿素的几何优化
本文参考中科大的MaterialsStudio教程的Forcite内容。
现在开始对于尿素进行几何优化,在对于晶体进行结构优化的时候,Forcite保持对称性,本文主要验证的是对称性对于实验结果是否重要。
首先导入尿素的XSD文件,点击打开Materials Studio软件所在的位置,然后选择share,选择Structures,再选择repeat-unit,再选择urea导入xsd文件:
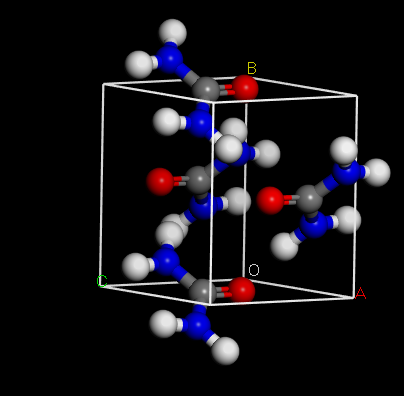
然后开始检查晶体的对称性,点击Build选项栏中Symmetry,然后点击showSymmetry,结果如下所示:
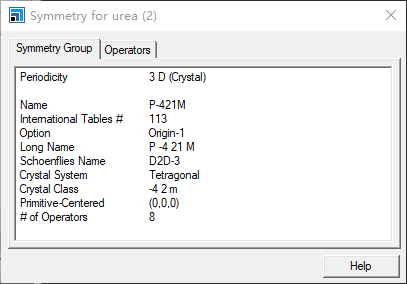
可以看到空间群为P-421M。
然后开始进行几何优化,点击Forcite工具,进入下面的界面,将Task设置为Ceometry Optimization,然后将Quality设置为Fine:
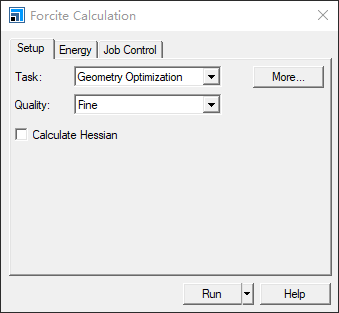
然后点击More,进入Forcite Gemotery Optimization,然后点击Optimize cell关闭:
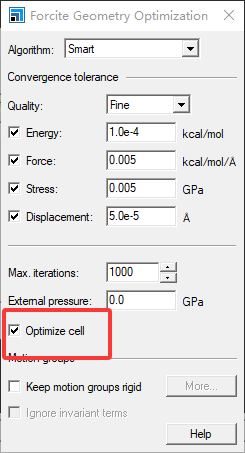
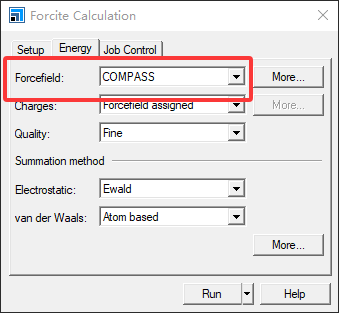
然后点击Energy,然后Forcefield选择为COMPASS,其他保持不变,然后点击run:
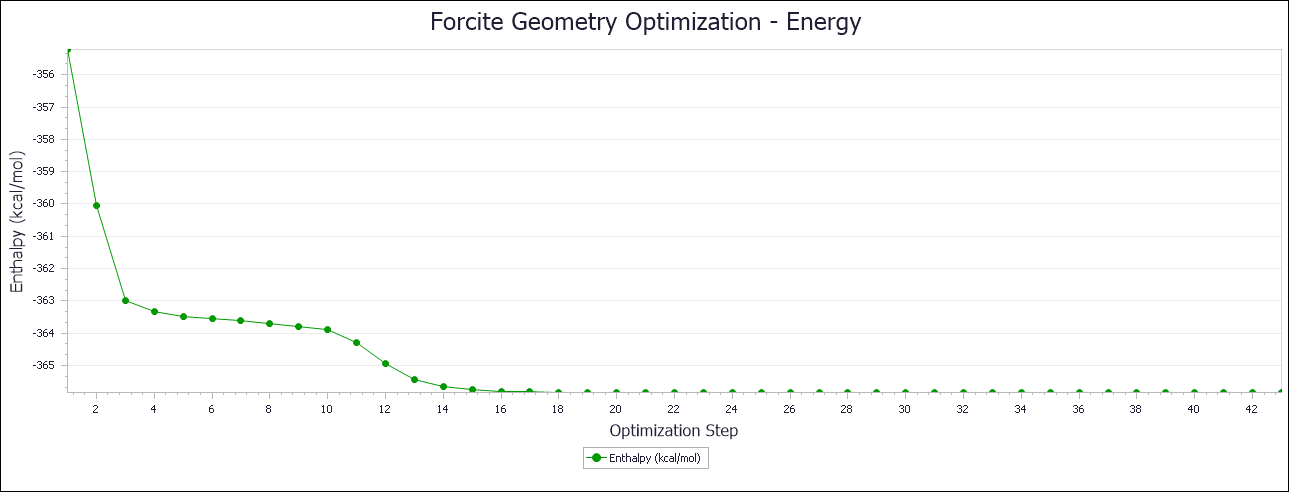
运行完毕之后,再文件夹中有下面几个文件。其中Status.txt文件包含job的最后执行状态。Energy图标显示了在优化过程中总能量的变化,Convergence图表显示收敛标准的变化,即Energe Charge和Gradient Norm对于优化步骤的函数,当所需要的标准到达时模拟就停止了。
首先打开能量文件,结果如下所示:
现在应该确保对称性没有发生改变,点击CeomeOpt,然后查看xsd文件的对称性,打开xsd文件,然后点击Build选项栏中Symmetry中Show Symmetry,结果如下所示:
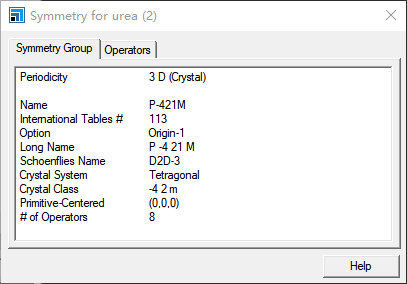
可以看到仍然是P-421M,可以看到对称性没有发生改变。
现在将原来刚刚导入的尿素文件进行复制,然后新建一个xsd文件,复制到新的xsd文件中:
现在开始移除对成型,点击Build选项栏中的Symmetry中的Make P1。然后点击Build选项栏中Symmetry中的show Symmetry,显示如下所示:
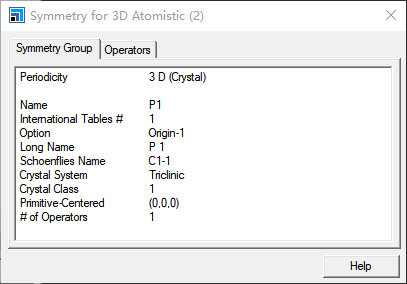
可以看到对称性为P1。
然后保持和之前完全相同的参数,点击Forcite模块的Calculation,然后和前面保持不变,点击run。计算完成之后,点击保存,下面开始比较两次计算的结算结果。
打开两者的状态输出文件,比较总能量,结果如下所示:
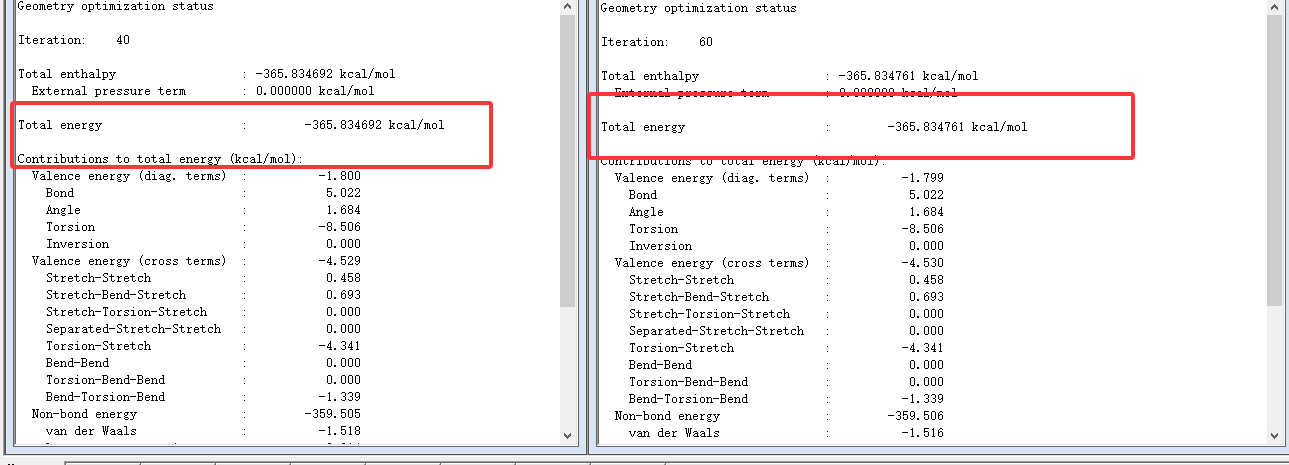
可以看到总能量约为 -365.8347 kcal/mol左右,能量基本接近。
打开两侧的xcd文件,然后进行比较如下面两图所示:
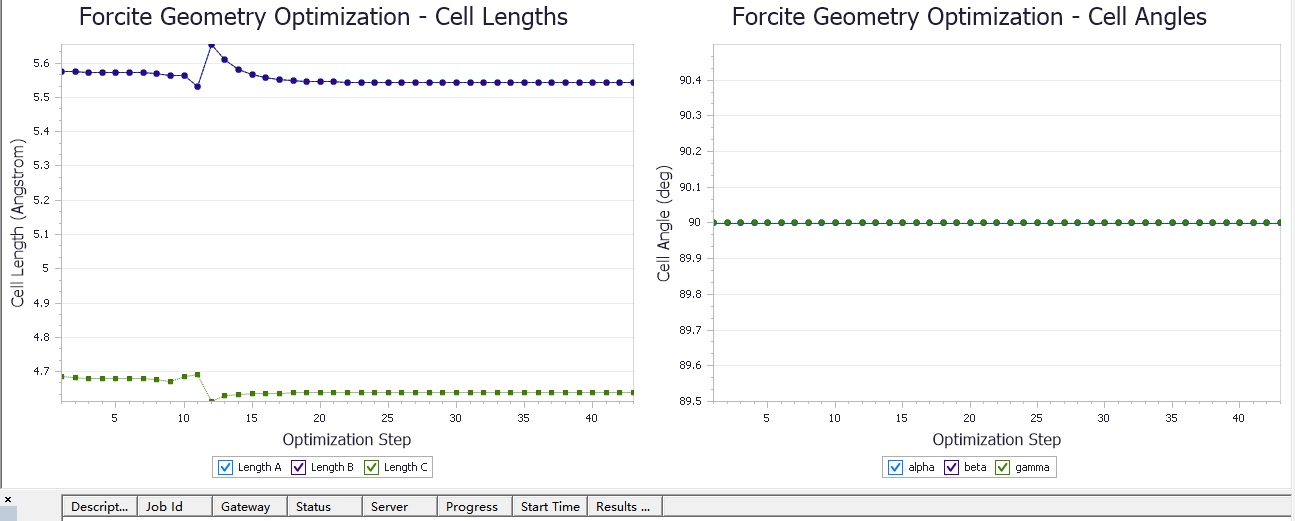
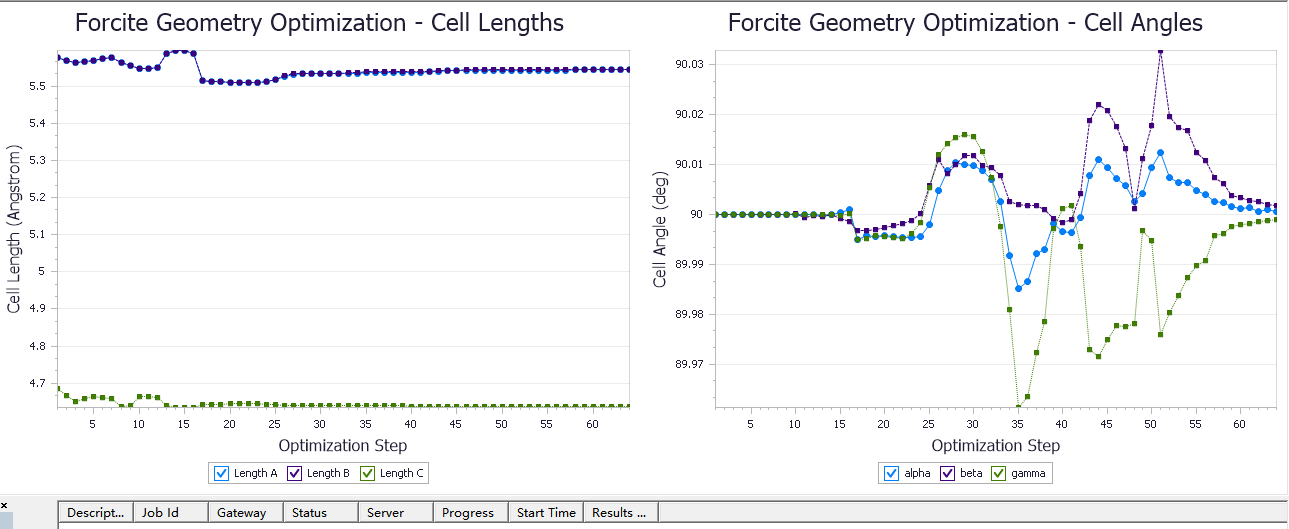
对于上面两张图中,第一张是具有完全对称性的尿素晶体,可以看到晶胞长度有轻微的减少,然后晶胞夹角仍然为90°。在没有对称结构的P1结构时,晶胞夹角发生的变化比较敏感,最终收缩在90°。
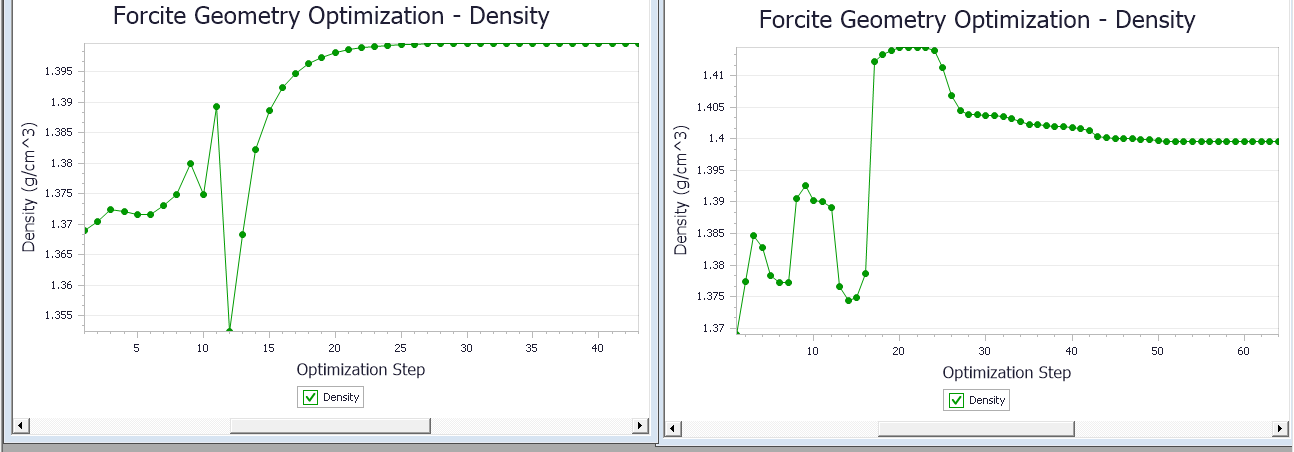
对于具有对称性的尿素晶胞,密度有轻微的增加,最终值略低于,而对于没有对称性最终也是略低于
。
可以看到两种情况下的几何情况是完全相同的,但是最后计算的结果是非常相近的。
